Содержание
- 2. При аналізі первинних структур процедура вирівнювання виявляє сходство між послідовностями (sequence similarity), яке може свідчити про
- 3. Гомологичные последовательности – последовательности, имеющие общее происхождение (общего предка). Признаки гомологичности белков сходная 3D-структура в той
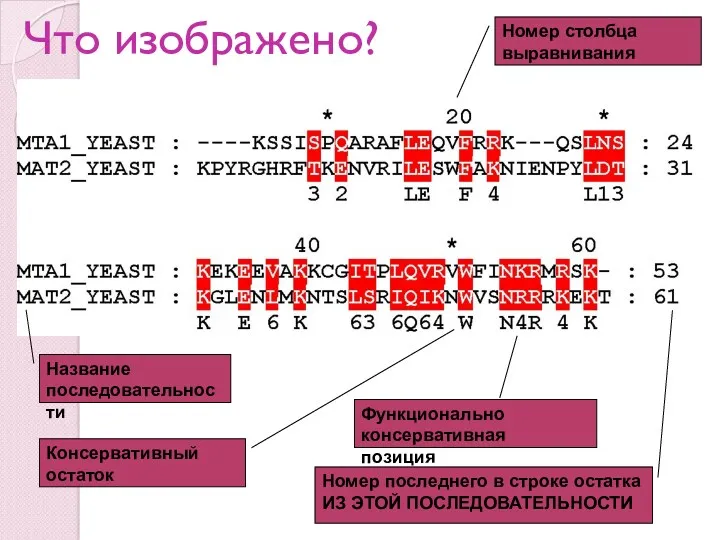
- 4. Что изображено? Название последовательности Номер столбца выравнивания Номер последнего в строке остатка ИЗ ЭТОЙ ПОСЛЕДОВАТЕЛЬНОСТИ Консервативный
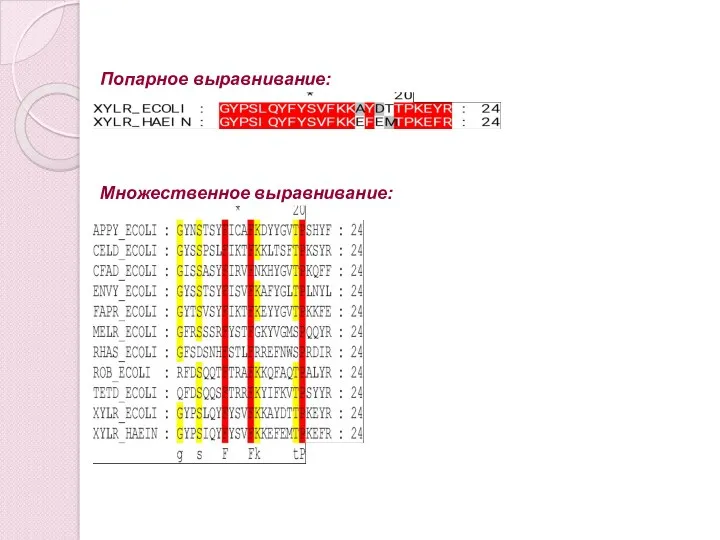
- 5. «Идеальное» выравнивание – запись последовательностей одна под другой так, чтобы гомологичные фрагменты оказались друг под другом.
- 7. Ортологи и паралоги Ортологи – гени з різних організмів, що розійшлися при видоутворенні. Мається на увазі,
- 8. Множественное выравнивание: содержание Определение, разновидности, решаемые задачи, общие проблемы Глобальное выравнивание Прогрессивное выравнивание Итерационные методы Локальные
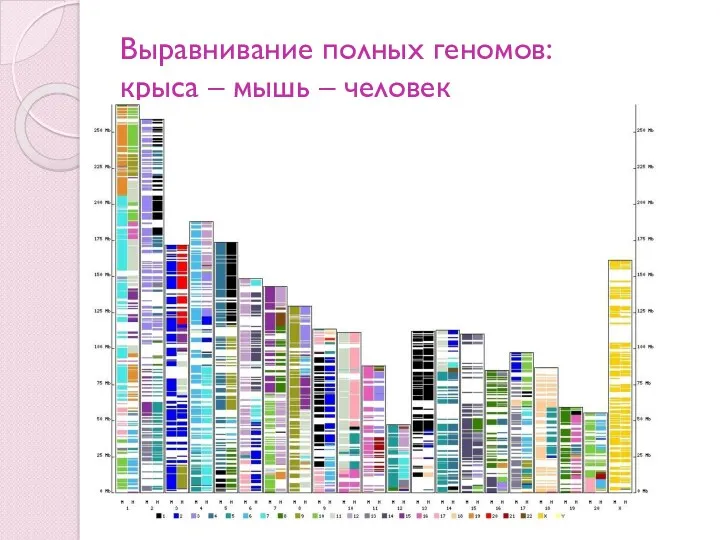
- 9. Выравнивание полных геномов: крыса – мышь – человек
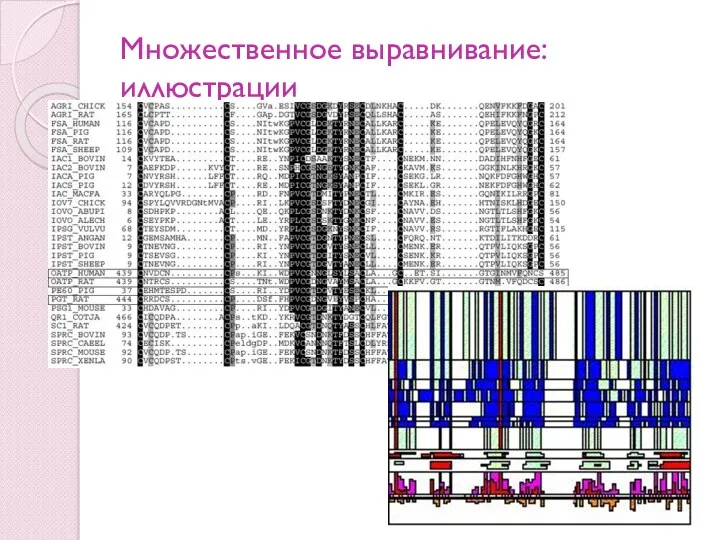
- 10. Множественное выравнивание: иллюстрации
- 11. Множественное выравнивание: определение и проблемы Определение: найти оптимальное соответствие между несколькими последовательностями, если заданы Матрица соответствия
- 12. Множественное выравнивание: проблемы (прод.) Проблемы: Локальные минимумы накопление первоначальных ошибок в иерархических алгоритмах лучшее дерево соответствует
- 13. Множественное выравнивание: решаемые задачи Поиск мотивов (блоков) – коротких сигнатур, идентифицируемых в консервативных участках множественного выравнивания
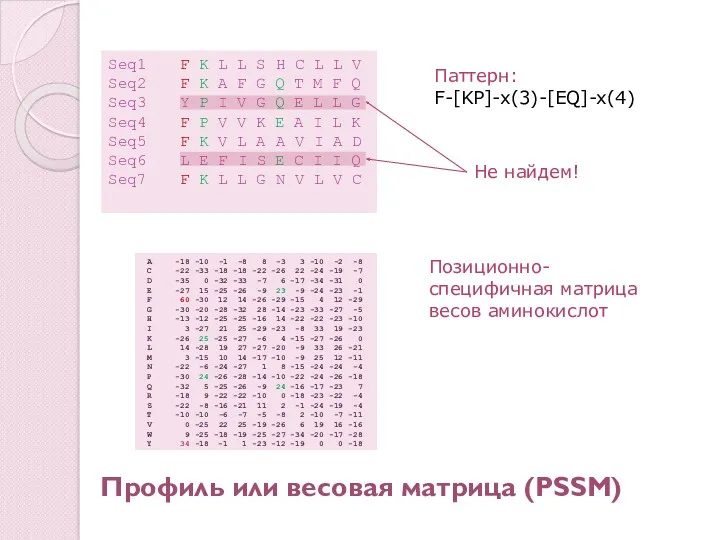
- 14. Паттерн – регулярное выражение UNIX’a: [AC]-x-V-x(4)-{ED} Ala или Cys- х-Val- х- х- х - х- (любой,
- 15. Профиль или весовая матрица (PSSM) Seq1 F K L L S H C L L V
- 16. Множественное выравнивание: области применения Один из ключевых методов в современной молекулярной биологии Сферы применения Филогенетический анализ,
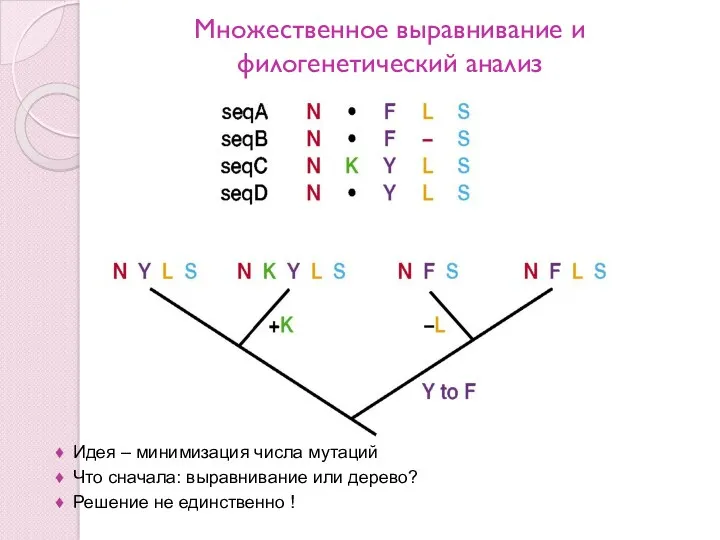
- 17. Множественное выравнивание и филогенетический анализ Идея – минимизация числа мутаций Что сначала: выравнивание или дерево? Решение
- 18. Множественное выравнивание: консервативные участки во многих пос-стях Консервативный – не значит «совпадающий» !
- 19. Множественное выравнивание: белки vs. ДНК Выравнивание белковых семейств В алфавите много «букв» Эволюционная близость белковых молекул,
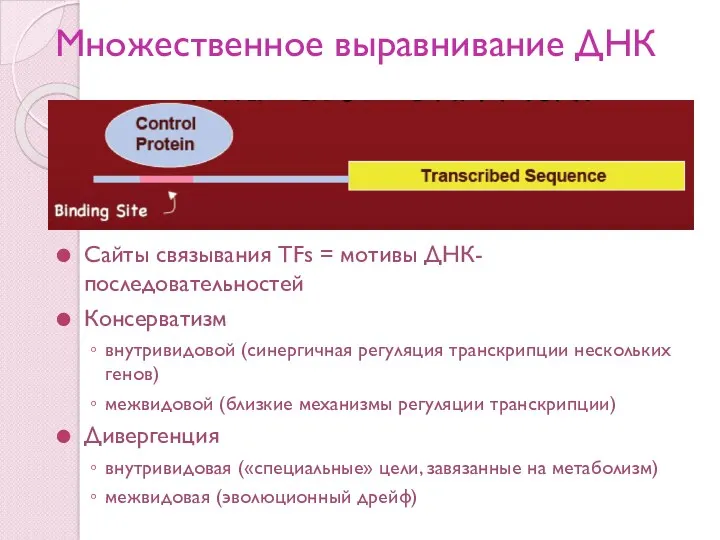
- 20. Множественное выравнивание ДНК Сайты связывания TFs = мотивы ДНК-последовательностей Консерватизм внутривидовой (синергичная регуляция транскрипции нескольких генов)
- 21. Множественное выравнивание ДНК: проблемы и варианты решения Гораздо сложнее выравнивания белков всего 4 «буквы» Отсутствие «золотого
- 22. Множественное выравнивание: четыре группы методов Прогрессивное глобальное выравнивание начать с наиболее близких пос-стей Итерационные процедуры выравнивание
- 23. Множественное выравнивание: история До 1987 г. множественные выравнивания строились вручную Sankoff (1975 и 1987) – первый
- 24. Глобальное выравнивание (обобщение ДП)
- 25. Глобальное выравнивание Обобщение метода динамического программирования программа MSA (Lipman et al., 1989) результат далек от оптимального
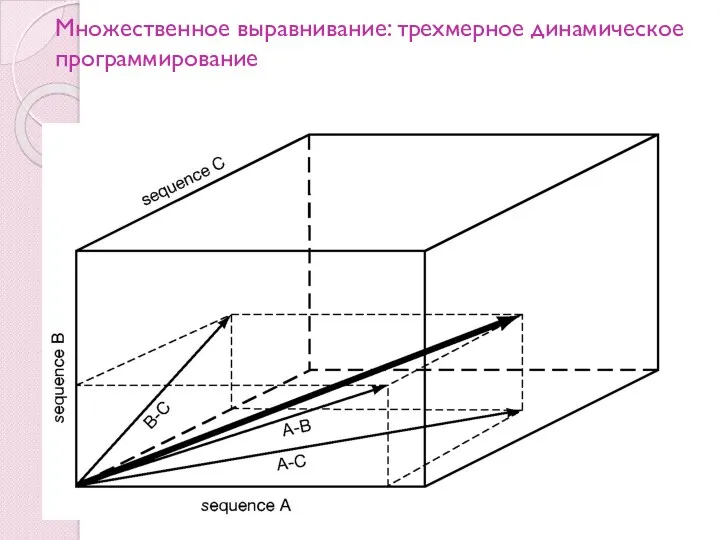
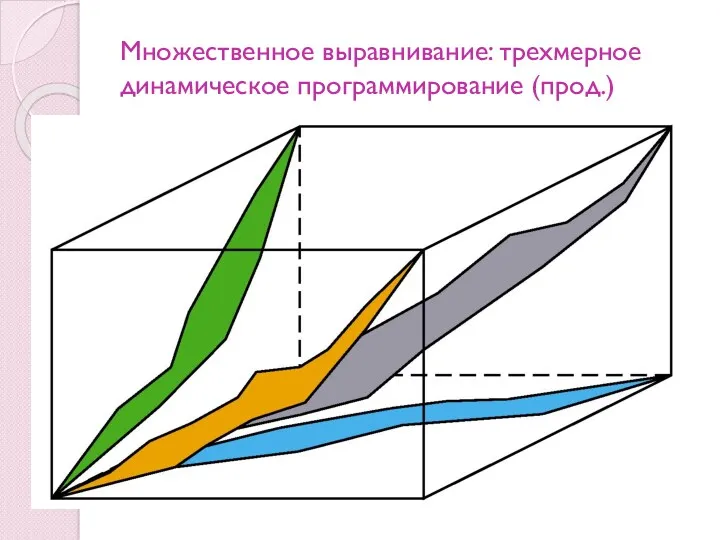
- 26. Множественное выравнивание: трехмерное динамическое программирование
- 27. Множественное выравнивание: трехмерное динамическое программирование (прод.)
- 28. Глобальное выравнивание (прод.) Оценка качества веса множественных выравниваний (SP score) = сумме весов попарных выравниваний поиск
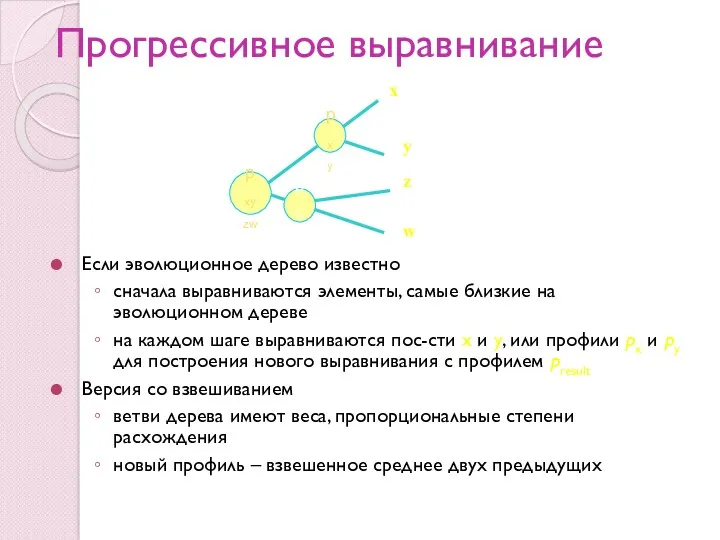
- 29. Прогрессивное выравнивание
- 30. Прогрессивное выравнивание: идея Сначала – эволюционно наиболее близкие пос-сти Постепенное добавление новых пос-стей / групп пос-стей
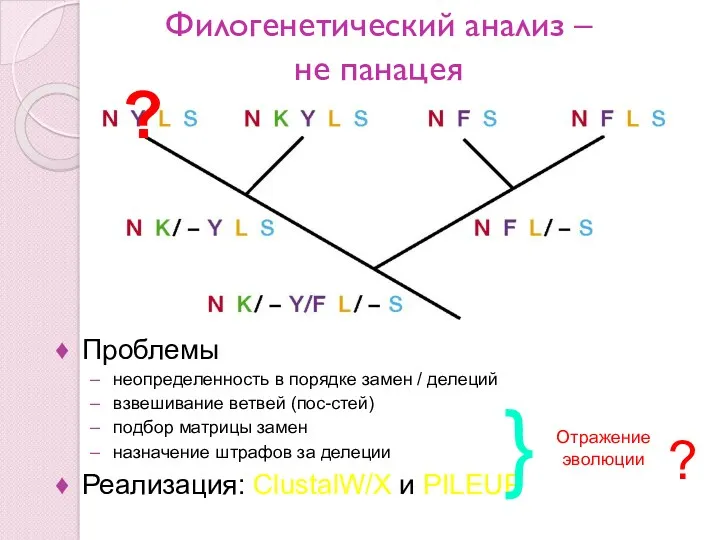
- 31. Филогенетический анализ – не панацея Проблемы неопределенность в порядке замен / делеций взвешивание ветвей (пос-стей) подбор
- 32. Прогрессивное выравнивание Если эволюционное дерево известно сначала выравниваются элементы, самые близкие на эволюционном дереве на каждом
- 33. Прогрессивное выравнивание (прод.) Если эволюционное дерево неизвестно: построить всевозможные парные выравнивания определить матрицу расстояний D, элементы
- 34. Прогрессивное выравнивание: детали алгоритма Три этапа попарные выравнивания «каждая с каждой» филогенетическое дерево по весам парных
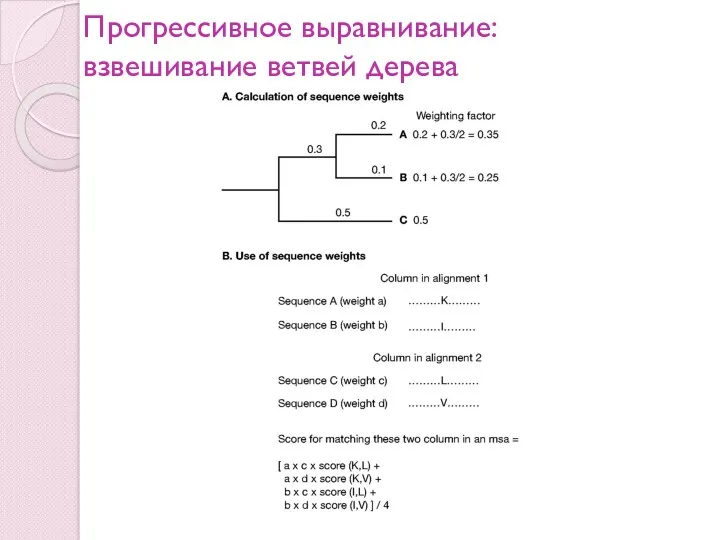
- 35. Прогрессивное выравнивание: детали алгоритма (прод.) Взвешивание пос-стей (ветвей дерева) мультипликативная модель Штрафы за делеции предыдущие делеции
- 36. Прогрессивное выравнивание: взвешивание ветвей дерева
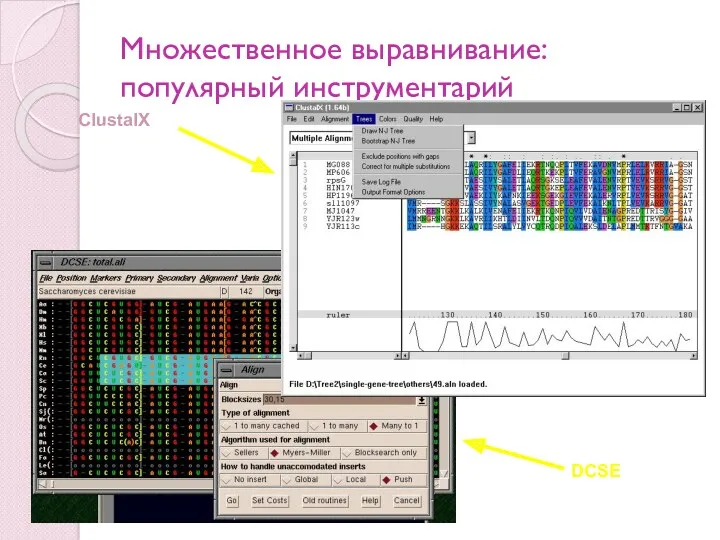
- 37. Множественное выравнивание: популярный инструментарий ClustalX DCSE
- 38. Прогрессивное выравнивание: штрафы за делеции Существующие делеции влияют на выравнивание следующих пос-стей их позиции фиксируются ClustalW:
- 39. Прогрессивное выравнивание: штрафы за делеции (прод.) Компенсационная модификация штрафов средний вес соответствий по матрице замен уровень
- 40. Прогрессивное выравнивание: проблемы Результат зависит от начальных парных выравниваний ошибки первых выравниваний накапливаются выравнивание непохожих пос-стей
- 41. Итерационное выравнивание
- 42. Итерационное выравнивание: идея метода Задача избежать накопления ошибок начальных выравнива-ний, свойственных прогрессивным методам Вариант решения многократные
- 43. Итерационное выравнивание: варианты реализации MultAlin (Corpet, 1998) пересчет весов парных выравниваний в прогрессивном алгоритме использование весов
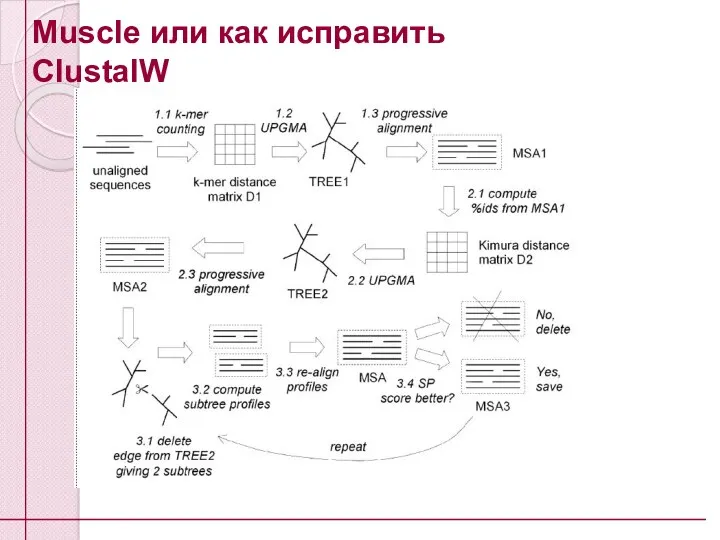
- 44. Muscle или как исправить ClustalW
- 45. Локальные множественные выравнивания
- 46. Локальные множественные выравнивания: виды алгоритмов Анализ профилей Блочное выравнивание Поиск мотивов Статистические методы
- 47. Анализ профилей: введение Идея: MSA для группы пос-стей Выделение высоко консервативных участков в мини-MSA Профиль -
- 48. Анализ профилей: идентификация в семействе белков теплового шока (hsp70) Матрица весов (профиль) содержит вероятности встречаемости АК
- 49. Блочное выравнивание: семейство из 34 тубулиновых белков
- 50. Анализ профилей: ограничения Профиль отражает вариабельность в данном MSA смещение в сторону похожих пос-стей вариант коррекции:
- 51. Профиль – позиционно-специфическая матрица замен 21 столбец и N строк N – длина последовательностей в выравнивании
- 52. Множественное выравнивание на базе вероятностно-статистических методов Максимизация математического ожидания Сэмплирование Гиббса Скрытые марковские модели see Russ
- 53. Множественное выравнивание, весна 2008 Наиболее известные программы множественного выравнивания: MSA => оптимальное выравнивание, если дождаться результата
- 54. Структурное выравнивание
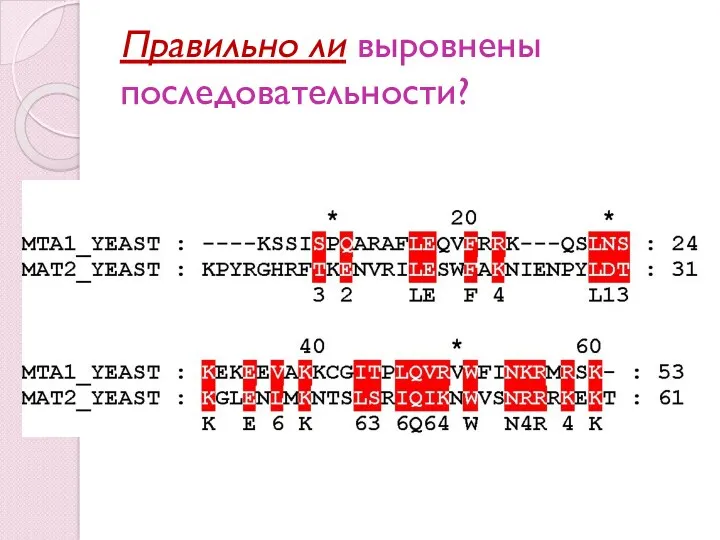
- 55. Правильно ли выровнены последовательности?
- 56. В чем биологический смысл выравнивания? Буквы в одной колонке определяют сопоставление аминокислотных остатков двух белков Сопоставленные
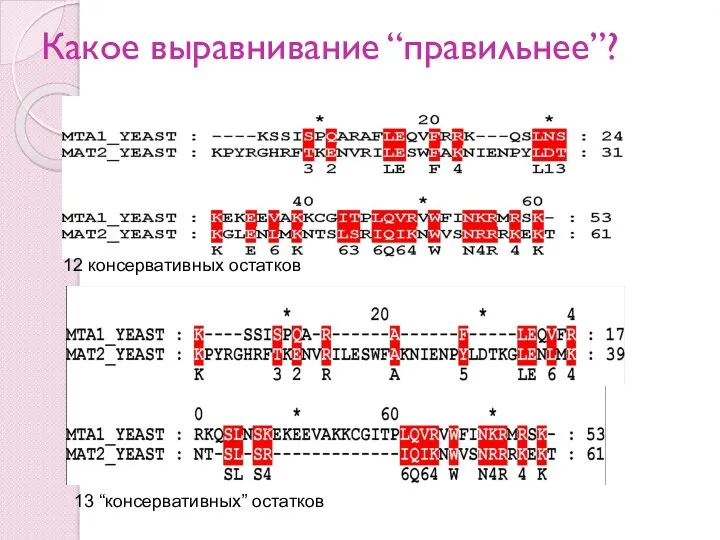
- 57. Какое выравнивание “правильнее”? 13 “консервативных” остатков 12 консервативных остатков
- 58. Чтобы понять смысл выравнивания, вернемся к тому, что такое последовательность аминокислотных остатков и что такое белок
- 59. (i)Последовательность – удобный способ закодировать структурную (химическую) формулу молекулы белка (до посттрансляционных модификаций) (iii) Последовательность однозначно
- 60. Пространственное совмещение полипептидных цепей белков mta1_yeast и mat2_yeast На плоской картинке видно плохо ☹
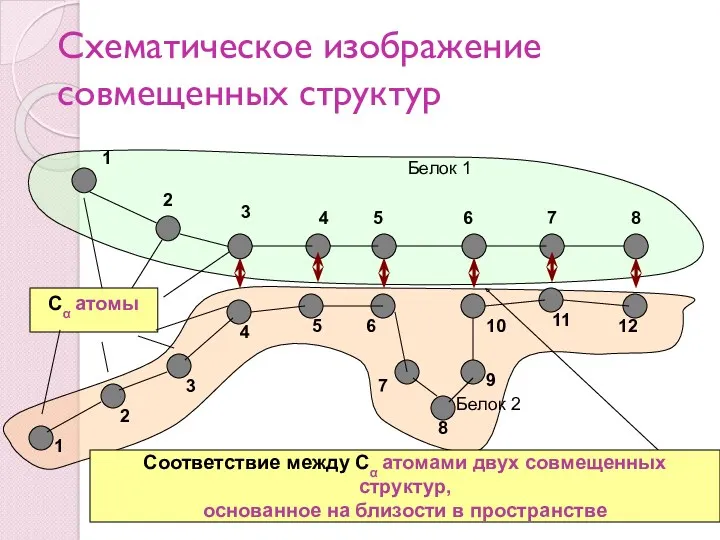
- 61. Схематическое изображение совмещенных структур
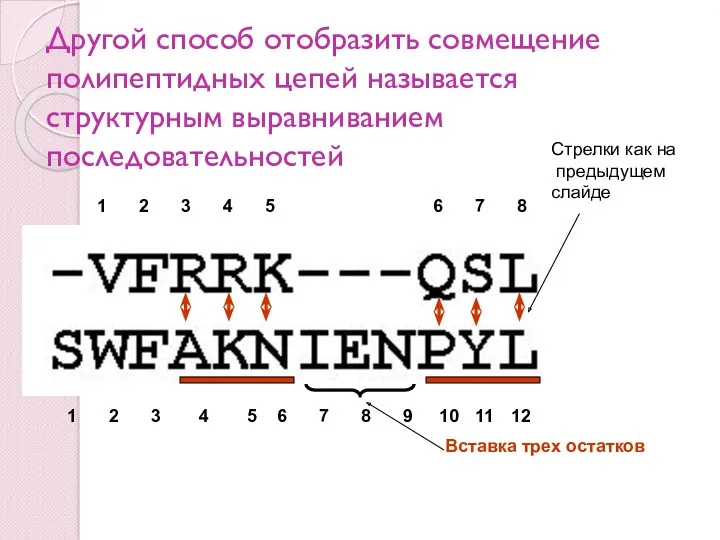
- 62. Другой способ отобразить совмещение полипептидных цепей называется структурным выравниванием последовательностей Стрелки как на предыдущем слайде
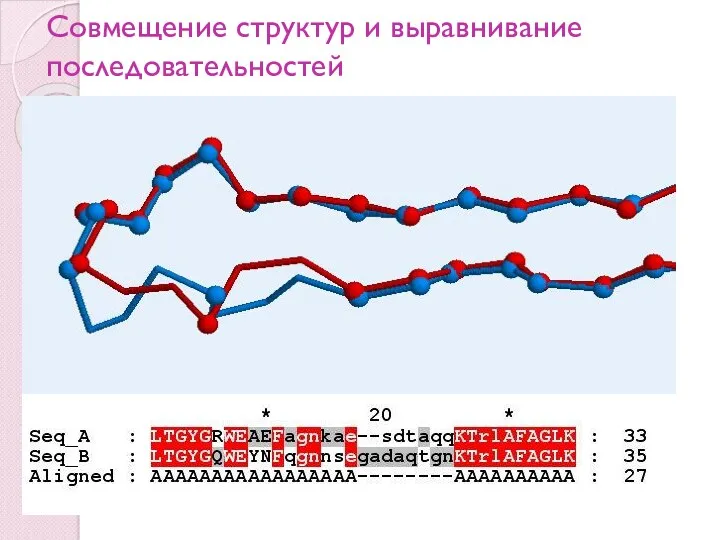
- 63. Совмещение структур и выравнивание последовательностей
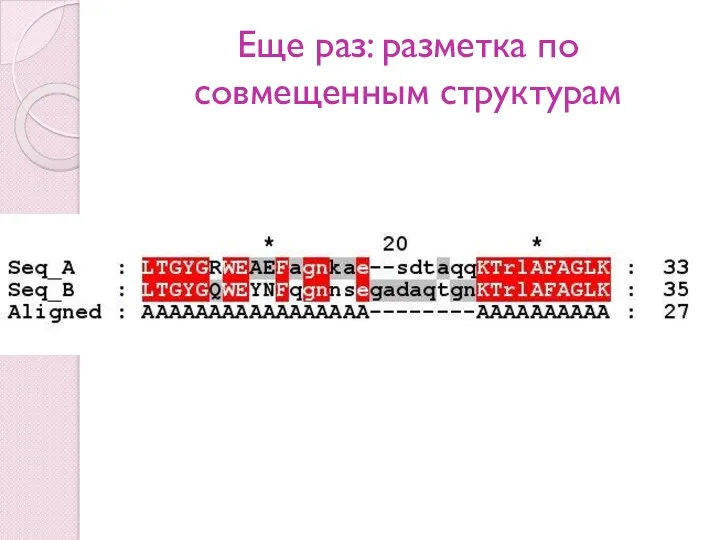
- 64. Еще раз: разметка по совмещенным структурам
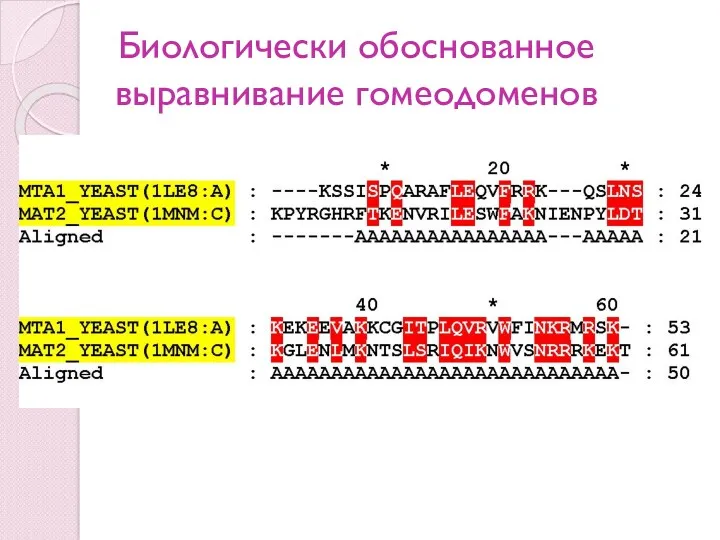
- 65. Биологически обоснованное выравнивание гомеодоменов
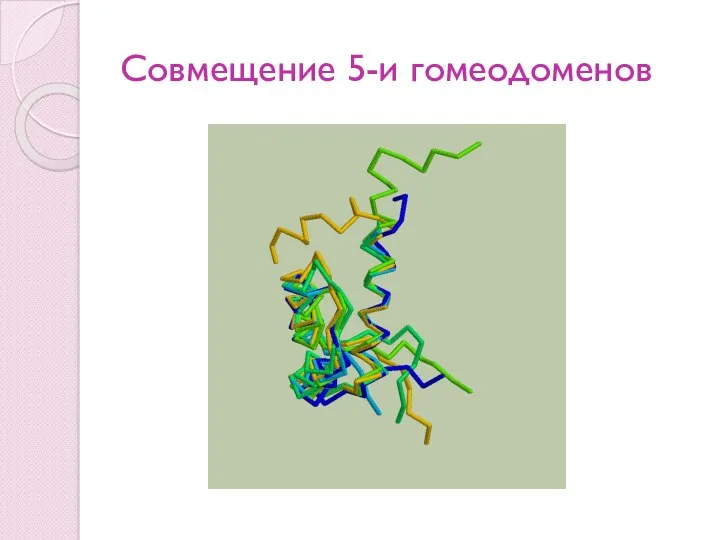
- 66. Совмещение 5-и гомеодоменов
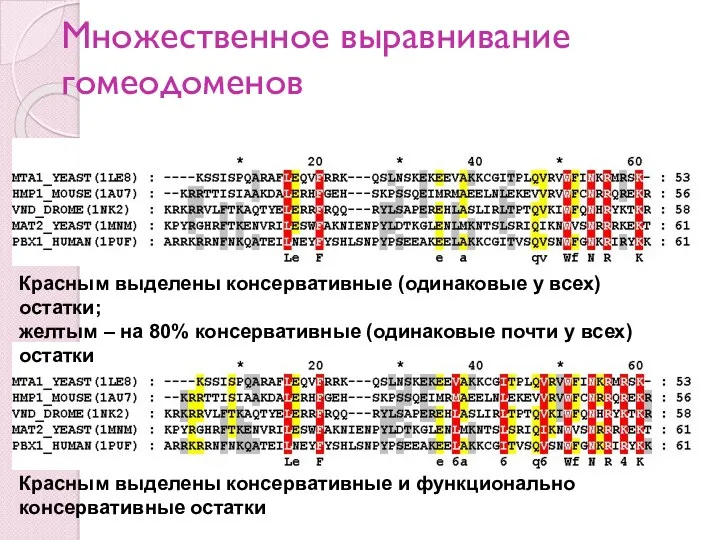
- 67. Множественное выравнивание гомеодоменов Красным выделены консервативные (одинаковые у всех) остатки; желтым – на 80% консервативные (одинаковые
- 68. Размеченное множественное выравнивание
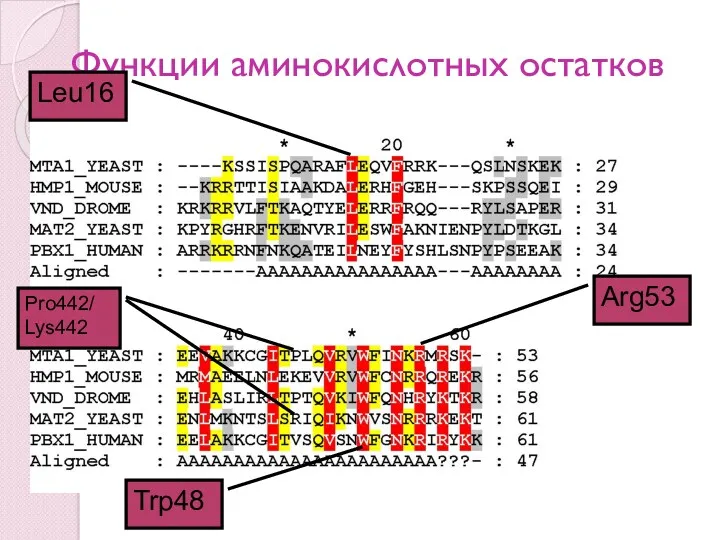
- 69. Функции аминокислотных остатков Trp48 Arg53 Leu16 Pro442/ Lys442
- 70. В “правильном” выравнивании много консервативных аминокислотных остатков и функционально консервативных позиций
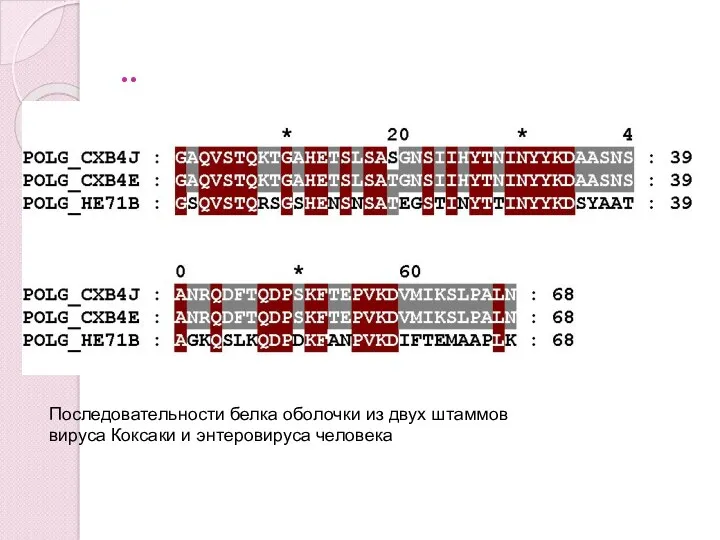
- 71. Выравнивание и эволюция Последовательности белка оболочки из двух штаммов вируса Коксаки
- 72. .. Последовательности белка оболочки из двух штаммов вируса Коксаки и энтеровируса человека
- 73. Аминокислотные остатки в одной колонке биологически обоснованного выравнивания, как правило, “произошли” из одного и того же
- 74. Алгоритмические решения проблемы воплощены в программах Программы выравнивания последовательностей тестируются путем сравнения с биологически обоснованными –
- 75. Предположим, известны структуры родственных белков и, значит, биологически обоснованное выравнивание последовательностей При > 60% совпадающих букв
- 76. Применения «золотой стандарт» для выравнивания высоко гомологичных белков – выявление общего предка идентификация общих значимых элементов
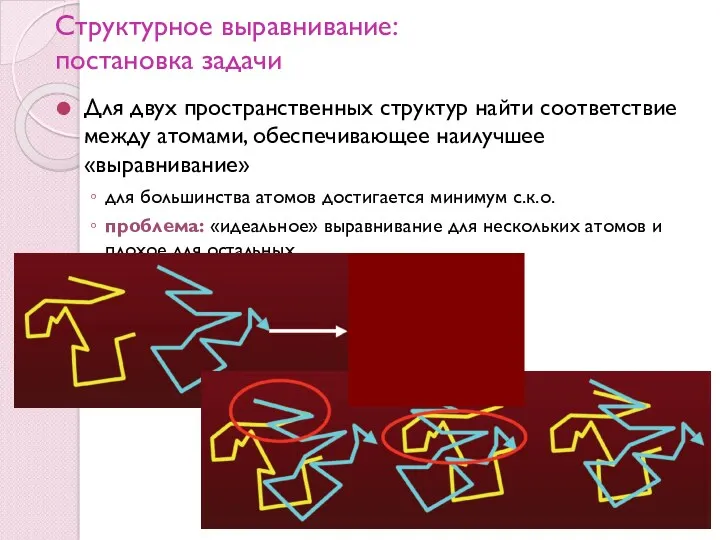
- 77. Структурное выравнивание: постановка задачи Для двух пространственных структур найти соответствие между атомами, обеспечивающее наилучшее «выравнивание» для
- 78. Структурное выравнивание: оценка результата Критерии число соответствий между АК суммарное евклидово расстояние между выровненными АК доля
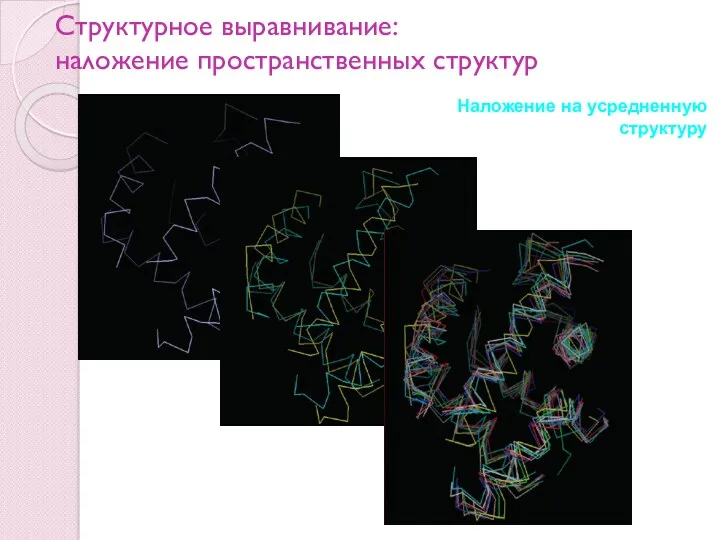
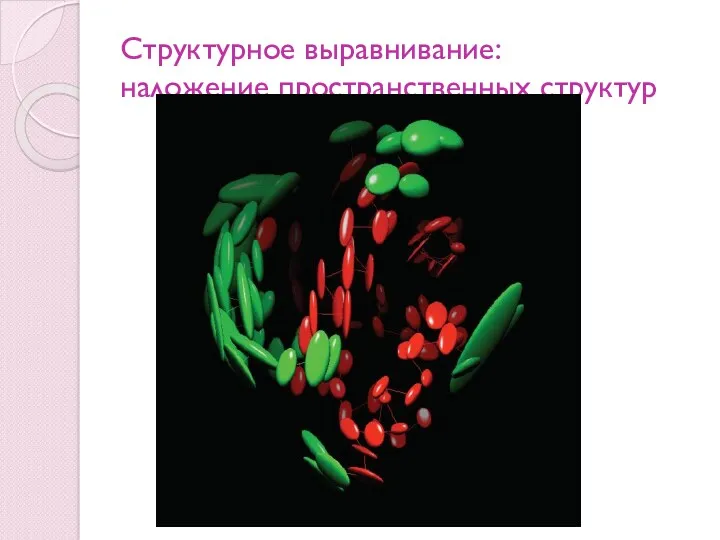
- 79. Структурное выравнивание: наложение пространственных структур Наложение на усредненную структуру
- 80. Структурное выравнивание: наложение пространственных структур
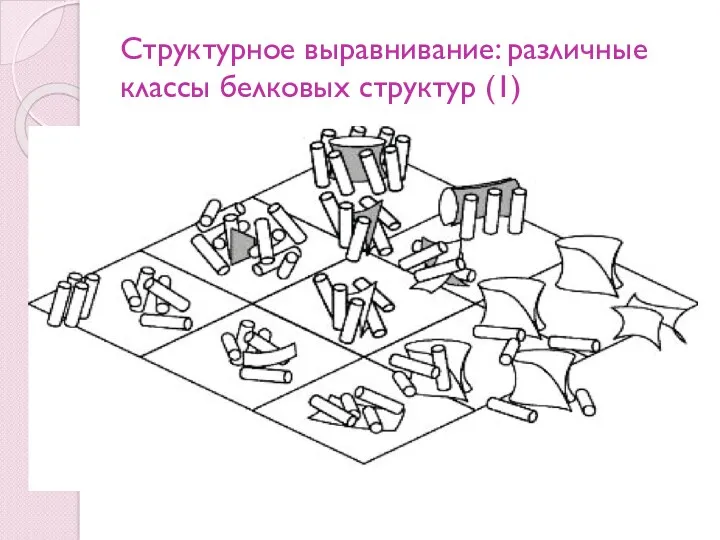
- 81. Структурное выравнивание: различные классы белковых структур (1)
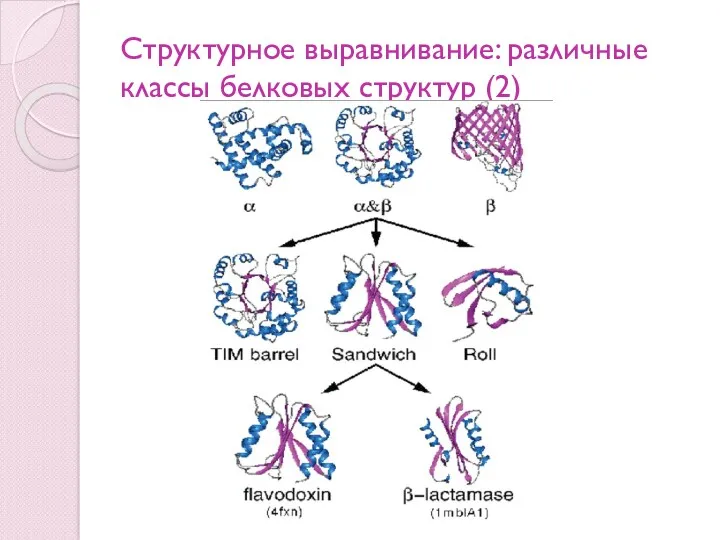
- 82. Структурное выравнивание: различные классы белковых структур (2)
- 83. Структурное выравнивание: различные классы белковых структур (3) Разные суперсемейства «бочонков»
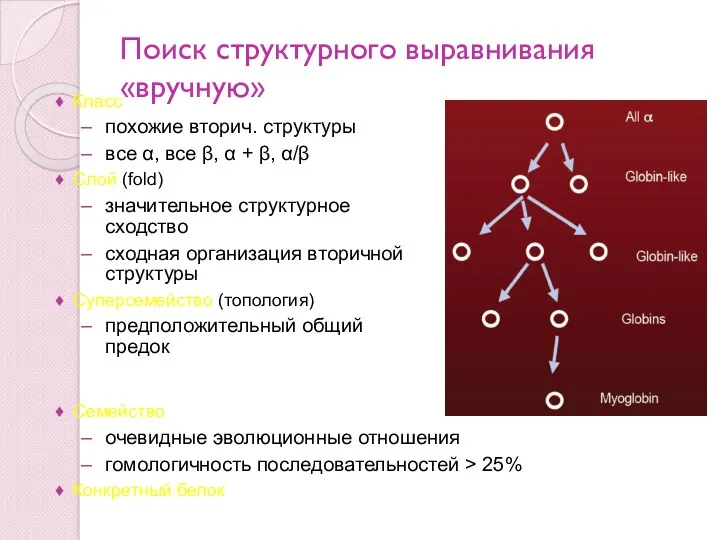
- 84. Поиск структурного выравнивания «вручную» Класс похожие вторич. структуры все α, все β, α + β, α/β
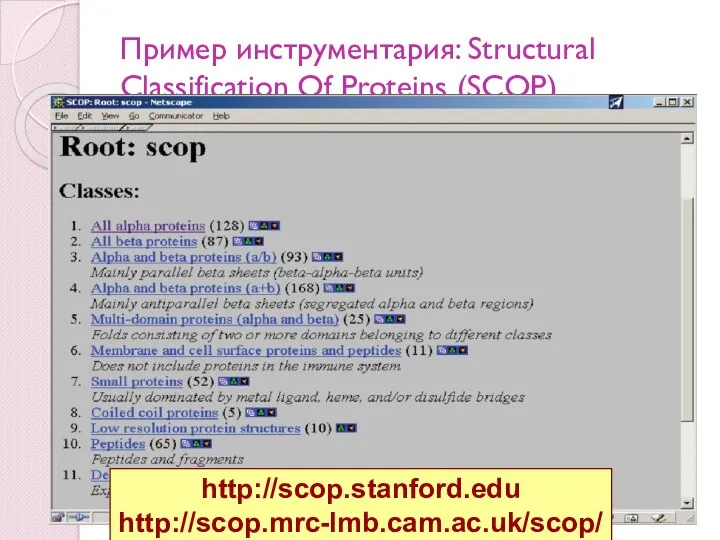
- 85. Пример инструментария: Structural Classification Of Proteins (SCOP) http://scop.stanford.edu http://scop.mrc-lmb.cam.ac.uk/scop/
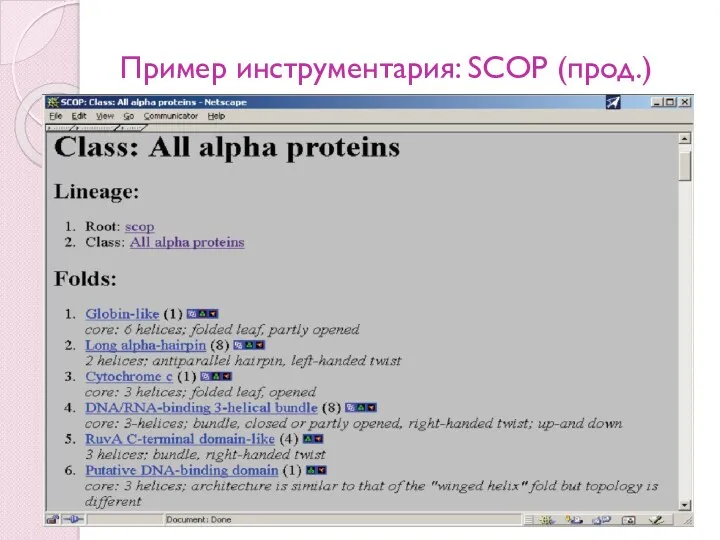
- 86. Пример инструментария: SCOP (прод.)
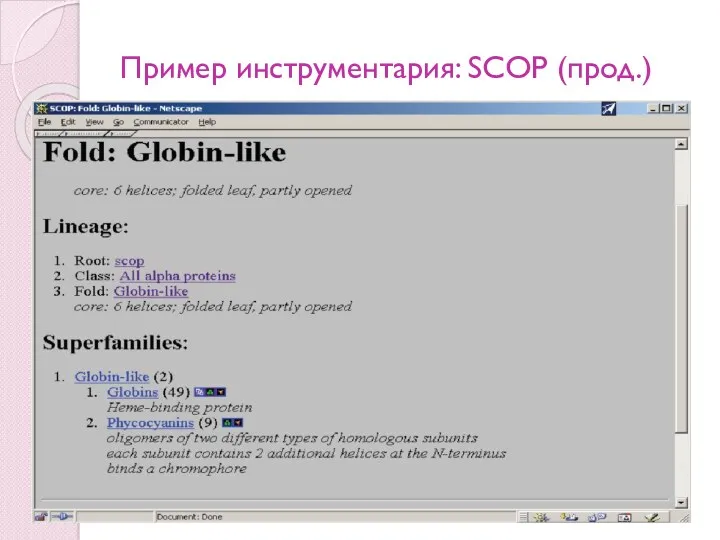
- 87. Пример инструментария: SCOP (прод.)
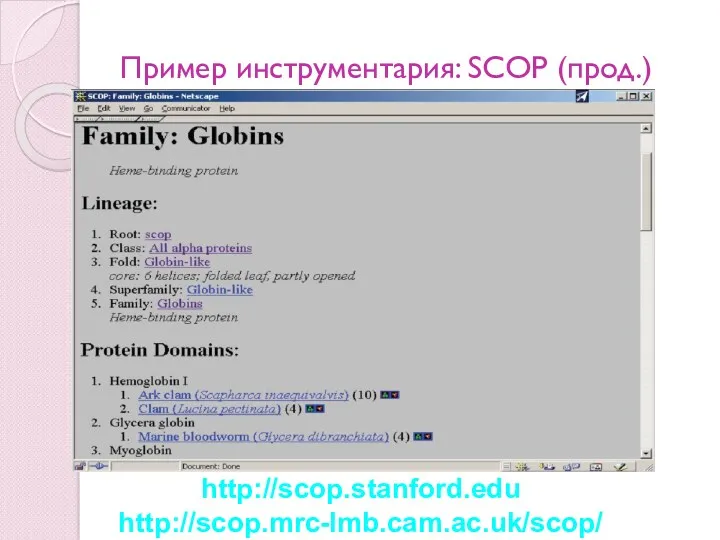
- 88. Пример инструментария: SCOP (прод.) http://scop.stanford.edu http://scop.mrc-lmb.cam.ac.uk/scop/
- 89. Как распознать близость структур? На глаз Алгоритмически точечные методы: установление соответствий по точечным свойствам (расстояниям) анализ
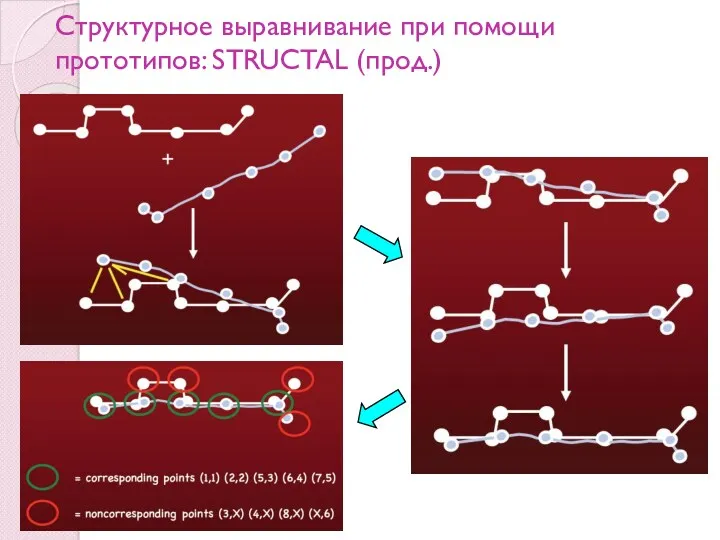
- 90. Структурное выравнивание при помощи прототипов: STRUCTAL Итерационное динамическое программирование для улучшения случайно выбранного начального выравнивания Шаги
- 91. Структурное выравнивание при помощи прототипов: STRUCTAL (прод.) Оценка выравнивания: чем лучше выравнивание, тем выше суммарный вес
- 92. Структурное выравнивание при помощи прототипов: STRUCTAL (прод.) Итерационное динамическое программирование
- 93. Структурное выравнивание при помощи прототипов: STRUCTAL (прод.)
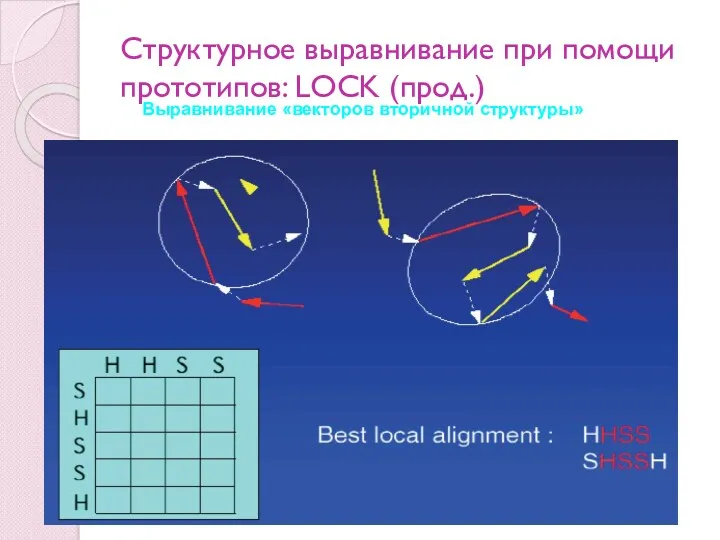
- 94. Структурное выравнивание при помощи прототипов: LOCK Основная идея: элементы вторичной структуры представляются при помощи векторов быстрый
- 95. Структурное выравнивание при помощи прототипов: LOCK (прод.) Сравнение «векторов вторичной структуры»
- 96. Структурное выравнивание при помощи прототипов: LOCK (прод.) Выравнивание «векторов вторичной структуры»
- 97. Структурное выравнивание при помощи прототипов: LOCK (прод.) Шаги алгоритма определить локальные элементы вторичной структуры построить начальное
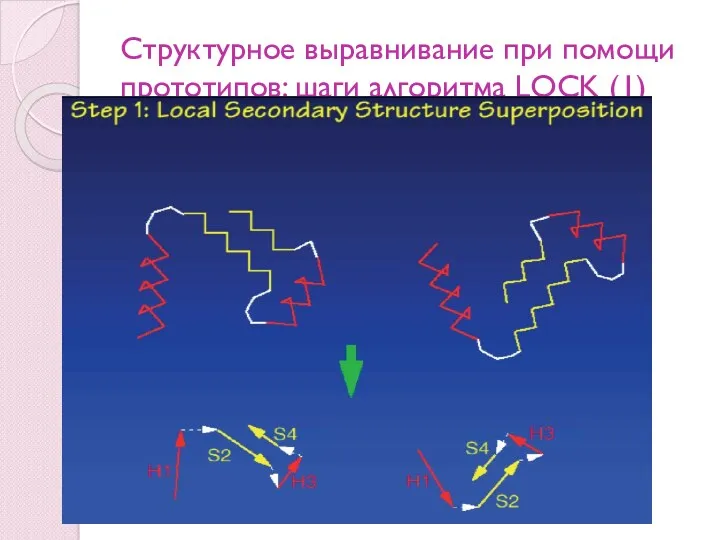
- 98. Структурное выравнивание при помощи прототипов: шаги алгоритма LOCK (1)
- 99. Структурное выравнивание при помощи прототипов: шаги алгоритма LOCK (1a)
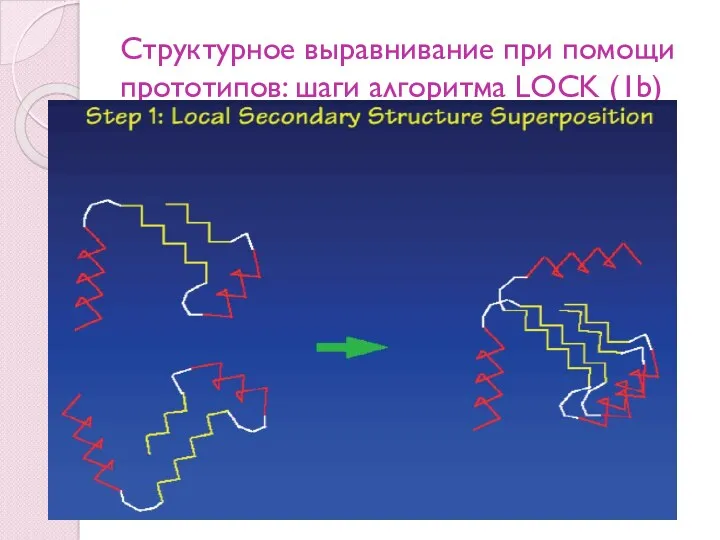
- 100. Структурное выравнивание при помощи прототипов: шаги алгоритма LOCK (1b)
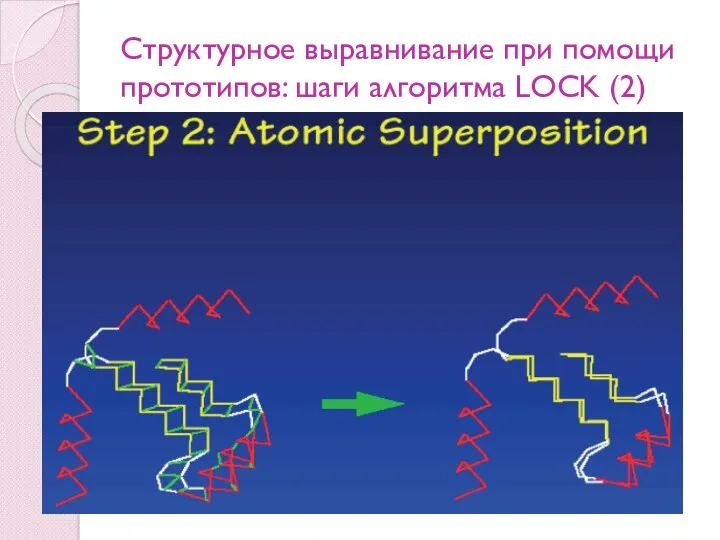
- 101. Структурное выравнивание при помощи прототипов: шаги алгоритма LOCK (2)
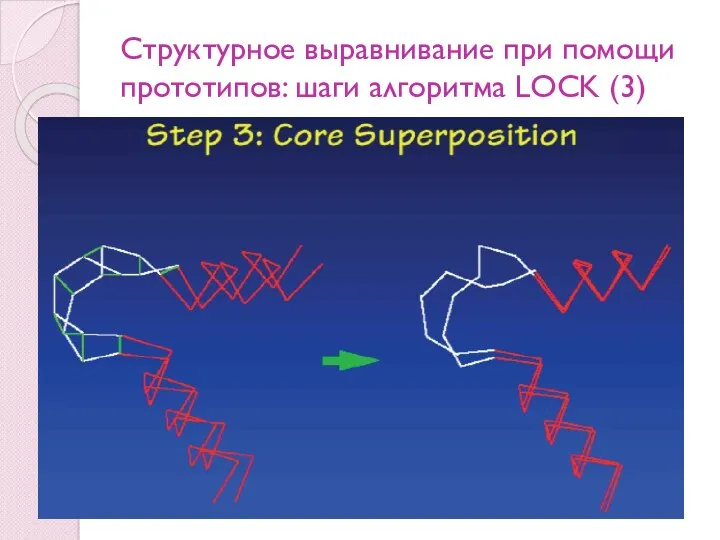
- 102. Структурное выравнивание при помощи прототипов: шаги алгоритма LOCK (3)
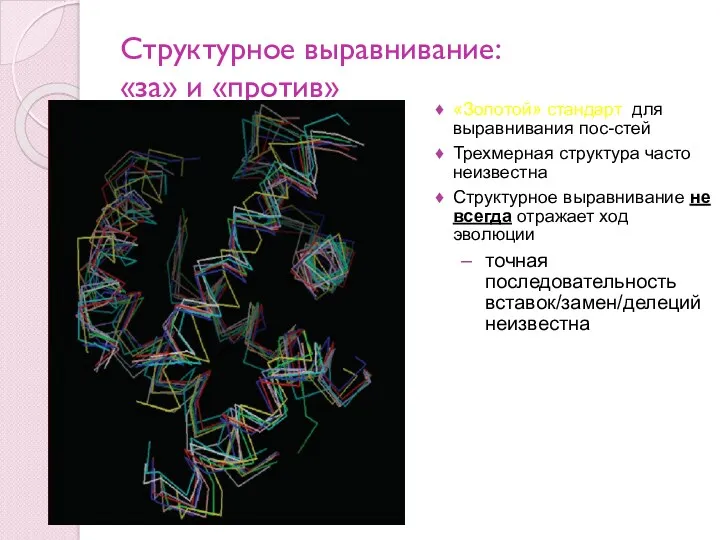
- 103. Структурное выравнивание: «за» и «против» «Золотой» стандарт для выравнивания пос-стей Трехмерная структура часто неизвестна Структурное выравнивание
- 104. ПРОБЛЕМА: как построить “правильное” выравнивание последовательностей белков если структуры белков неизвестны?
- 105. На сегодня известны: более 10 млн(!!!) последовательностей белков (включая фрагменты и трансляты) пространственные структуры около 70
- 107. Скачать презентацию








![Паттерн – регулярное выражение UNIX’a: [AC]-x-V-x(4)-{ED} Ala или Cys- х-Val-](/_ipx/f_webp&q_80&fit_contain&s_1440x1080/imagesDir/jpg/425913/slide-13.jpg)




















































 Нервная система человека. Клетки нервной ткани
Нервная система человека. Клетки нервной ткани Фитонцидная активность комнатных растений
Фитонцидная активность комнатных растений Семейства рыб
Семейства рыб Мінез-құлық қалыптасуының жалпы принцептері. ЖЖІӘ туралы түсінік. Туа біткен және жүре пайда болған іс әрекеттерінің түрлері
Мінез-құлық қалыптасуының жалпы принцептері. ЖЖІӘ туралы түсінік. Туа біткен және жүре пайда болған іс әрекеттерінің түрлері Стебель. Внутреннее и внешнее строение стебля. Значение стебля
Стебель. Внутреннее и внешнее строение стебля. Значение стебля Организменный уровень жизни, его роль в природе
Организменный уровень жизни, его роль в природе Строение и работа сердца. 8 класс
Строение и работа сердца. 8 класс Рост и развитие животных
Рост и развитие животных Биология кошек
Биология кошек Достижение селекции растений
Достижение селекции растений ОГЭ-ЕГЭ Биология. Зоология
ОГЭ-ЕГЭ Биология. Зоология Ес және назар
Ес және назар Сердечнососудистая система. Сердце
Сердечнососудистая система. Сердце Закономерности регуляции физиологических функций организма. Понятие физиологии. (Лекция 1)
Закономерности регуляции физиологических функций организма. Понятие физиологии. (Лекция 1) Основные направления эволюции
Основные направления эволюции Невидимый мир, работа с микроскопом
Невидимый мир, работа с микроскопом Семейство растений капустные
Семейство растений капустные Презентация педагогического опыта по теме Методологические основы интегрированных уроков
Презентация педагогического опыта по теме Методологические основы интегрированных уроков Зернобобові культури. Лекція 3
Зернобобові культури. Лекція 3 Биосоциальная сущность человека. Потребности
Биосоциальная сущность человека. Потребности 20231023_rasteniya_v_interere_doma
20231023_rasteniya_v_interere_doma тема Кораллы и Моллюски
тема Кораллы и Моллюски Бактериялардың көбеюі. Бактериялардың жыныстық, жыныссыз, вегетативті көбеюі. Споралардың түзілуі, формасы, төзімділігі
Бактериялардың көбеюі. Бактериялардың жыныстық, жыныссыз, вегетативті көбеюі. Споралардың түзілуі, формасы, төзімділігі В гости к весне
В гости к весне Ворон и ворона
Ворон и ворона Extinction of animals
Extinction of animals Квітка. Будова квітки
Квітка. Будова квітки Биологическое действие ионизирующих излучений
Биологическое действие ионизирующих излучений