Содержание
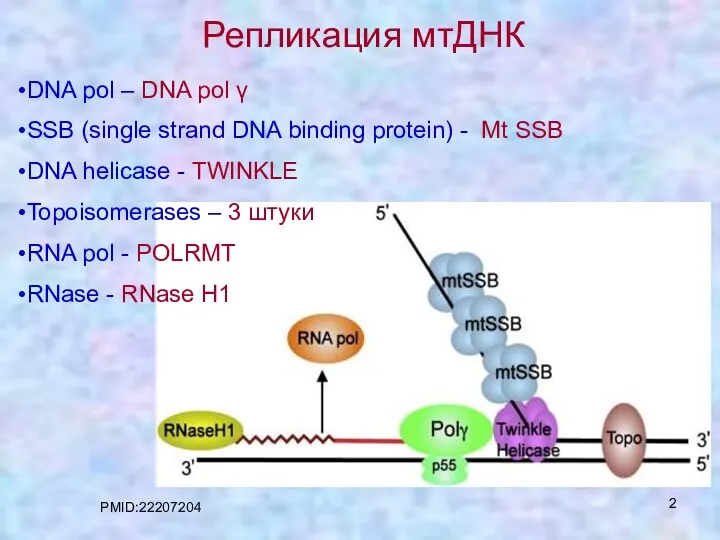
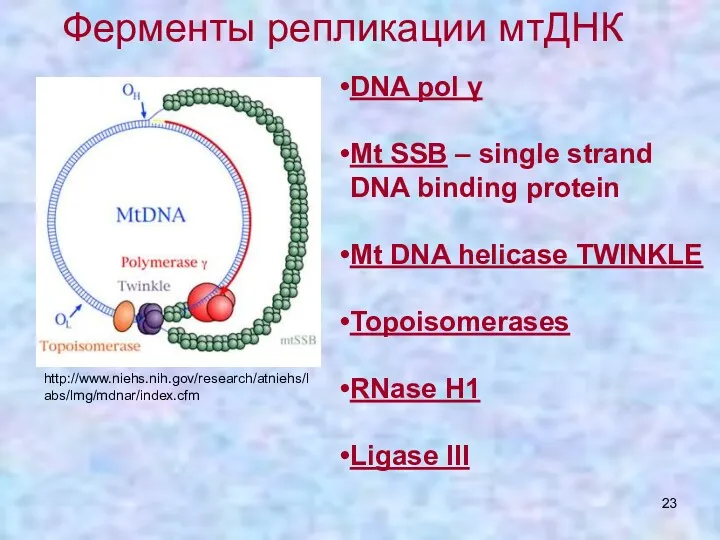
- 2. Репликация мтДНК DNA pol – DNA pol γ SSB (single strand DNA binding protein) - Mt
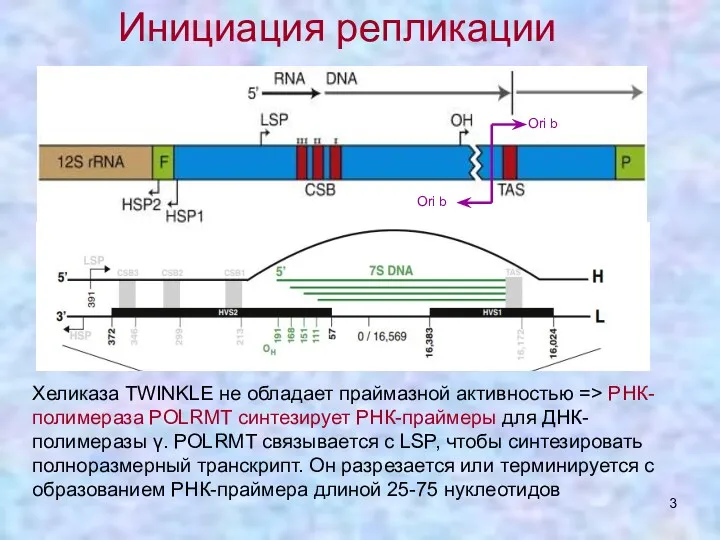
- 3. Хеликаза TWINKLE не обладает праймазной активностью => РНК-полимераза POLRMТ синтезирует РНК-праймеры для ДНК-полимеразы γ. POLRMT связывается
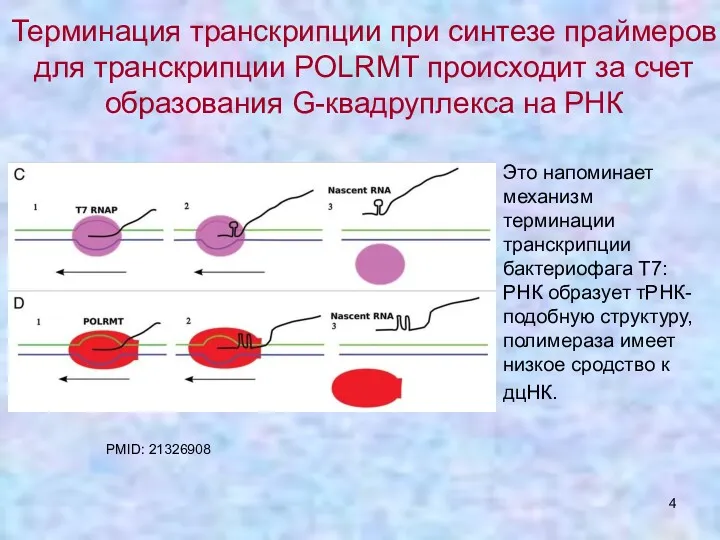
- 4. Это напоминает механизм терминации транскрипции бактериофага Т7: РНК образует тРНК-подобную структуру, полимераза имеет низкое сродство к
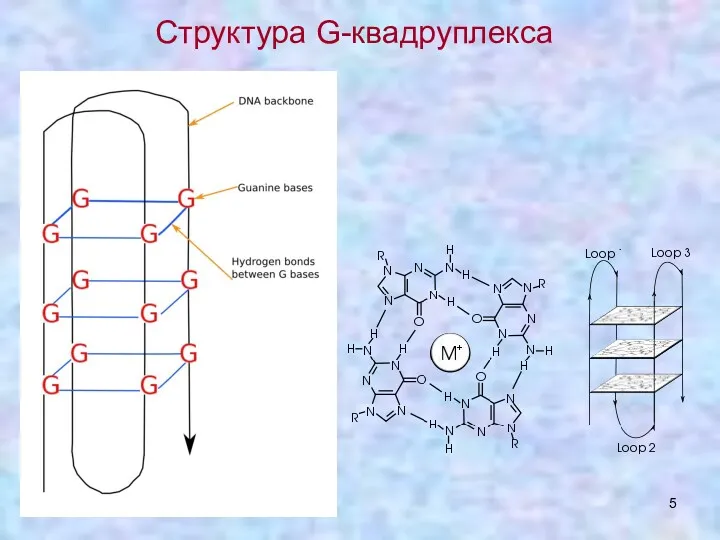
- 5. Структура G-квадруплекса
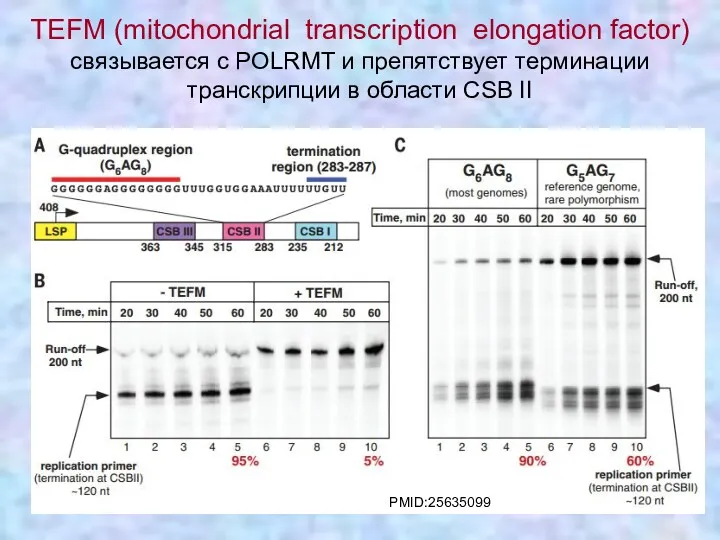
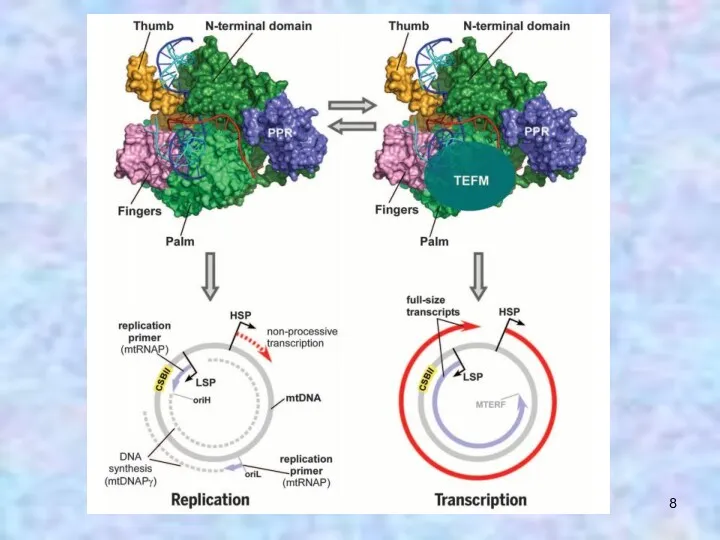
- 6. TEFM (mitochondrial transcription elongation factor) связывается с POLRMT и препятствует терминации транскрипции в области CSB II
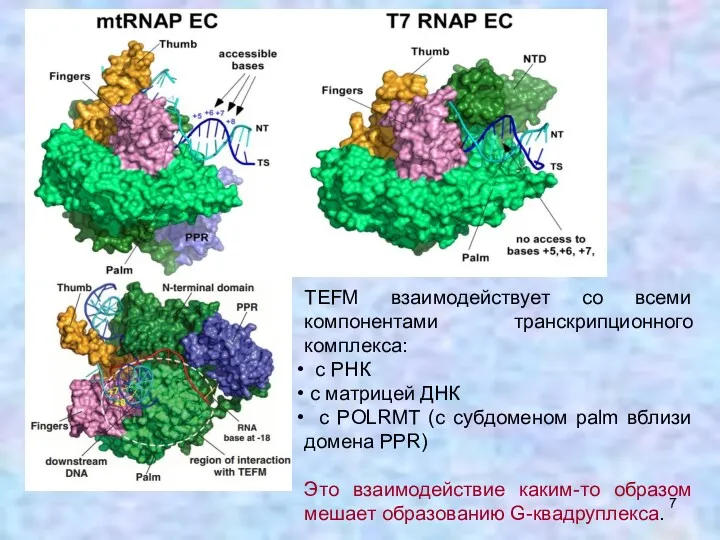
- 7. TEFM взаимодействует со всеми компонентами транскрипционного комплекса: с РНК с матрицей ДНК с POLRMT (c субдоменом
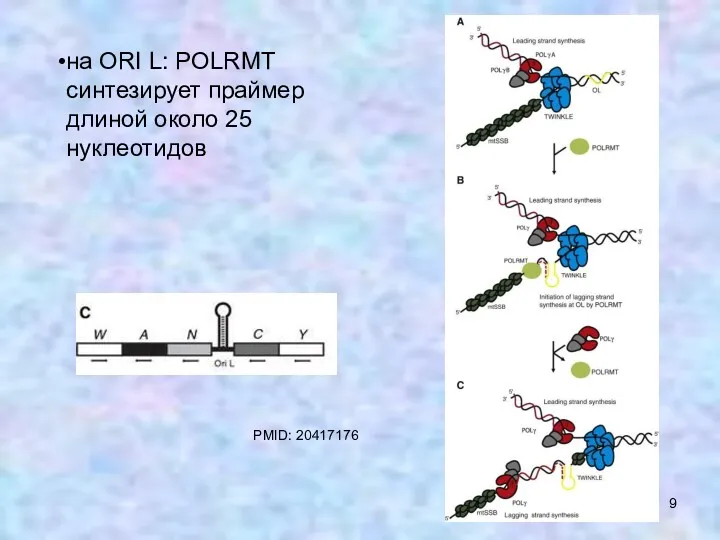
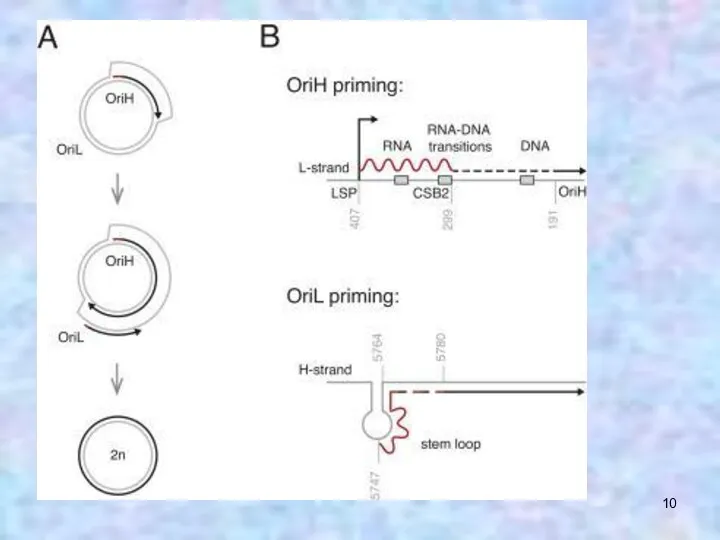
- 9. на ORI L: POLRMT синтезирует праймер длиной около 25 нуклеотидов PMID: 20417176
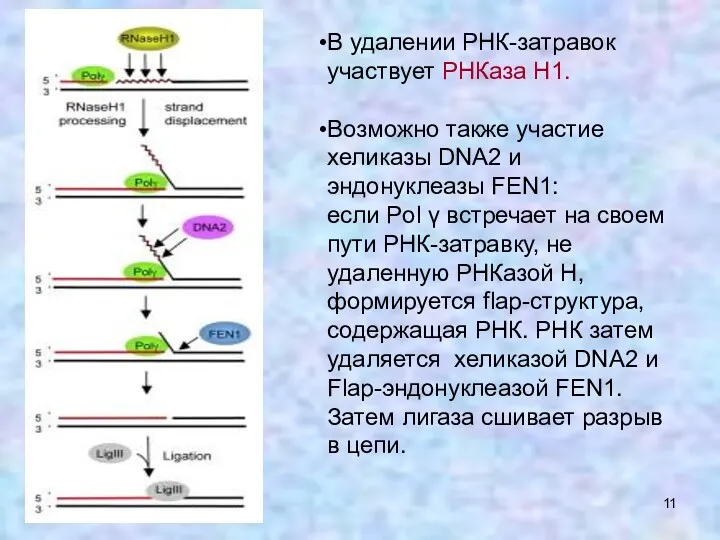
- 11. В удалении РНК-затравок участвует РНКаза Н1. Возможно также участие хеликазы DNA2 и эндонуклеазы FEN1: если Pol
- 12. При репликации РНК-праймеры для ДНК-полимеразы γ синтезирует РНК-полимераза POLRMТ. На ori Н синтез РНК-праймера начинается с
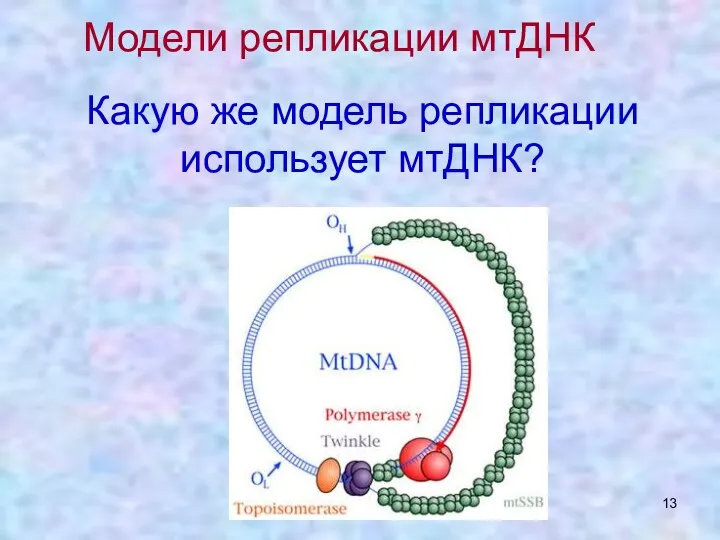
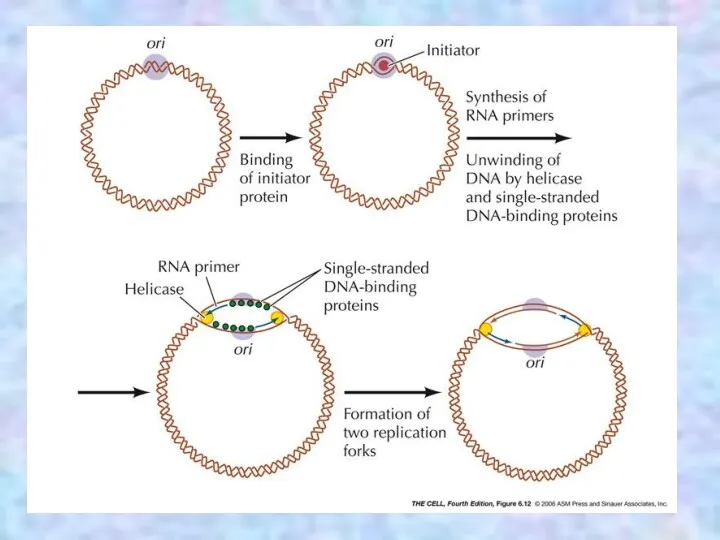
- 13. Какую же модель репликации использует мтДНК? Модели репликации мтДНК
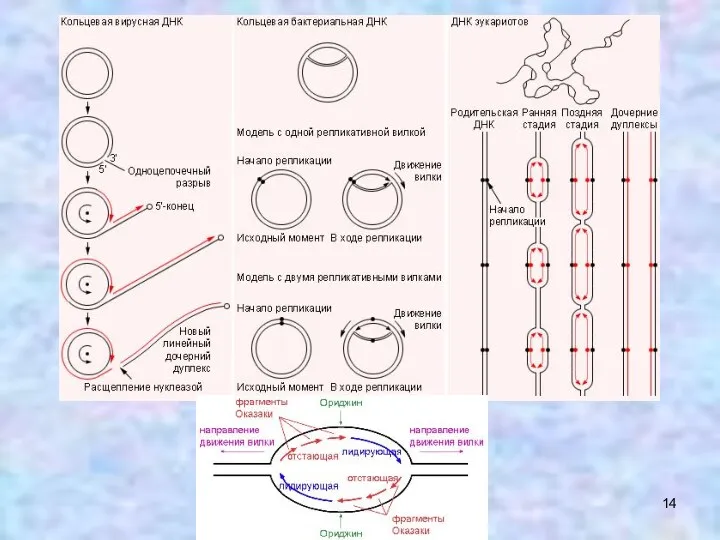
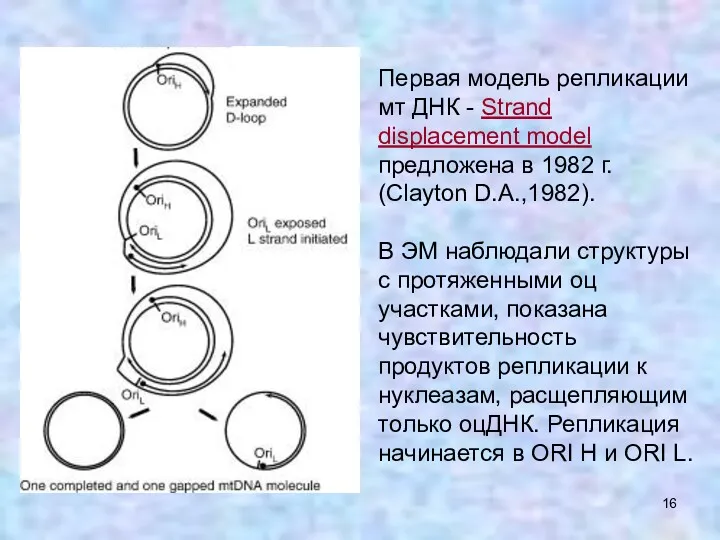
- 16. Первая модель репликации мт ДНК - Strand displacement model предложена в 1982 г. (Clayton D.A.,1982). В
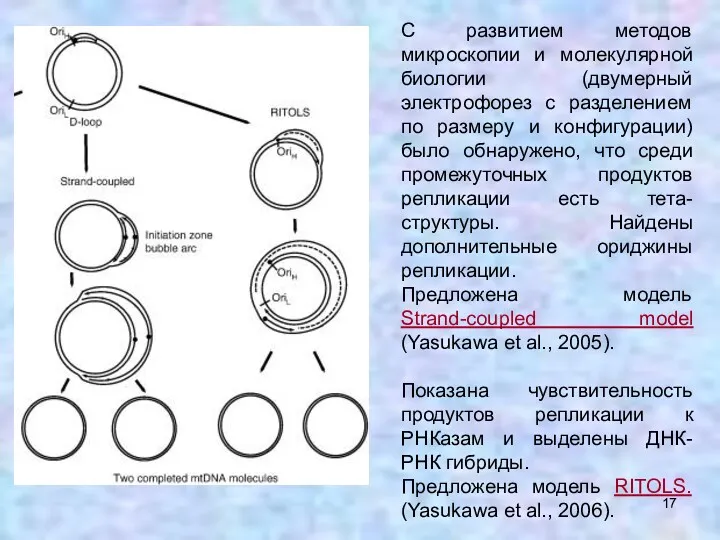
- 17. С развитием методов микроскопии и молекулярной биологии (двумерный электрофорез с разделением по размеру и конфигурации) было
- 18. Репликация инициируется вблизи ORIH, отстающая цепь состоит из РНК, затем заменяется на ДНК: Печень цыпленка: А+С
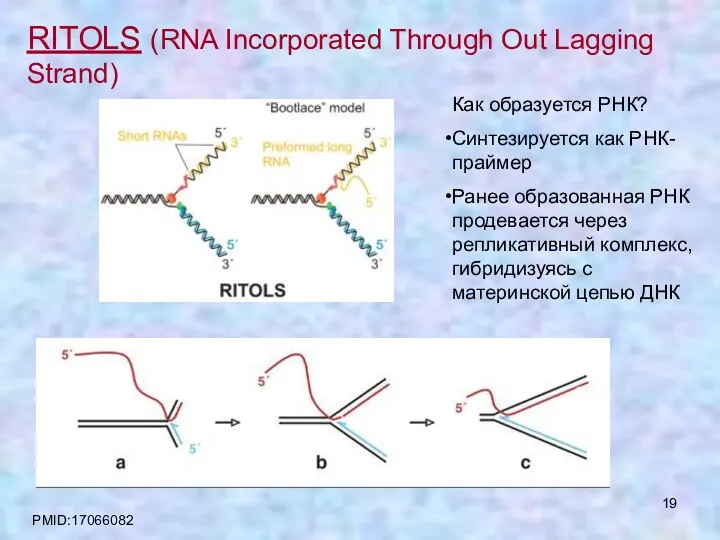
- 19. Как образуется РНК? Синтезируется как РНК-праймер Ранее образованная РНК продевается через репликативный комплекс, гибридизуясь с материнской
- 20. 1. Существует 3 модели репликации мтДНК: Strand displacement model – однонаправленный ассиметричный синтез с Ori Н,
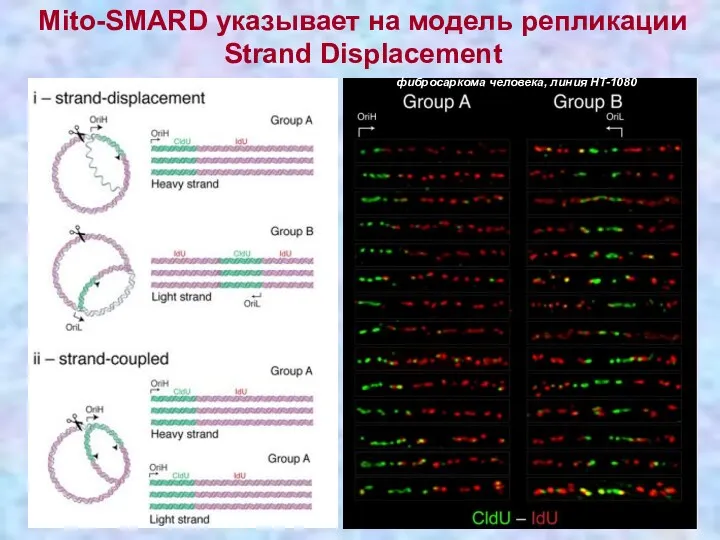
- 21. Mito-SMARD: Single-Molecule Analysis of Replicating mtDNA PMID:28111015 [IdU] - 5-Iodo-2‘-deoxyuridine [CldU]- 5-Chloro-2‘-deoxyuridine
- 22. Mito-SMARD указывает на модель репликации Strand Displacement фибросаркома человека, линия HT-1080
- 23. DNA pol γ Mt SSB – single strand DNA binding protein Mt DNA helicase TWINKLE Topoisomerases
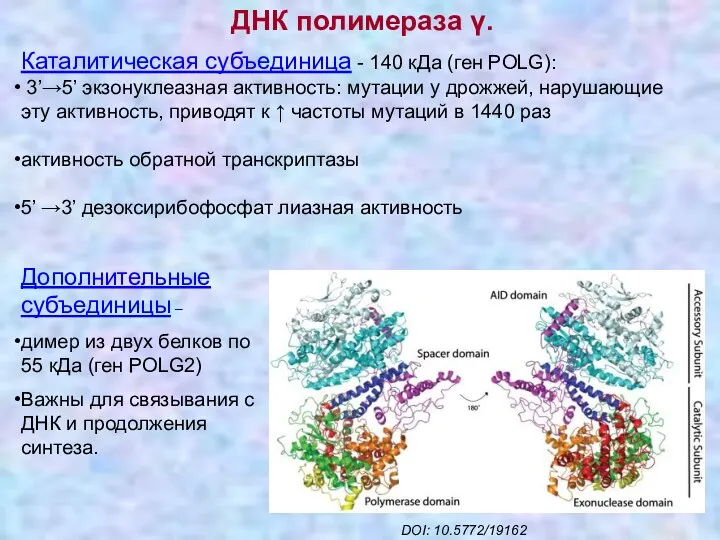
- 25. ДНК полимераза γ. Дополнительные субъединицы – димер из двух белков по 55 кДа (ген POLG2) Важны
- 26. Хроническая прогрессирующая офтальмоплегия (CPEO) – результат мутаций в генах POLG и POLG2.
- 27. N-концевой экзонуклеазный домен С-концевой полимеразный домен Спейсер: 2 субдомена – IP (intrinsic processivity) AID (accessory-interacting determinant)
- 28. 1) Проксимальная - ↑ аффинность связывания с ДНК 2) Дистальная – увеличивает скорость полимеразной реакции. Связан
- 29. В мтДНК присутствуют рибонуклеотиды: 10-30 rNTP на 500 нуклеотидов. Это возможно: Из-за неполного удаления РНК (модель
- 30. DNA pol γ способна проходить рибонуклеотиды при репликации, т.к. она обладает активностью обратной транскриптазы. Но на
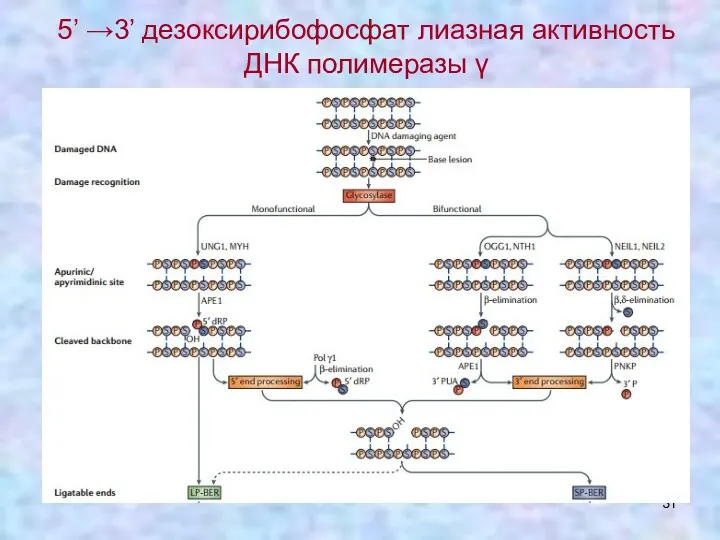
- 31. 5’ →3’ дезоксирибофосфат лиазная активность ДНК полимеразы γ
- 32. ДНК полимераза γ состоит из одной каталитической и двух дополнительных субъединиц Каталитическая субъединица имеет гомологию с
- 33. гомолог С-концевого участка хеликазы-праймазы фага T7. Содержит 5 хеликазных мотивов в С-концевом хеликазном домене. В N-концевом
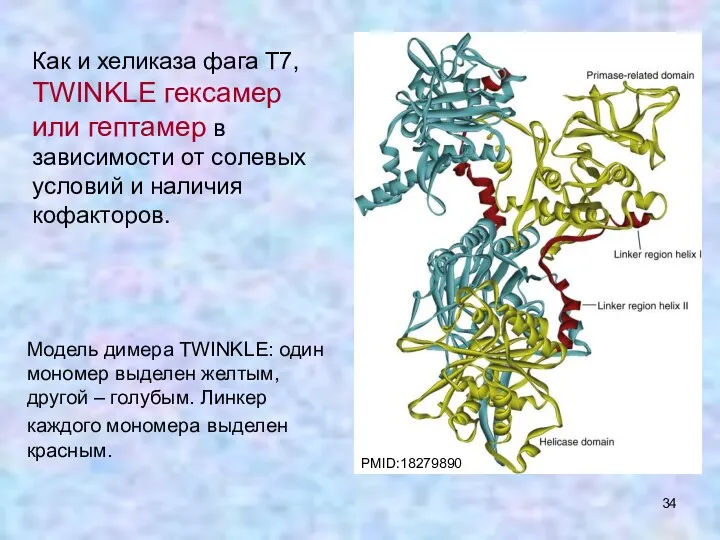
- 34. Как и хеликаза фага Т7, TWINKLE гексамер или гептамер в зависимости от солевых условий и наличия
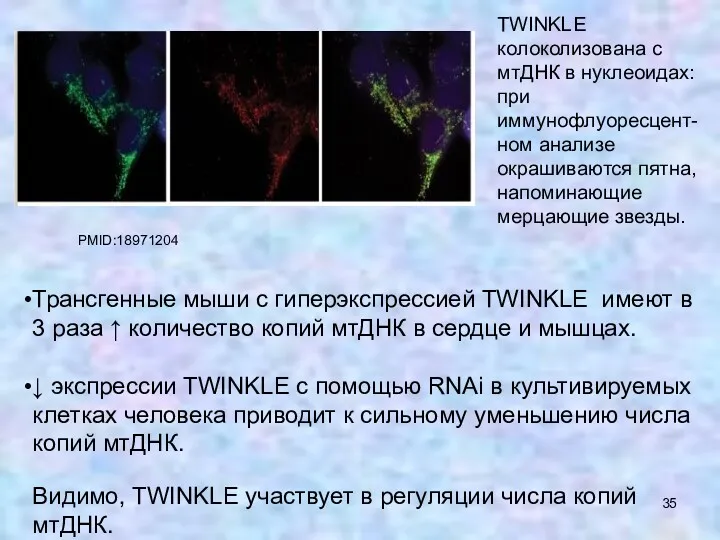
- 35. Трансгенные мыши с гиперэкспрессией TWINKLE имеют в 3 раза ↑ количество копий мтДНК в сердце и
- 36. Хеликазная активность стимулируется mtSSB, без него может расплетать только короткие субстраты. В присутствии mtSSB расплетает дцДНК
- 37. Выявление задержки репликативной вилки при помощи метода Mito-SMARD
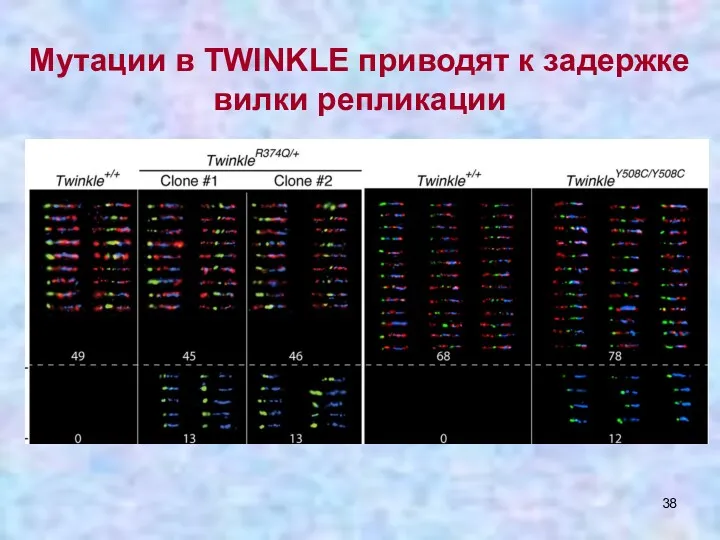
- 38. Мутации в TWINKLE приводят к задержке вилки репликации
- 39. Другие митохондриальные хеликазы hDNA2 – колокализована с мтДНК и TWINKLE в нуклеоидах. Колокализация с TWINKLE возрастает
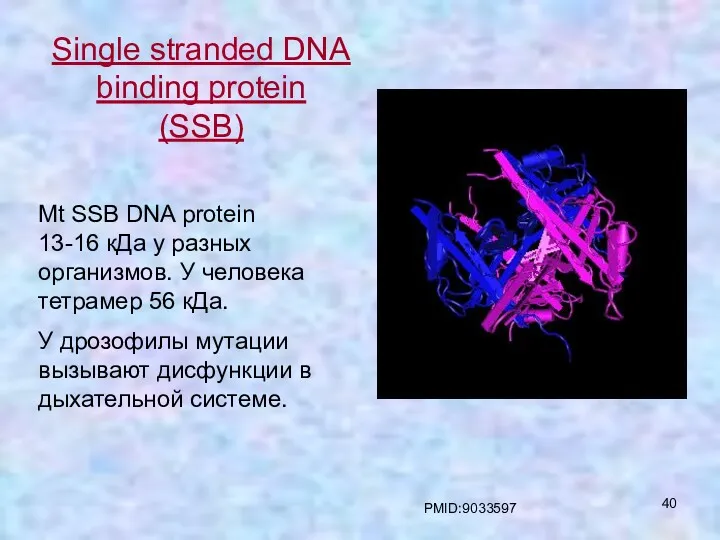
- 40. Mt SSB DNA protein 13-16 кДа у разных организмов. У человека тетрамер 56 кДа. У дрозофилы
- 41. Нокдаун mtSSB в клетках HeLa приводит к плавному снижению количества копий мтДНК и резкому снижению синтеза
- 42. mtSSB стимулирует активность TWINKLE и ДНК-полимеразы ɣ in vitro. Участки, важные для этой стимуляции разные. Это
- 43. Топоизомеразы Топоизомеразы вносят в ДНК разрыв, снимая супернапряжение внесенное за счет закрученности во время репликации и
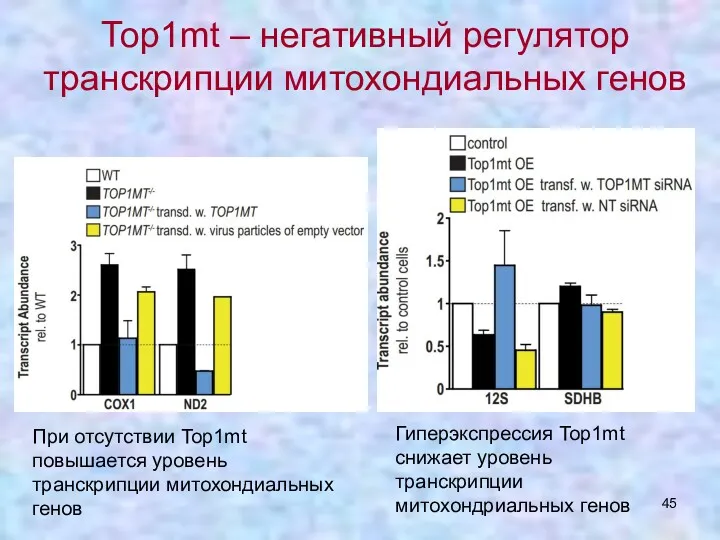
- 44. Тор1mt Такой же фермент, как в ядре, но с «митохондриальным адресом». Участвует в инициации транскрипции. Нокаутные
- 45. При отсутствии Top1mt повышается уровень транскрипции митохондиальных генов Гиперэкспрессия Top1mt снижает уровень транскрипции митохондриальных генов Тор1mt
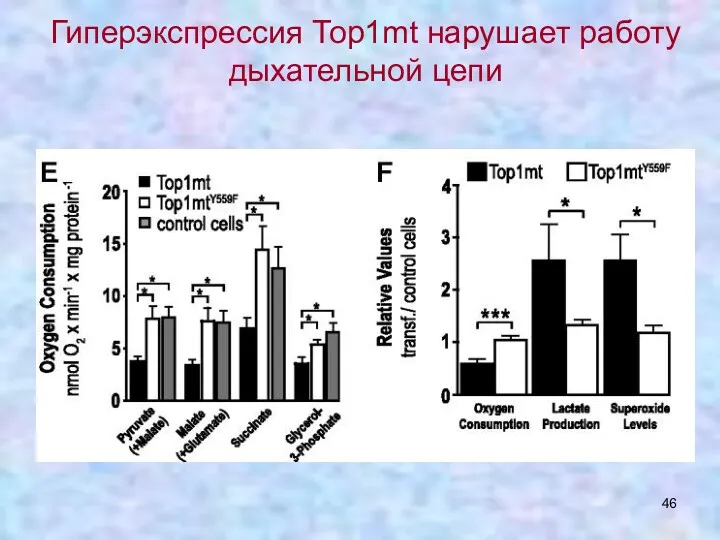
- 46. Гиперэкспрессия Тор1mt нарушает работу дыхательной цепи
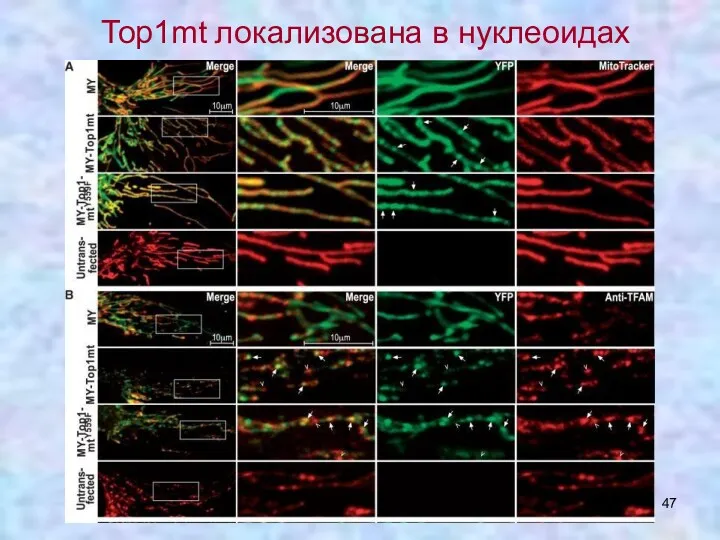
- 47. Тор1mt локализована в нуклеоидах
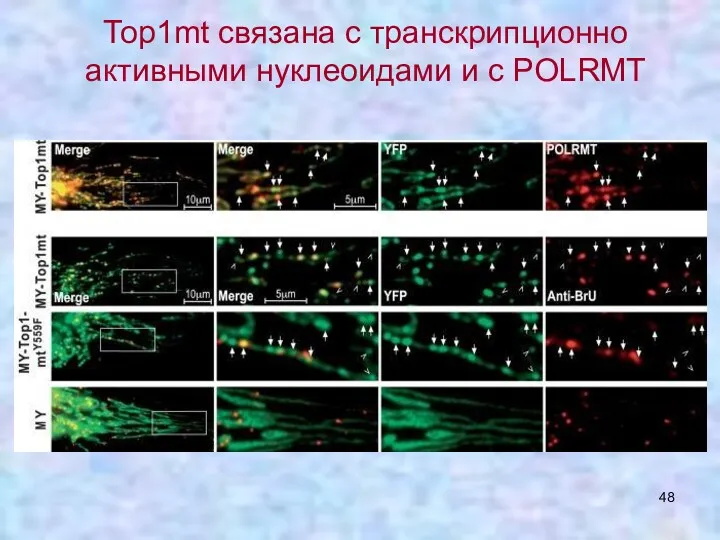
- 48. Тор1mt связана с транскрипционно активными нуклеоидами и с POLRMT
- 49. DNA topoisomerase IIIα PMID:12209014 Top3α – в ядре участвует в рекомбинации. Предполагается её участие в окончании
- 50. Top2B в ядре активирует транскрипцию. В митохондриях она может расплетать сплетенные в ходе репликации молекулы ДНК.
- 51. РНКазы Н RNase Н I – уничтожение РНК в РНК-ДНК гибридах RNase Н II – удаляет
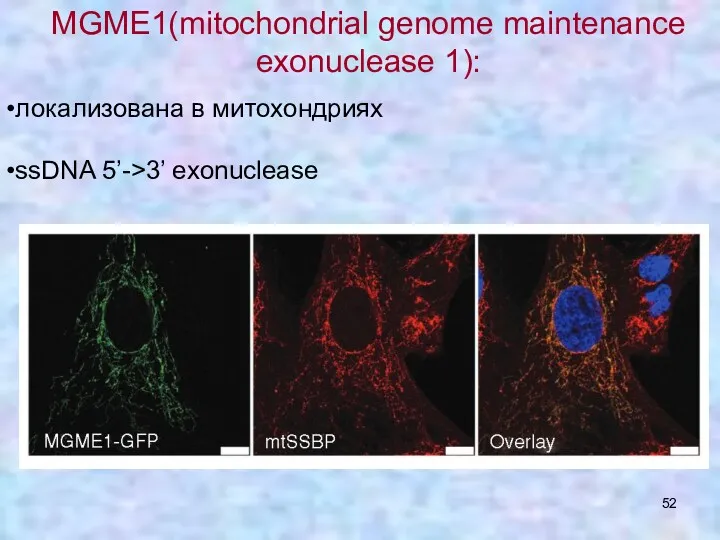
- 52. MGME1(mitochondrial genome maintenance exonuclease 1): локализована в митохондриях ssDNA 5’->3’ exonuclease
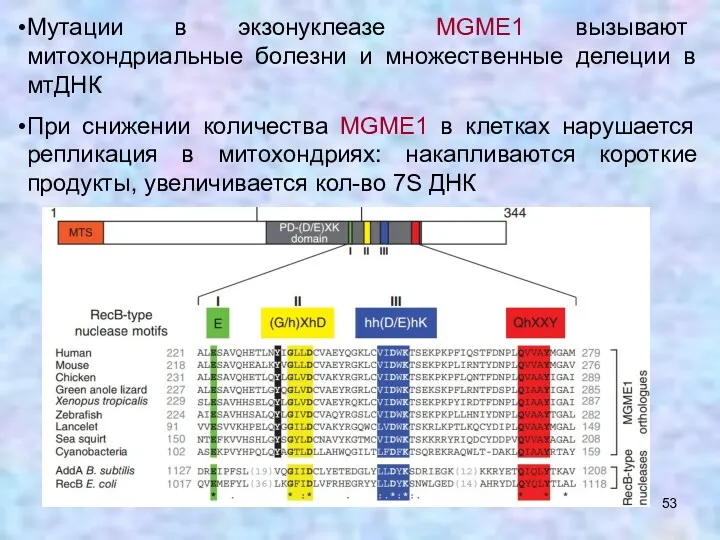
- 53. Мутации в экзонуклеазе MGME1 вызывают митохондриальные болезни и множественные делеции в мтДНК При снижении количества MGME1
- 55. Скачать презентацию



![Mito-SMARD: Single-Molecule Analysis of Replicating mtDNA PMID:28111015 [IdU] - 5-Iodo-2‘-deoxyuridine [CldU]- 5-Chloro-2‘-deoxyuridine](/_ipx/f_webp&q_80&fit_contain&s_1440x1080/imagesDir/jpg/277792/slide-20.jpg)


















 Гортань как орган фонации
Гортань как орган фонации Родственные браки
Родственные браки Биология – наука о живых организмах
Биология – наука о живых организмах Урок по биологии 8 класс Строение органа слуха
Урок по биологии 8 класс Строение органа слуха о вреде мобильных телефонов
о вреде мобильных телефонов зрительный_анализатор
зрительный_анализатор Ноосфера
Ноосфера Почему осенью осыпаются листья
Почему осенью осыпаются листья Закон гомологических рядов в наследственной изменчивости
Закон гомологических рядов в наследственной изменчивости Компоненты организма человека
Компоненты организма человека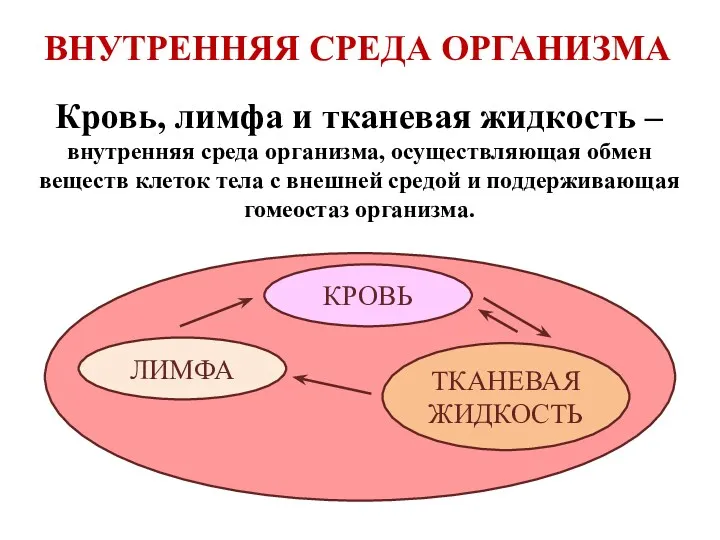 Внутренняя среда организма: кровь, лимфа и тканевая жидкость
Внутренняя среда организма: кровь, лимфа и тканевая жидкость Газообмен в лёгких. Внутреннее дыхание
Газообмен в лёгких. Внутреннее дыхание Птичьи посиделки
Птичьи посиделки Раздел Билатеральносимметричные (Bilateria). Подраздел Бесполостные (Acoelomata)
Раздел Билатеральносимметричные (Bilateria). Подраздел Бесполостные (Acoelomata) Генетика пола
Генетика пола Стебель, его строение и значение
Стебель, его строение и значение Фитодизайн школьного кабинета биологии
Фитодизайн школьного кабинета биологии Характеристика класса птицы. Внешнее строение птиц
Характеристика класса птицы. Внешнее строение птиц Урок-практикум Опорно-двигательная система
Урок-практикум Опорно-двигательная система Митоз. Апоптоз. Канцерогенез
Митоз. Апоптоз. Канцерогенез Морфология и строение вирусов
Морфология и строение вирусов Божья коровка
Божья коровка Чорноморський біосферний заповідник
Чорноморський біосферний заповідник Зовнішня та внутрішня будова лягушки
Зовнішня та внутрішня будова лягушки Семейство Гвоздичные
Семейство Гвоздичные Красные водоросли
Красные водоросли Презентация к уроку биологии в 7 классе Особенности организации одноклеточных, их классификация
Презентация к уроку биологии в 7 классе Особенности организации одноклеточных, их классификация Антропогенез. Происхождение человечества
Антропогенез. Происхождение человечества