Неконтролируемое размножение генно-модифицированных деревьев - эпический провал биоинженеров презентация
Содержание
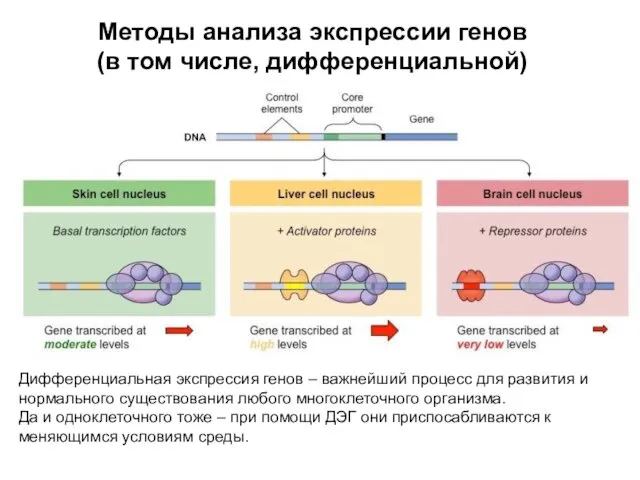
- 2. Методы анализа экспрессии генов (в том числе, дифференциальной) Дифференциальная экспрессия генов – важнейший процесс для развития
- 3. Как можно измерить экспрессию генов? По количеству РНК По количеству белка Какой подход вам нравится больше?
- 4. С одной стороны, по белку судить корректней – это же финальный продукт экспрессии генов. Но возникает
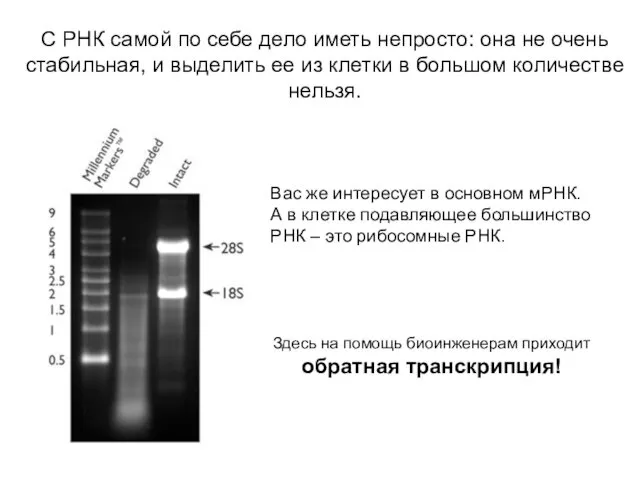
- 5. С РНК самой по себе дело иметь непросто: она не очень стабильная, и выделить ее из
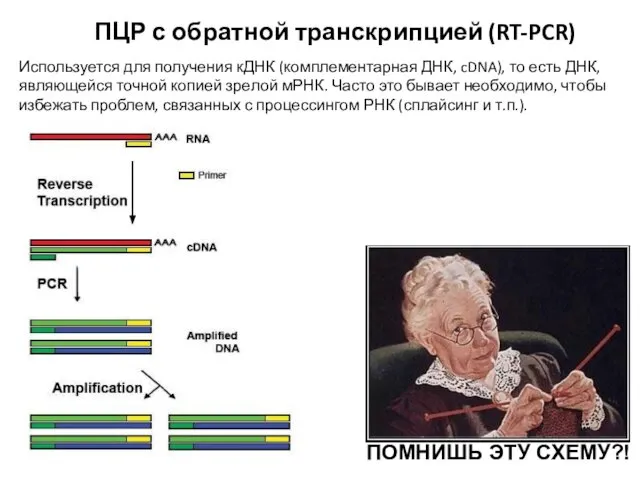
- 6. ПЦР с обратной транскрипцией (RT-PCR) Используется для получения кДНК (комплементарная ДНК, cDNA), то есть ДНК, являющейся
- 7. ПРОБЛЕМЫ СИНТЕЗА СУММАРНОЙ кДНК • Для выделения полиА+ фракции нужно значительное количество РНК (>100 мкг). •
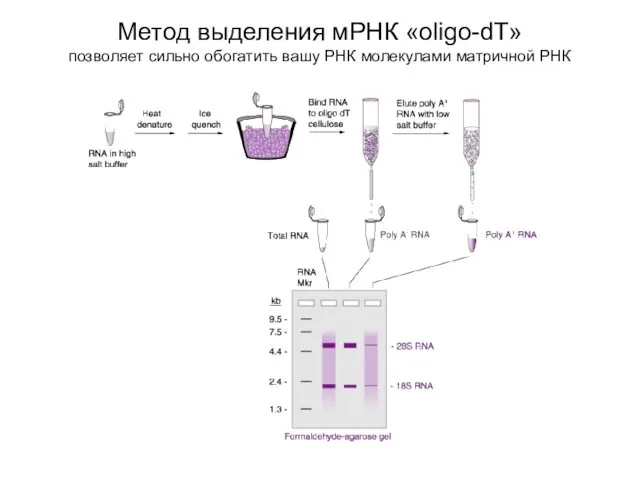
- 8. Метод выделения мРНК «oligo-dT» позволяет сильно обогатить вашу РНК молекулами матричной РНК
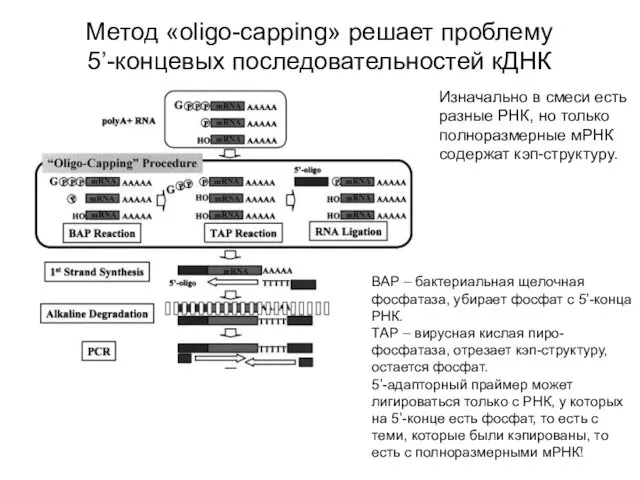
- 9. Метод «oligo-capping» решает проблему 5’-концевых последовательностей кДНК BAP – бактериальная щелочная фосфатаза, убирает фосфат с 5’-конца
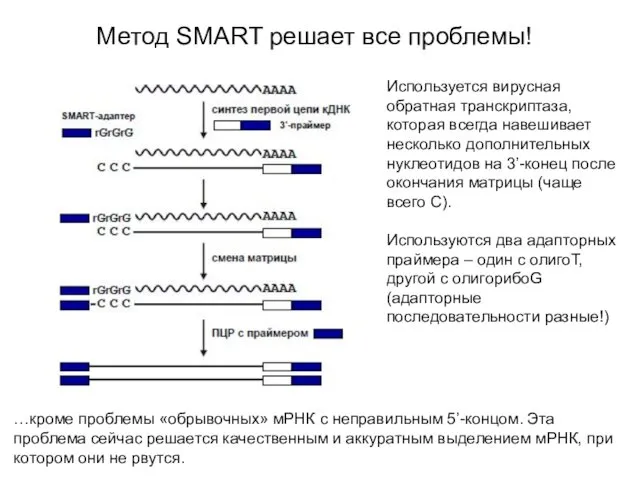
- 10. Метод SMART решает все проблемы! Используется вирусная обратная транскриптаза, которая всегда навешивает несколько дополнительных нуклеотидов на
- 11. Хорошо. Выделили мРНК, амплифицировали кДНК. А мы вроде как собирались экспрессию генов смотреть. Этот человек как
- 12. EST (Expressed Sequence Tags) Метод массово использовался для анализа экспрессии генов до начала эпохи NGS. Подготовьте
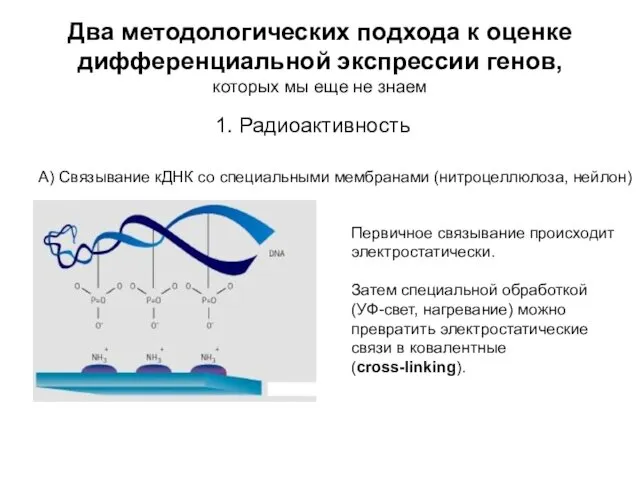
- 13. Два методологических подхода к оценке дифференциальной экспрессии генов, которых мы еще не знаем 1. Радиоактивноcть А)
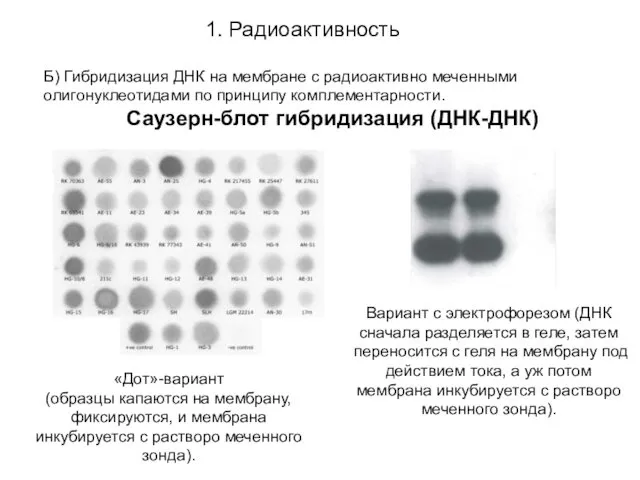
- 14. 1. Радиоактивноcть Б) Гибридизация ДНК на мембране с радиоактивно меченными олигонуклеотидами по принципу комплементарности. Саузерн-блот гибридизация
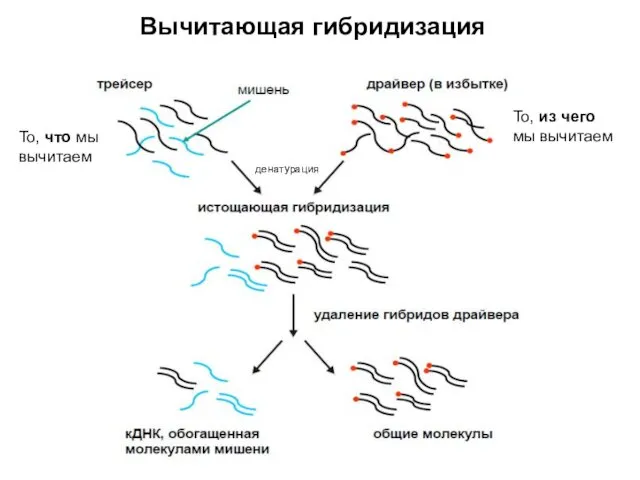
- 15. Вычитающая гибридизация денатурация То, что мы вычитаем То, из чего мы вычитаем
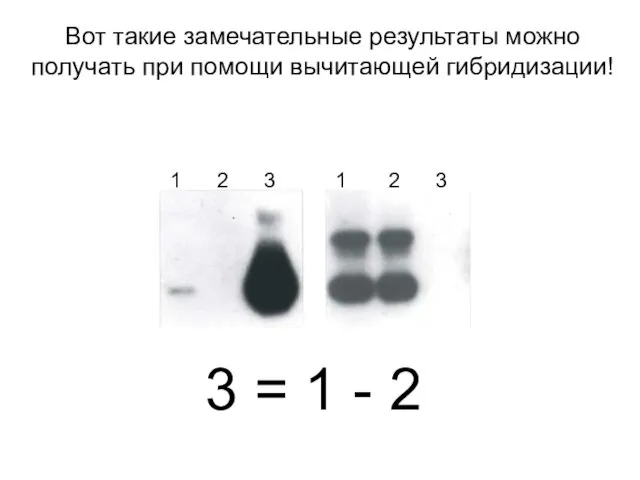
- 16. Вот такие замечательные результаты можно получать при помощи вычитающей гибридизации! 3 = 1 - 2
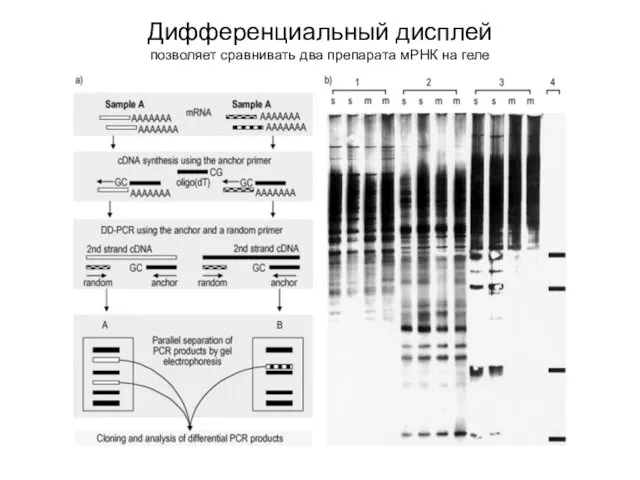
- 17. Дифференциальный дисплей позволяет сравнивать два препарата мРНК на геле
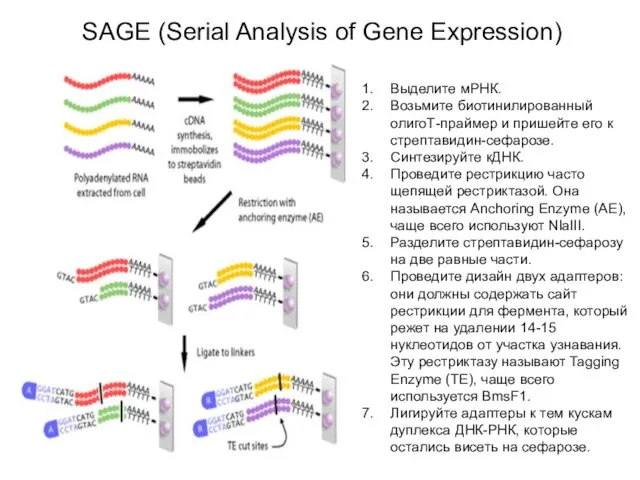
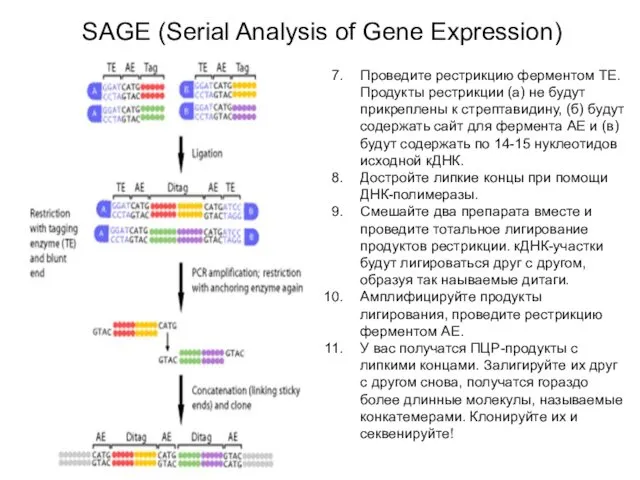
- 18. SAGE (Serial Analysis of Gene Expression) Выделите мРНК. Возьмите биотинилированный олигоТ-праймер и пришейте его к стрептавидин-сефарозе.
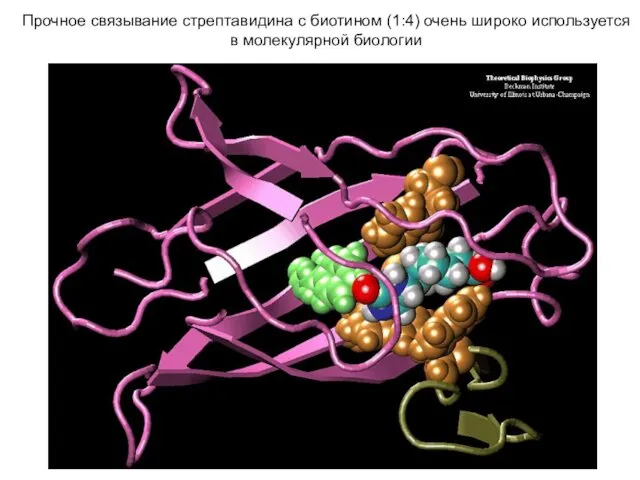
- 19. Прочное связывание стрептавидина с биотином (1:4) очень широко используется в молекулярной биологии
- 20. SAGE (Serial Analysis of Gene Expression) Проведите рестрикцию ферментом TE. Продукты рестрикции (а) не будут прикреплены
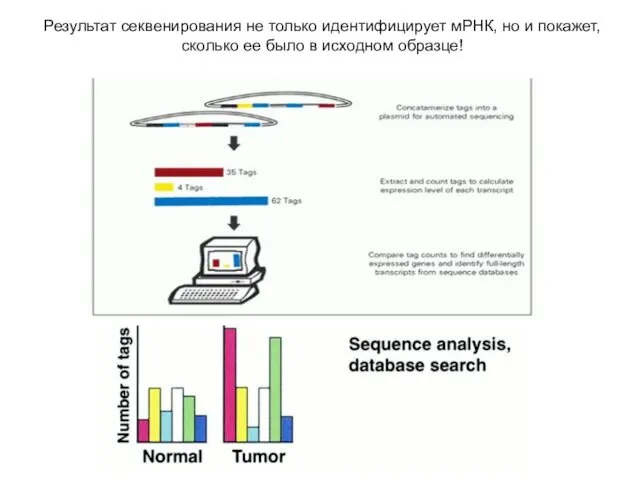
- 21. Результат секвенирования не только идентифицирует мРНК, но и покажет, сколько ее было в исходном образце!
- 22. Технологии ДНК-чипов Микрочип – это, как вы знаете, компьютерная микросхема. А ДНК-чип – это некая совокупность
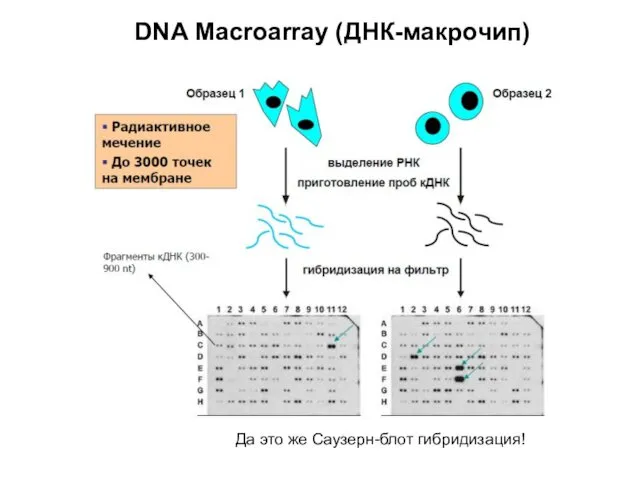
- 23. DNA Macroarray (ДНК-макрочип) Да это же Саузерн-блот гибридизация!
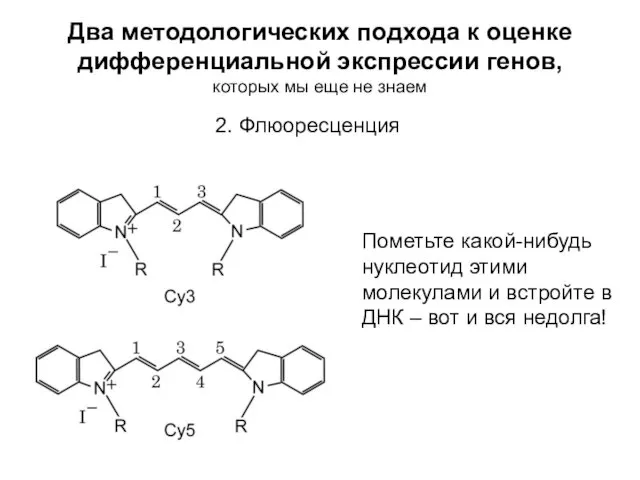
- 24. 2. Флюоресценция Пометьте какой-нибудь нуклеотид этими молекулами и встройте в ДНК – вот и вся недолга!
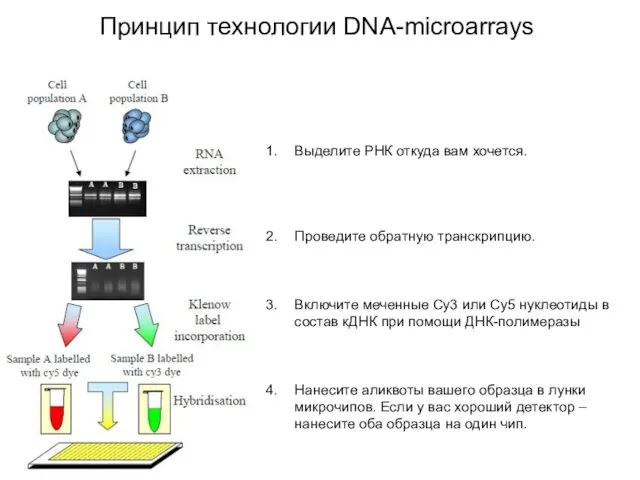
- 25. Принцип технологии DNA-microarrays Выделите РНК откуда вам хочется. Проведите обратную транскрипцию. Включите меченные Cy3 или Cy5
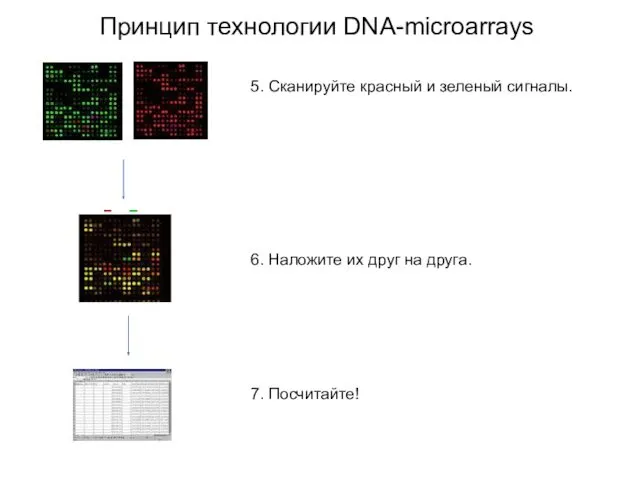
- 26. Принцип технологии DNA-microarrays 5. Сканируйте красный и зеленый сигналы. 6. Наложите их друг на друга. 7.
- 27. Анализ экспрессии дрожжевых генов в зависимости от типа питания (аэробное / анаэробное)
- 28. Cy3 Cy5 Cy5 Cy3 log2 Анализ результатов использования ДНК-чипов Наложите друг на друга картинки с Cy3
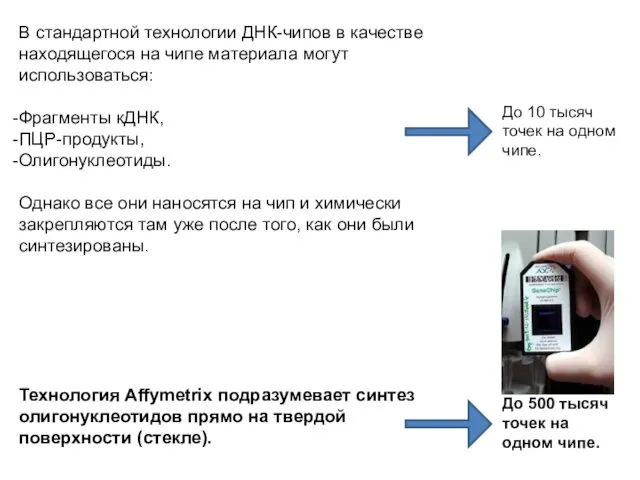
- 29. В стандартной технологии ДНК-чипов в качестве находящегося на чипе материала могут использоваться: Фрагменты кДНК, ПЦР-продукты, Олигонуклеотиды.
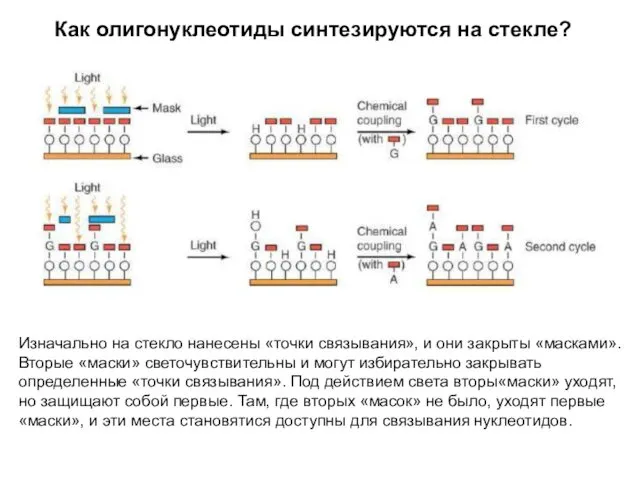
- 30. Как олигонуклеотиды синтезируются на стекле? Изначально на стекло нанесены «точки связывания», и они закрыты «масками». Вторые
- 32. Скачать презентацию








 Сестринский процесс при хроническом энтероколите
Сестринский процесс при хроническом энтероколите Методы синтеза и анализа цифровых фильтров. Введение
Методы синтеза и анализа цифровых фильтров. Введение Systemy transportowe – transport globalny
Systemy transportowe – transport globalny Колонизация Сибири русским народом
Колонизация Сибири русским народом Дедуктивные выводы из сложных суждений. Логика
Дедуктивные выводы из сложных суждений. Логика Михаил Васильевич Ломоносов
Михаил Васильевич Ломоносов Вербальная модель в сервисе
Вербальная модель в сервисе Глаз, как оптическая система
Глаз, как оптическая система 2015.03.20.Open house.Odesa.Application
2015.03.20.Open house.Odesa.Application Паровой двигатель
Паровой двигатель Организация объединенных наций (ООН)
Организация объединенных наций (ООН) Пластиковый Апокалипсис
Пластиковый Апокалипсис Развитие коммуникативных игр
Развитие коммуникативных игр Мини Мастер конструктор керамический Печь для итальянской пиццы, как настоящий, 1 : 10
Мини Мастер конструктор керамический Печь для итальянской пиццы, как настоящий, 1 : 10 Литосфера. Основные особенности литосферы. Влияние деятельности человека
Литосфера. Основные особенности литосферы. Влияние деятельности человека Дикорастущие плодовые и орехоплодные растения разных типов леса, их запасы и заготовка
Дикорастущие плодовые и орехоплодные растения разных типов леса, их запасы и заготовка Стипендия. Виды стипендий
Стипендия. Виды стипендий Классификация извещателей пожарных (ИП)
Классификация извещателей пожарных (ИП) Манометры и гидравлическое оборудование
Манометры и гидравлическое оборудование Презентация Стихийные бедствия 2 часть
Презентация Стихийные бедствия 2 часть Числа 3 и 8 (1 класс)
Числа 3 и 8 (1 класс) Увольнение с военной службы
Увольнение с военной службы Интерактивный тренажер Процент от числа
Интерактивный тренажер Процент от числа Материалы и изделия из древесины. Тема 2
Материалы и изделия из древесины. Тема 2 Кишечный шов
Кишечный шов Мотивация учения- основное условие успешного обучения Выступление на педсовете
Мотивация учения- основное условие успешного обучения Выступление на педсовете Салтыков – Щедрин История одного города
Салтыков – Щедрин История одного города Строительство высокоскоростной магистрали ВСМ-2 Москва-Казань
Строительство высокоскоростной магистрали ВСМ-2 Москва-Казань