Содержание
- 2. Банки (бази) данних – це: Колекції структурованих індексованих ? дає можливість проведення пошуку за заданими критеріями
- 3. Банки данних обов’язково містять в себе також набір програмних інструментів, які забезпечують : доступ до банка
- 4. Найчастіше, та сама інформація існує в різних форматах у різних базах даних, і різні сервери надають
- 5. MCB, 6 sep 2004 EMBnet Еволюція баз даних Books, articles 1968 -> 1985 Computer tapes 1982
- 6. Всі існуючі БД можуть бути класифіковані певним чином, зокрема їх підрозділяють: на первинні та вторинні (похідні)
- 7. Архівні БД характеризуються тим, що вся відповідальність за інформацію, яка міститься в цих базах, лежить на
- 8. Вміст записів курованих БД визначається спеціальними експертами (кураторами), які безпосередньо формують інформаційне наповнення цих банків даних.
- 9. Вміст автоматичних БД, як видно з назви, генерується за допомогою комп‘ютерних програм і веб-сервісів на основі
- 10. Інтегровані бази даних містять різнорідну інформацію (архівну, куровану, згенеровану автоматично), яка підбирається за принципом систематизованого опису
- 11. Під первинними базами даних, як правило, розуміють бази, які містять безпосередні результати молекулярно-біологічних експериментів, зокрема дані
- 12. Вторинні або, похідні БД містять т.зв. процесовану інформацію, тобто, інформацію, яка виникає в результаті обробки і
- 13. Незалежно від типу банку даних, записи/статті банку містять певні поля (метадані), що дозволяють індексувати вміст банка
- 14. Accession Number ? унікальний ідентифікатор статті, дозволяє формувати швидкі запити до неї Source та\або Taxonomy ?
- 15. Основные биоинформатические базы данных Основные БД последовательностей: EMBL, GeneBank, UniProt, SwissProt. Производные PFAM,PROSITE, INTERPRO, dbEST, dbSNP…….
- 16. MCB, 6 sep 2004 EMBnet Categories of databases for Life Sciences Sequences (DNA, protein) Genomics Mutation/polymorphism
- 17. MCB, 6 sep 2004 EMBnet Sequence databases DNA/RNA Proteins
- 18. MCB, 6 sep 2004 EMBnet Ideal minimal content of a sequence database entry Sequences !! Accession
- 19. MCB, 6 sep 2004 EMBnet Sequence Databases: some « technical » definitions Data storage management: flat
- 20. Sequence database : example ID EPO_HUMAN STANDARD; PRT; 193 AA. AC P01588; Q9UHA0; Q9UEZ5; Q9UDZ0; DT
- 21. MCB, 6 sep 2004 EMBnet Sequence database: example (cont.) FT SIGNAL 1 27 FT CHAIN 28
- 22. MCB, 6 sep 2004 EMBnet Sequence database: example …The fasta format: > My_Sequence_Name MGVHECPAWLWLLLSLLSLPLGLPVLGAPPRLICDSRVLERYLLEAKEAE NITTGCAEHCSLNENITVPDTKVNFYAWKRMEVGQQAVEVWQGLALLSEA VLRGQALLVNSSQPWEPLQLHVDKAVSGLRSLTTLLRALGAQKEAISPPD
- 23. MCB, 6 sep 2004 EMBnet Database 1: nucleotide sequences The 3 main public nucleic acid sequence
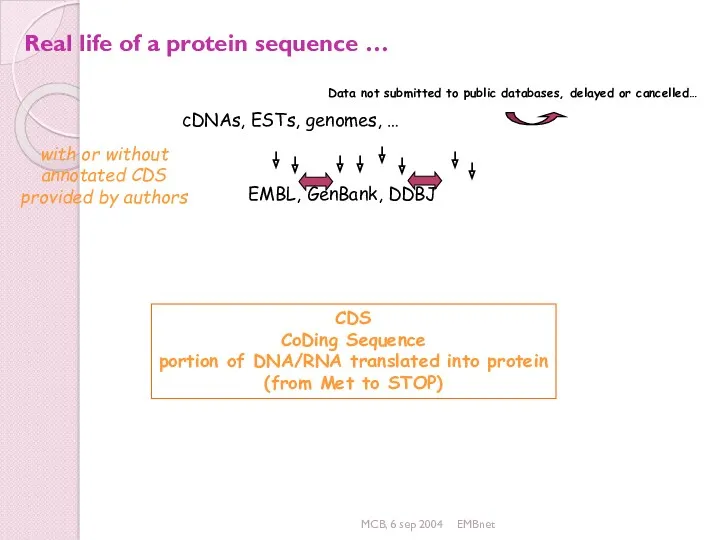
- 24. MCB, 6 sep 2004 EMBnet Real life of a protein sequence … cDNAs, ESTs, genomes, …
- 25. MCB, 6 sep 2004 EMBnet Serve as archives Contain all public sequences derived from: Genome projects
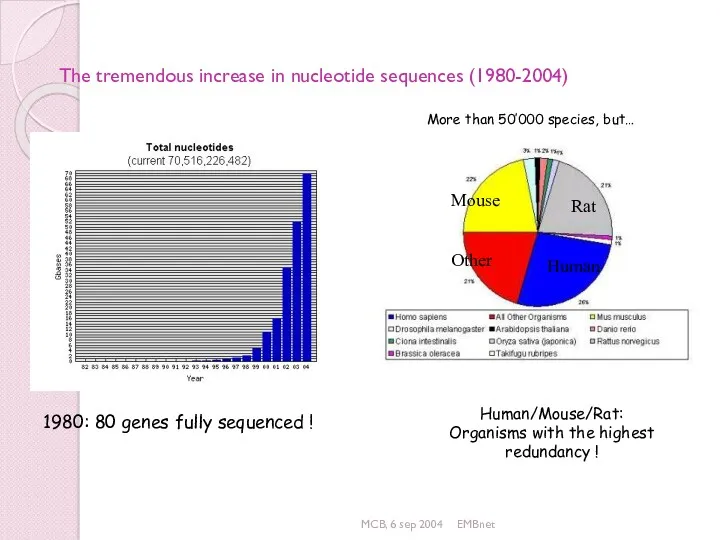
- 26. MCB, 6 sep 2004 EMBnet Human/Mouse/Rat: Organisms with the highest redundancy ! The tremendous increase in
- 27. MCB, 6 sep 2004 EMBnet CC Data kindly reviewed (24-FEB-1986) by K. Jacobs FH Key Location/Qualifiers
- 28. MCB, 6 sep 2004 EMBnet EMBL/GenBank/DDBJ Sort of sequence museum, where sequences are preserved for eternity
- 29. EMBL/GenBank/DDBJ Unexpected information you can find in these db: FT source 1..124 FT /db_xref="taxon:4097" FT /organelle="plastid:chloroplast"
- 30. The second generation of nucleotide sequence databases Gene-centric databases All the sequence information relevant to a
- 31. MCB, 6 sep 2004 EMBnet Working with whole genome databases: Genome-centric databases « Browsing resources »
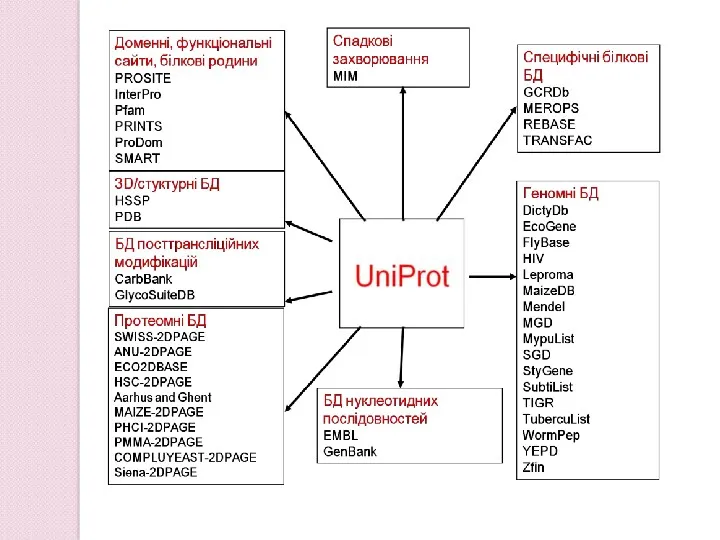
- 33. MCB, 6 sep 2004 EMBnet Database 2: protein sequences UNIPROT: PIR-PSD: Protein Information Resources -> UniProt
- 34. MCB, 6 sep 2004 EMBnet Swiss-Prot -> ExPASy (www.expasy.org); TrEMBL -> EBI (European Bioinformatics Institute) (www.ebi.ac.uk/trembl/).
- 35. MCB, 6 sep 2004 EMBnet In a UniProt entry, you can expect to find: All the
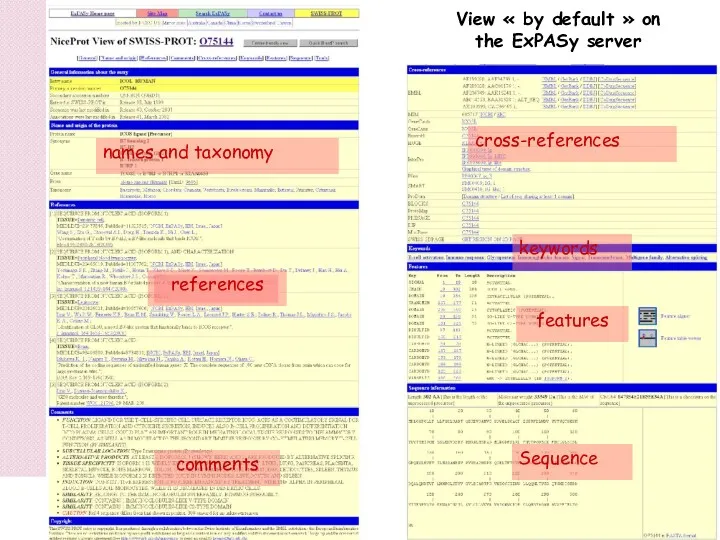
- 36. MCB, 6 sep 2004 EMBnet View « by default » on the ExPASy server comments features
- 37. MCB, 6 sep 2004 EMBnet Annotation/Curation (Comment lines) Function(s) and role(s); enzymes: a. Catalytic activity (if
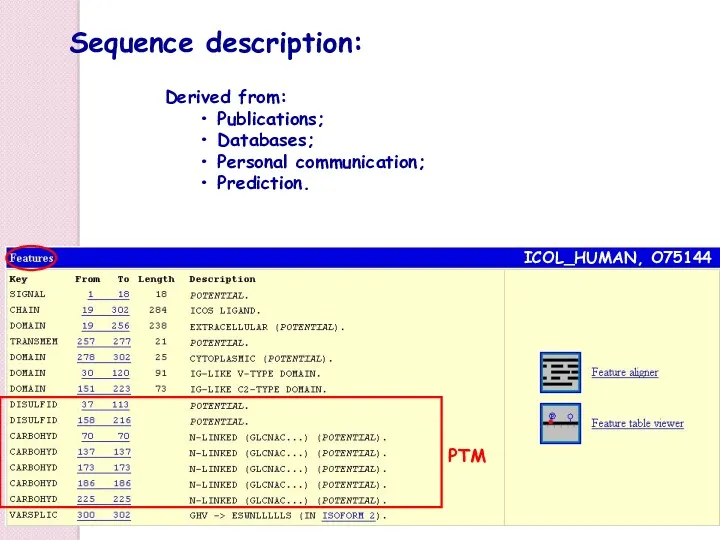
- 38. MCB, 6 sep 2004 EMBnet Information is derived from: Publications; Databases; Personal communication; Prediction; Brain storming…
- 40. MCB, 6 sep 2004 EMBnet Cross-references ADN (Index of low redundancy) ICE8_HUMAN Q14790
- 41. ICOL_HUMAN, O75144
- 42. MCB, 6 sep 2004 EMBnet Databases 3: ‘genomics’ Contain informations on gene chromosomal location (mapping) and
- 43. Databases 4: mutation/polymorphism Contain informations on sequence variations linked or not to genetic diseases; Mainly human
- 44. MCB, 6 sep 2004 EMBnet Mutation/polymorphism: definitions SNPs: single nucleotide polymorphisms; occur approximately once every 100
- 45. MCB, 6 sep 2004 EMBnet Database 5: protein domain/family
- 46. MCB, 6 sep 2004 EMBnet Protein domain/family: some definitions Most proteins have « modular » structures
- 47. Protein domain/family: some definitions Domains (conserved sequences or structures) are identified by multiple sequence alignments Domains
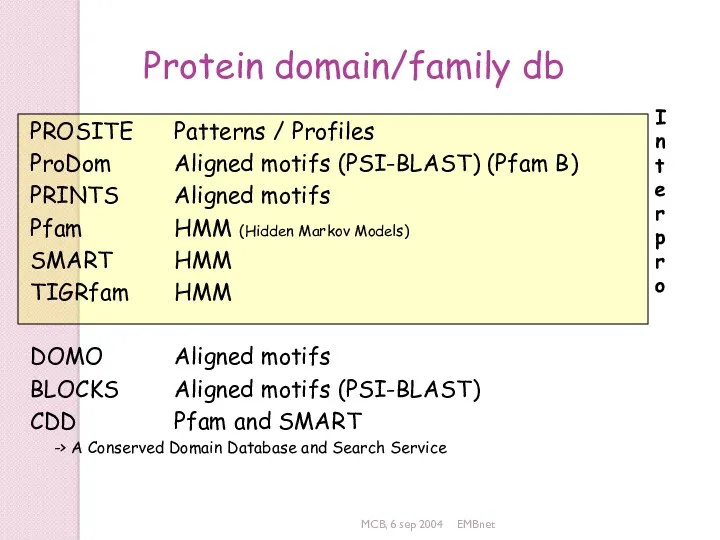
- 48. MCB, 6 sep 2004 EMBnet Protein domain/family databases Contains biologically significant « pattern / profiles/ HMM
- 49. MCB, 6 sep 2004 EMBnet Protein domain/family db PROSITE Patterns / Profiles ProDom Aligned motifs (PSI-BLAST)
- 50. MCB, 6 sep 2004 EMBnet Prosite http://www.expasy.org/prosite/ Created in 1988 (SIB) Contains functional domains fully annotated,
- 51. PFAM (HMMs): an entry http://www.sanger.ac.uk/Software/Pfam/
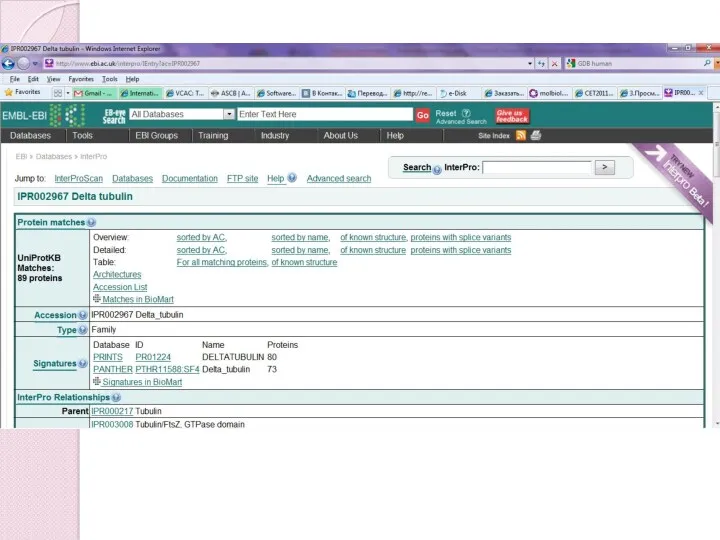
- 52. MCB, 6 sep 2004 EMBnet InterPro www.ebi.ac.uk/interpro Search simultaneously many domain databases. Single set of documents
- 54. MCB, 6 sep 2004 EMBnet Databases 6: proteomics Contain informations obtained by 2D-PAGE: images of master
- 55. MCB, 6 sep 2004 EMBnet Databases 7: 3D structure
- 56. Формати структурних даних правила та засоби зберігання даних щодо просторової структури макромолекул базова інформація – просторове
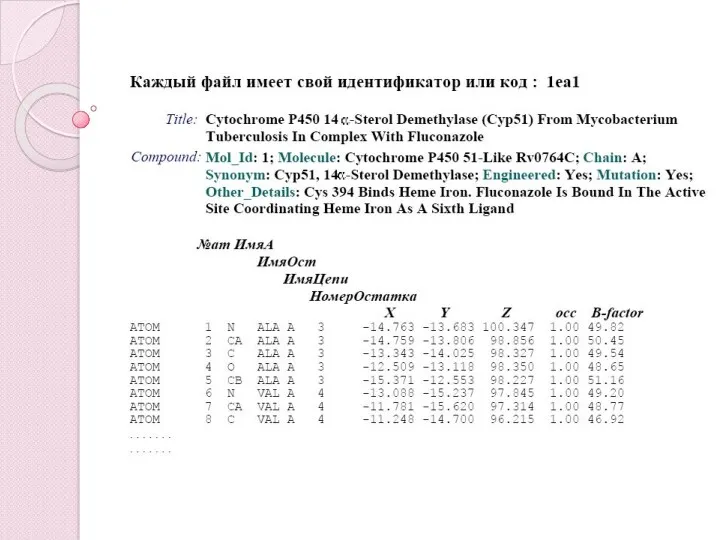
- 57. Формат PDB (Protein Data Bank) – один з основних форматів зберігання молекулярних даних забезпечує стандартне представлення
- 58. Остання версія керівництва по формату PDB – Atomic Coordinate Entry Format Description Version 3.1, July 19,
- 60. Типи записів в заголовному розділі HEADER – описує надходження банку через унікальний номер, класифікацію та дату
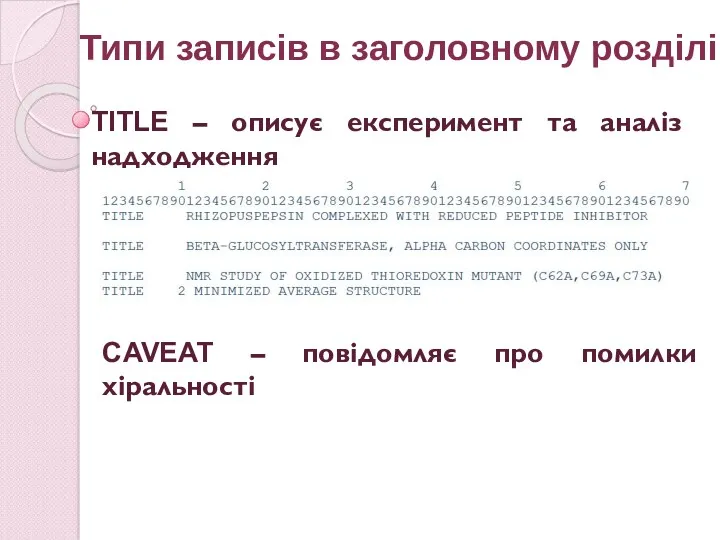
- 61. Типи записів в заголовному розділі TITLE – описує експеримент та аналіз надходження CAVEAT – повідомляє про
- 62. Типи записів в заголовному розділі COMPND – описує макромолекулярний компонент надходження
- 63. розшифровка деталей запису COMPND
- 64. Типи записів в заголовному розділі SOURCE – описує біологічне та/або хімічне джерело кожної біологічної молекули в
- 65. Типи записів в заголовному розділі EXPDTA – містить інформацію щодо експерименту
- 66. Типи записів в заголовному розділі KEYWDS – ключові слова, що стосуються надходження AUTHOR – імена людей,
- 67. Типи записів в заголовному розділі SPRSDE – які застарілі надходження замінені на поточне JRNL – основне
- 68. http://www.rcsb.org/ PDB – міжнародний банк даних білкових струкутр
- 69. http://ndbserver.rutgers.edu/ NDB - база даних просторових структур нуклеїнових кислот
- 70. http://www.ccdc.cam.ac.uk/ CSD (Cambridge Crystallographic Data Centre) – банк кристалографічних даних низькомолекулярних сполук
- 71. http://www.bmrb.wisc.edu/ BMRB - банк даних ЯМР-спектроскопії макромолекул
- 72. http://pqs.ebi.ac.uk/ PQS - база даних четвертинних структур білків
- 73. http://www.ebi.ac.uk/thornton-srv/databases/profunc/ Profunc – аналіз структури для пердбачення функцій
- 74. http://sfld.rbvi.ucsf.edu/ SFLDB – база даних “структура-функція”
- 75. http://scop.mrc-lmb.cam.ac.uk/scop/ SCOP – структурна класифікація білків
- 76. http://cathwww.biochem.ucl.ac.uk/latest/index.html CATH – структурна класифікація білків
- 77. http://scor.lbl.gov/ SCOR – структурна класифікація РНК
- 78. http://www.kinasenet.org/pkr/Welcome.do The Protein Kinase Resource – структури кіназ
- 79. http://mcl1.ncifcrf.gov/hivdb/index.html HIV Protease Database
- 80. MCB, 6 sep 2004 EMBnet Databases 8: metabolic Contain informations that describe enzymes, biochemical reactions and
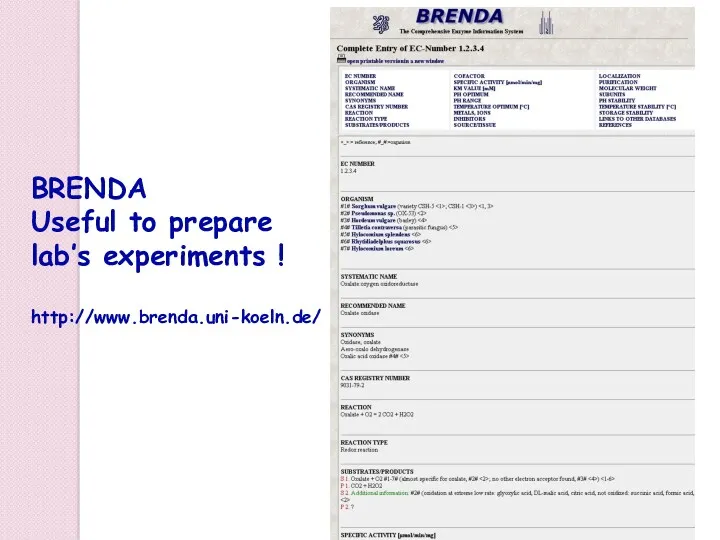
- 81. BRENDA Useful to prepare lab’s experiments ! http://www.brenda.uni-koeln.de/
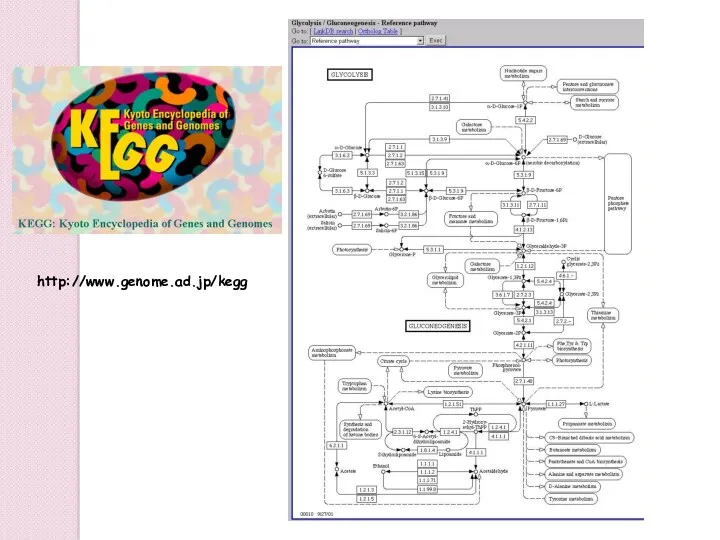
- 82. http://www.genome.ad.jp/kegg
- 83. MCB, 6 sep 2004 EMBnet Databases 9: bibliographic Bibliographic reference databases contain citations and abstract informations
- 84. MCB, 6 sep 2004 EMBnet Databases 10: others There are many databases that cannot be classified
- 85. MCB, 6 sep 2004 EMBnet Proliferation of databases What is the best db for sequence analysis
- 86. MCB, 6 sep 2004 EMBnet Some important practical remarks Databases: many errors (automated annotation) ! Not
- 87. Представление аминокислотной последовательности в Raw формате: MSEPQRLFFAIDLPAEIREQIIHWRATHFPPEAGRPVAADNLHLT LAFLGEVSAEKEKALSLLAGRIRQPGFTLTLDDAGQWLRSRVVWL GMRQPPRGLIQLANMLRSQAARSGCFQSNRPFHPHITLLRDASEA VTIPPPGFNWSYAVTEFTLYASSFARGRTRYTPLKRWALTQ
- 88. FASTA -формат FASTA - популярная программа предназначенная для выравнивания последователь- ностей и сканирования баз данных, созданная
- 89. ПРИМЕР: >gi|4885609|ref|NP_005408.1| proto-oncogene tyrosine-protein kinase SRC [Homo sapiens] MGSNKSKPKDASQRRRSLEPAENVHGAGGGAFPASQTPSKPASADGHRGPSAAFAPAAAEPKLFGGFNSS DTVTSPQRAGPLAGGVTTFVALYDYESRTETDLSFKKGERLQIVNNTEGDWWLAHSLSTGQTGYIPSNYV APSDSIQAEEWYFGKITRRESERLLLNAENPRGTFLVRESETTKGAYCLSVSDFDNAKGLNVKHYKIRKL DSGGFYITSRTQFNSLQQLVAYYSKHADGLCHRLTTVCPTSKPQTQGLAKDAWEIPRESLRLEVKLGQGC FGEVWMGTWNGTTRVAIKTLKPGTMSPEAFLQEAQVMKKLRHEKLVQLYAVVSEEPIYIVTEYMSKGSLL DFLKGETGKYLRLPQLVDMAAQIASGMAYVERMNYVHRDLRAANILVGENLVCKVADFGLARLIEDNEYT ARQGAKFPIKWTAPEAALYGRFTIKSDVWSFGILLTELTTKGRVPYPGMVNREVLDQVERGYRMPCPPEC PESLHDLMCQCWRKEPEERPTFEYLQAFLEDYFTSTEPQYQPGENL
- 90. Внимание!!! Некоторые программы могут быть чувствительны к формату записи в FASTA-формате: При написании однобуквенного кода всегда
- 91. Пример подачи последовательности в первичную базу данных Isolate P876, 16S rRNA gene sequence. Length: 1449 bp
- 92. Подача в GenBank при помощи инструмента BankIt
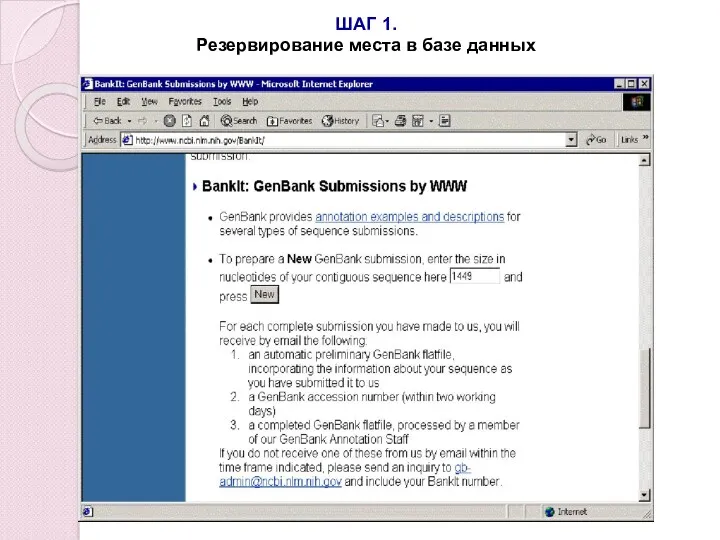
- 93. ШАГ 1. Резервирование места в базе данных
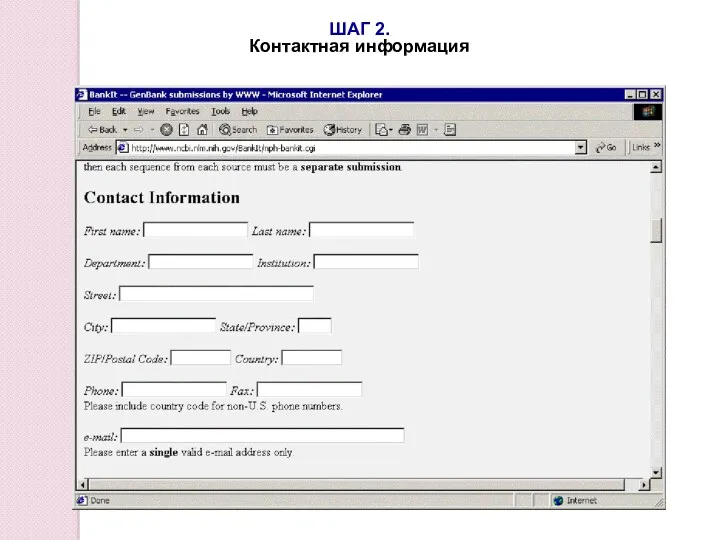
- 94. ШАГ 2. Контактная информация
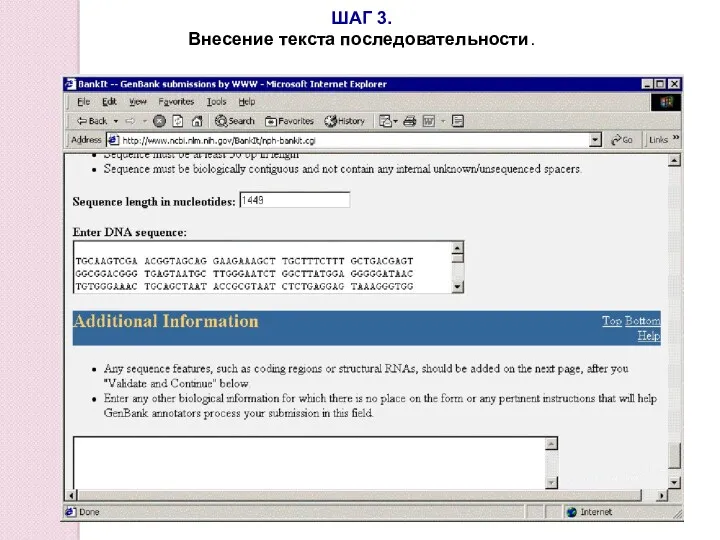
- 95. ШАГ 3. Внесение текста последовательности.
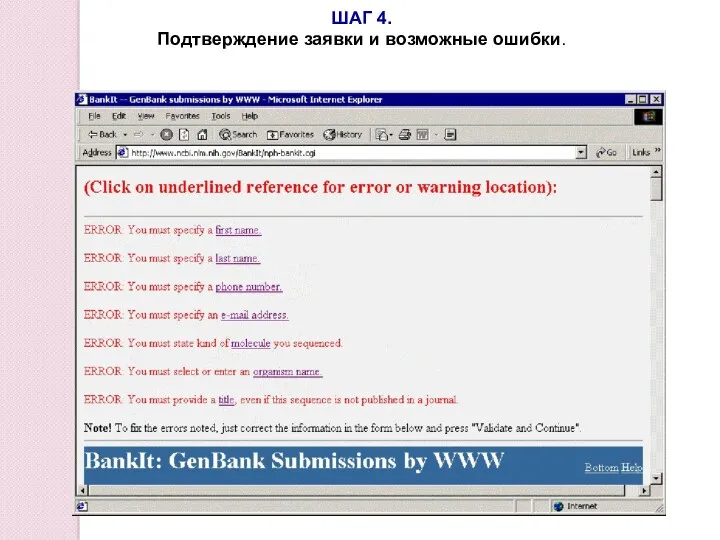
- 96. ШАГ 4. Подтверждение заявки и возможные ошибки.
- 98. Скачать презентацию





































































![ПРИМЕР: >gi|4885609|ref|NP_005408.1| proto-oncogene tyrosine-protein kinase SRC [Homo sapiens] MGSNKSKPKDASQRRRSLEPAENVHGAGGGAFPASQTPSKPASADGHRGPSAAFAPAAAEPKLFGGFNSS DTVTSPQRAGPLAGGVTTFVALYDYESRTETDLSFKKGERLQIVNNTEGDWWLAHSLSTGQTGYIPSNYV](/_ipx/f_webp&q_80&fit_contain&s_1440x1080/imagesDir/jpg/303651/slide-88.jpg)


 Бүйрек физиологиясы. Несеп түзілуінің механизмі
Бүйрек физиологиясы. Несеп түзілуінің механизмі Основные загрязнители почвы
Основные загрязнители почвы Функции нейрона. Передача возбуждения в синапсах
Функции нейрона. Передача возбуждения в синапсах Строение, свойства и функции углеводов и липидов в клетке
Строение, свойства и функции углеводов и липидов в клетке Пищеварительная система
Пищеварительная система Общее представление об организме
Общее представление об организме Презентация к уроку Эволюционное учение Ч. Дарвина
Презентация к уроку Эволюционное учение Ч. Дарвина Как звери готовятся к зиме
Как звери готовятся к зиме Приспособились ли потомки древних обитателей Земли – бактерии – к жизни на современной планете?
Приспособились ли потомки древних обитателей Земли – бактерии – к жизни на современной планете?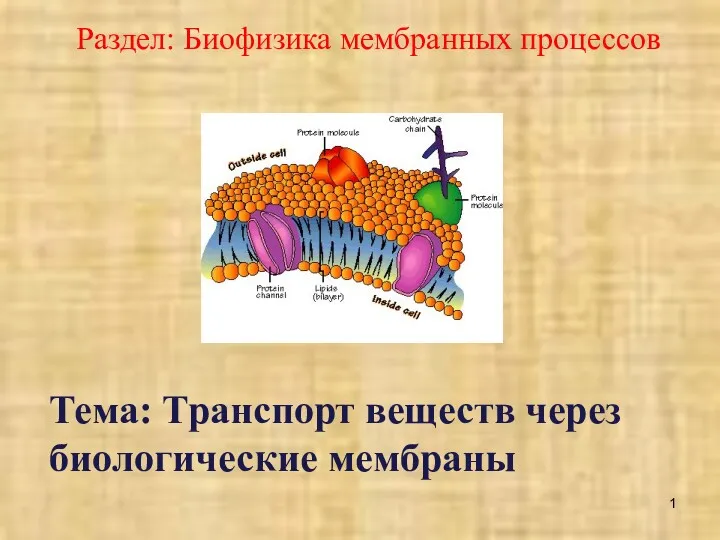 Транспорт веществ через биологические мембраны
Транспорт веществ через биологические мембраны Гидропонная система
Гидропонная система Анализирующее скрещивание. Дигибридное скрещивание. Третий закон Менделя
Анализирующее скрещивание. Дигибридное скрещивание. Третий закон Менделя Вплив електромагнітного випромінювання на рослини
Вплив електромагнітного випромінювання на рослини Презентация урока 6 класс ФГОС.
Презентация урока 6 класс ФГОС. Зимующие птицы. Подкармливание птиц зимой
Зимующие птицы. Подкармливание птиц зимой Дыхательная система человека
Дыхательная система человека Zoológico de Madrid
Zoológico de Madrid Игра Кто хочет стать отличником. Отборочный тур
Игра Кто хочет стать отличником. Отборочный тур Интересные факты о диких кошках
Интересные факты о диких кошках Тип хордовые
Тип хордовые Голонасінні. Загальна характеристика
Голонасінні. Загальна характеристика Клітинна радіобіологія. Теорія мішеней в клітинній радіобіології
Клітинна радіобіологія. Теорія мішеней в клітинній радіобіології Генетика, как наука. Взаимосвязь процессов наследственности и изменчивости
Генетика, как наука. Взаимосвязь процессов наследственности и изменчивости Информационный обзор по темам Ц. Бактерии, Ц. Грибы, Отделы растений, Вегетативные органы
Информационный обзор по темам Ц. Бактерии, Ц. Грибы, Отделы растений, Вегетативные органы Квантовые методы в медицине. Ядерный магнитный резонанс. Электронный парамагнитный резонанс. (Лекция 14)
Квантовые методы в медицине. Ядерный магнитный резонанс. Электронный парамагнитный резонанс. (Лекция 14) Органы чувств. Анализаторы
Органы чувств. Анализаторы Эмбриология (определение, виды)
Эмбриология (определение, виды) Семенные растения
Семенные растения