Содержание
- 2. 19 РЕПЛИКАЦИЯ ДНК а - полуконсервативная б - консервативная в - дисперсионная МОДЕЛИ РЕПЛИКАЦИИ ДНК РЕПЛИКАЦИЯ
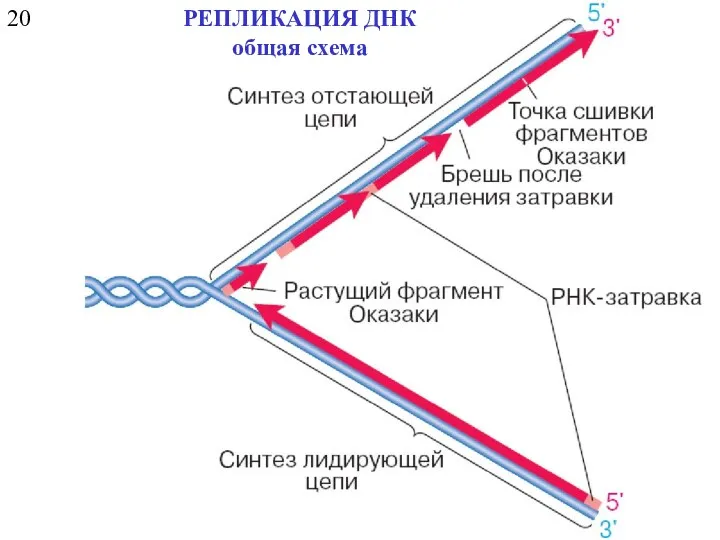
- 5. 20 РЕПЛИКАЦИЯ ДНК общая схема
- 6. 21 ДНК - ПОЛИМЕРАЗЫ 5’-3' - полимеразная активность DNArep.gif
- 7. 22 ДНК - ПОЛИМЕРАЗЫ 3' -5' - экзонуклеазная активность («редактирующая») 5'-3' - экзонуклеазная активность
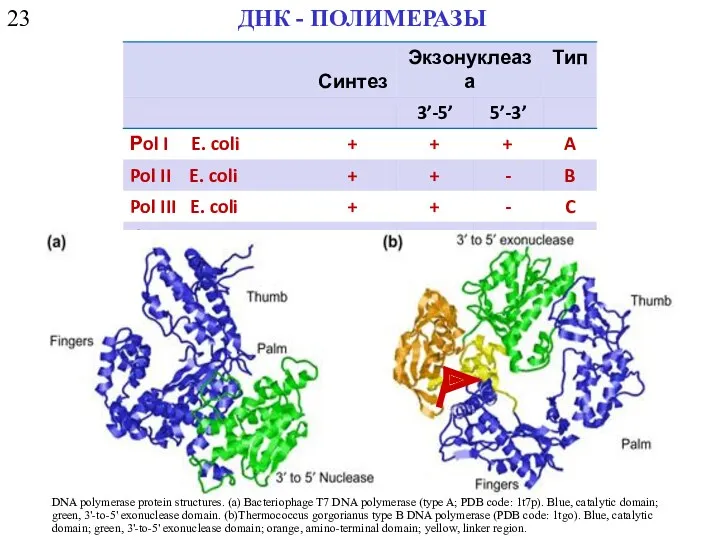
- 8. 23 ДНК - ПОЛИМЕРАЗЫ Синтез Экзонуклеаза 5’-3’ 3’-5’ 5’-3’ тип Рol I E. coli + +
- 9. 23 ДНК - ПОЛИМЕРАЗЫ
- 10. 24 ДНК - ПОЛИМЕРАЗЫ Структура комплекса Pol I ДНК- полимеразы с ДНК
- 11. 25 ДНК - ЛИГАЗЫ NAD+ никотинамидадениндинуклеотид Механизм действия ДНК-лигазы
- 12. 26 РАСКРУЧИВАНИЕ ДНК СПИРАЛИ ПРИ РЕПЛИКАЦИИ геликаза PcrA - комплекс с субстратом ДНК-геликаза Белки, связывающиеся с
- 13. 27 ДНК-ТОПОИЗОМЕРАЗЫ Топоизомераза I Топоизомераза II Размыкание кольцевых дуплексов Гираза Топоизомеразами типа I, уменьшают число сверхвитков
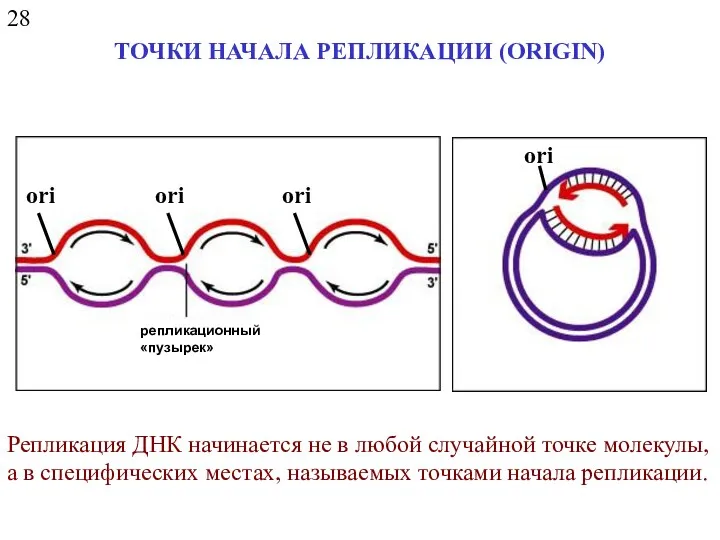
- 15. 28 ТОЧКИ НАЧАЛА РЕПЛИКАЦИИ (ORIGIN) репликационный «пузырек» ori ori Репликация ДНК начинается не в любой случайной
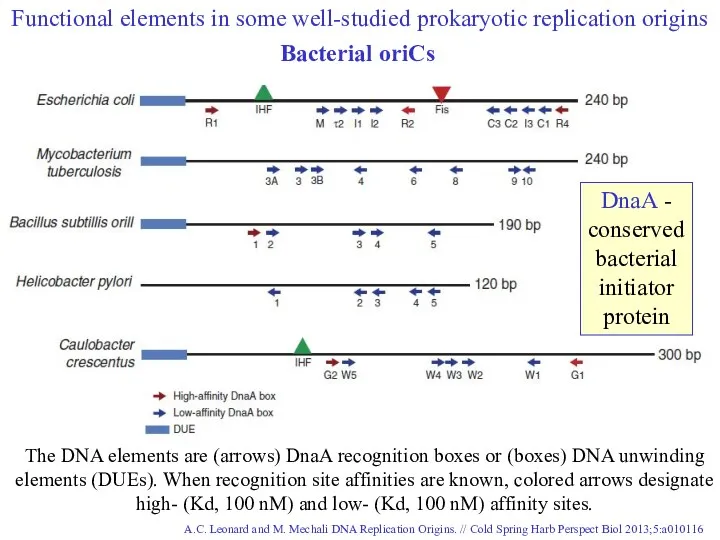
- 16. A.C. Leonard and M. Mechali DNA Replication Origins. // Cold Spring Harb Perspect Biol 2013;5:a010116 Revised
- 17. A.C. Leonard and M. Mechali DNA Replication Origins. // Cold Spring Harb Perspect Biol 2013;5:a010116 The
- 18. 29 ИНИЦИАЦИЯ ОБРАЗОВАНИЯ НОВЫХ ЦЕПЕЙ ДНК Инициация репликации одноцепочечной фаговой ДНК Инициация репликации плазмид типа Col
- 19. 30 Инициация синтеза отстающей цепи МОДЕЛЬ ПОЛУКОНСЕРВАТИВНОЙ ПРЕРЫВИСТОЙ РЕПЛИКАЦИИ ДНК В РЕПЛИКАТИВНОЙ ВИЛКЕ
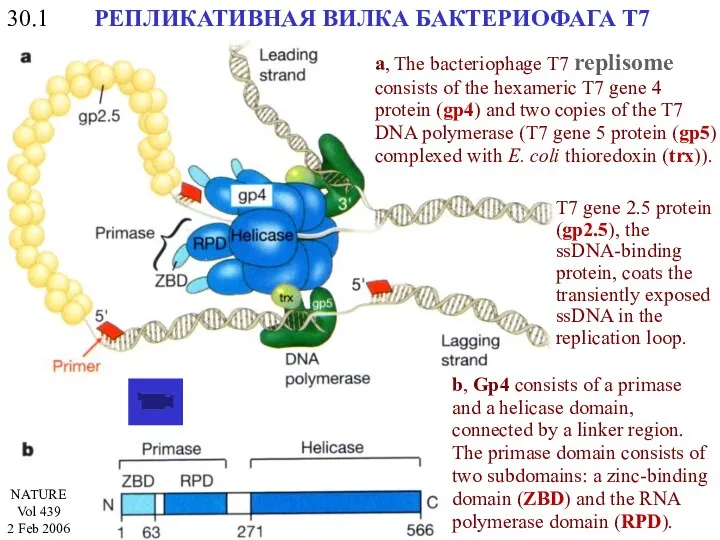
- 20. 30.1 РЕПЛИКАТИВНАЯ ВИЛКА БАКТЕРИОФАГА Т7 a, The bacteriophage T7 replisome consists of the hexameric T7 gene
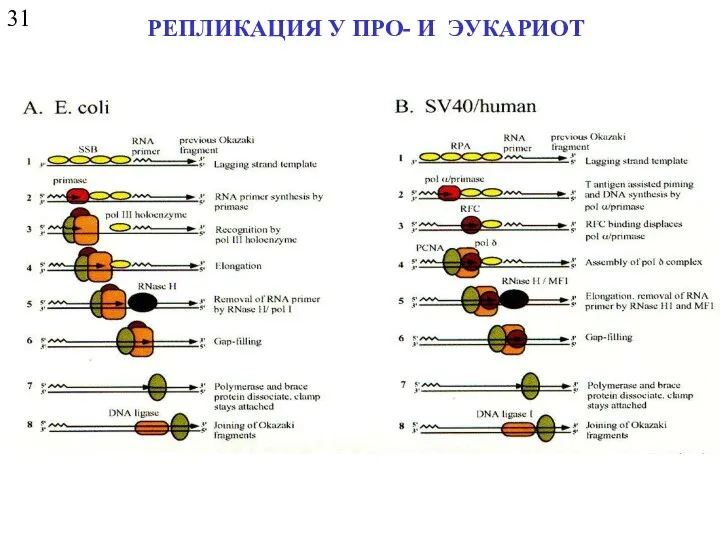
- 21. 31 РЕПЛИКАЦИЯ У ПРО- И ЭУКАРИОТ
- 22. 32 РЕПЛИКАЦИЯ ПО ТИПУ КАТЯЩЕГОСЯ КОЛЬЦА ПООЧЕРЕДНАЯ РЕПЛИКАЦИЯ ЦЕПЕЙ
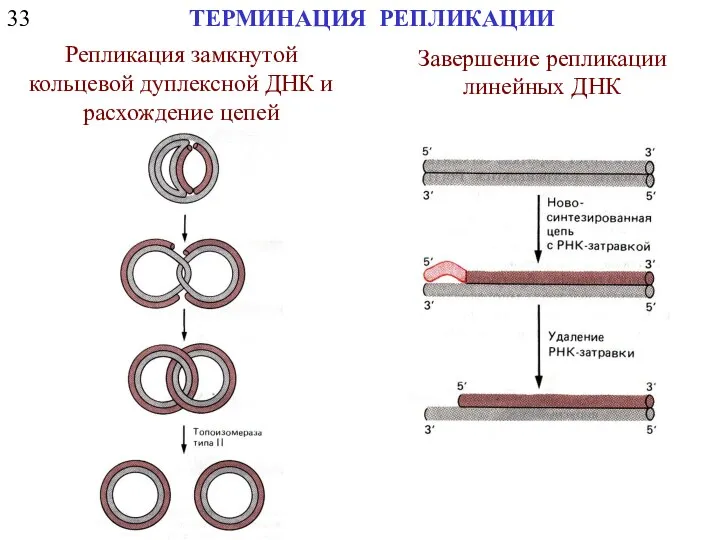
- 23. 33 ТЕРМИНАЦИЯ РЕПЛИКАЦИИ Репликация замкнутой кольцевой дуплексной ДНК и расхождение цепей Завершение репликации линейных ДНК
- 24. 34 НЕДОРЕПЛИКАЦИЯ КОНЦЕВОЙ ЧАСТИ МОЛЕКУЛЫ ДНК Барьер (лимит) Хейфлика – ограничение на число клеточных делений
- 25. 35 ОРГАНИЗАЦИЯ ТЕЛОМЕРНОГО КОНЦА ХРОМОСОМЫ Вид Теломерные повторы в хромосомах некоторых видов
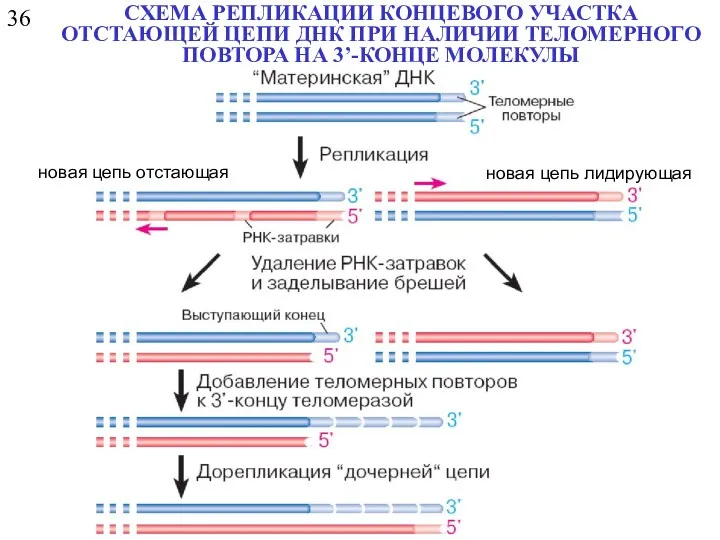
- 26. 36 СХЕМА РЕПЛИКАЦИИ КОНЦЕВОГО УЧАСТКА ОТСТАЮЩЕЙ ЦЕПИ ДНК ПРИ НАЛИЧИИ ТЕЛОМЕРНОГО ПОВТОРА НА 3’-КОНЦЕ МОЛЕКУЛЫ новая
- 27. 37 УДЛИНЕНИЕ ТЕЛОМЕРНОГО ПОВТОРА ПРИ ПОМОЩИ ФЕРМЕНТА ТЕЛОМЕРАЗЫ ТЕЛОМЕРАЗА (ДНК-нуклеотидилэкзотрансфераза, теломерная терминальная трансфераза) – ДНК полимераза,
- 28. 37 УДЛИНЕНИЕ ТЕЛОМЕРНОГО ПОВТОРА ПРИ ПОМОЩИ ФЕРМЕНТА ТЕЛОМЕРАЗЫ
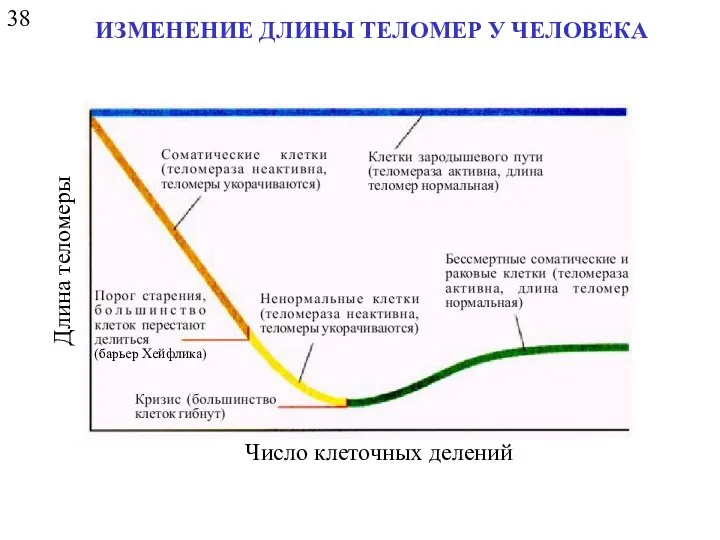
- 30. 38 ИЗМЕНЕНИЕ ДЛИНЫ ТЕЛОМЕР У ЧЕЛОВЕКА Число клеточных делений Длина теломеры (барьер Хейфлика)
- 31. 38 ИЗМЕНЕНИЕ ДЛИНЫ ТЕЛОМЕР У ЧЕЛОВЕКА
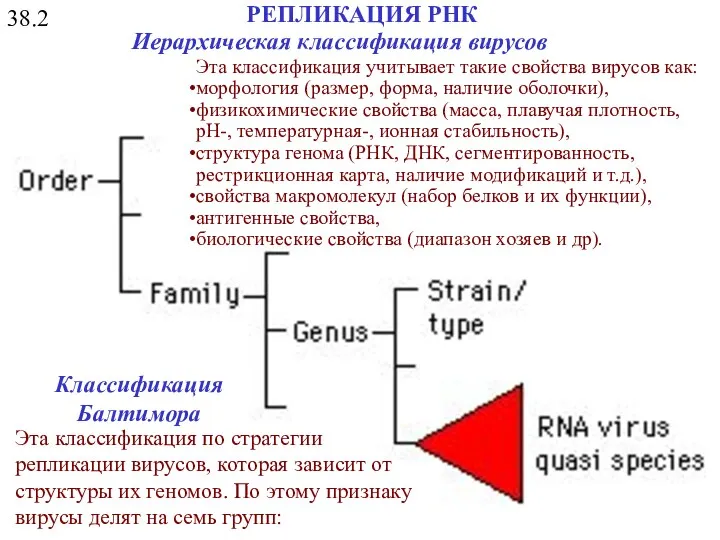
- 32. 38.2 РЕПЛИКАЦИЯ РНК Иерархическая классификация вирусов Эта классификация по стратегии репликации вирусов, которая зависит от структуры
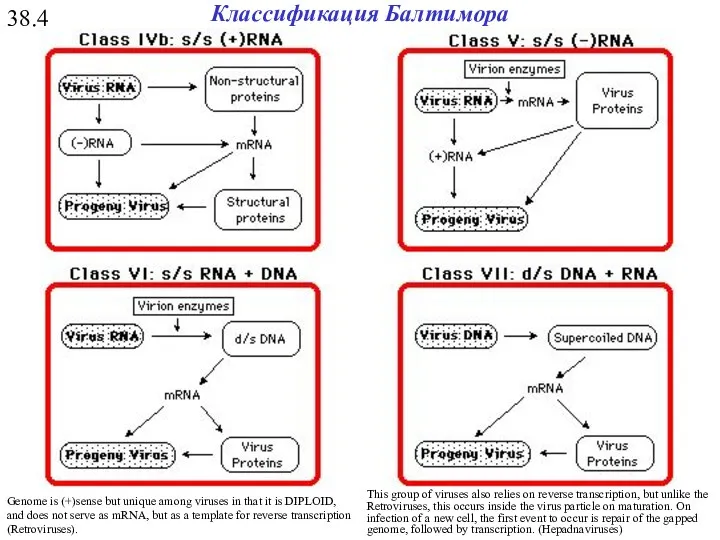
- 33. 38.3 Классификация Балтимора
- 34. 38.4 Genome is (+)sense but unique among viruses in that it is DIPLOID, and does not
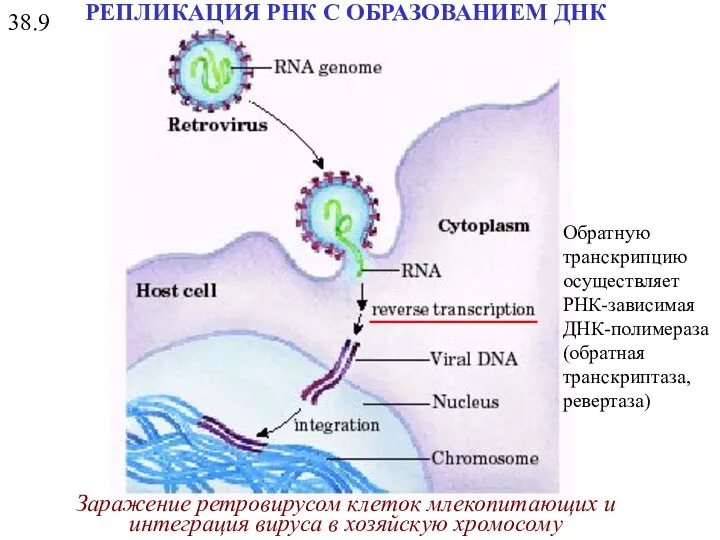
- 35. Заражение ретровирусом клеток млекопитающих и интеграция вируса в хозяйскую хромосому 38.9 РЕПЛИКАЦИЯ РНК С ОБРАЗОВАНИЕМ ДНК
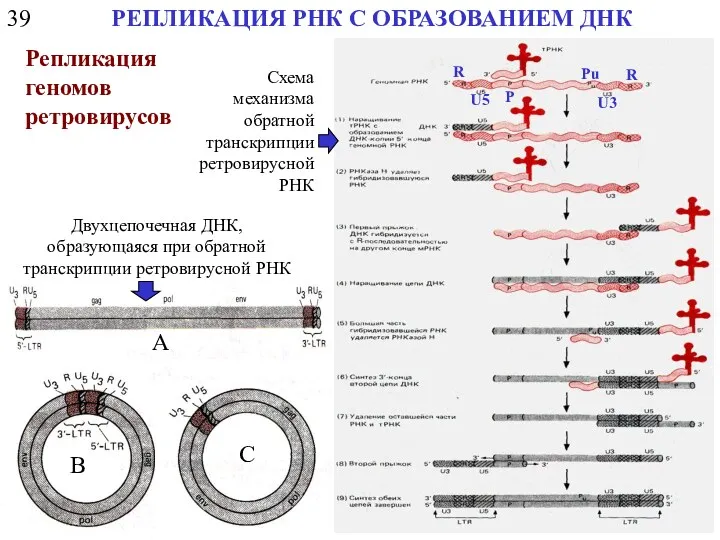
- 36. 39 РЕПЛИКАЦИЯ РНК С ОБРАЗОВАНИЕМ ДНК Репликация геномов ретровирусов Двухцепочечная ДНК, образующаяся при обратной транскрипции ретровирусной
- 40. Скачать презентацию
























 Physiological bases of hemo dynamic
Physiological bases of hemo dynamic Развитие органического мира
Развитие органического мира Презентация к уроку Класс птицы. Особенности строения связанные с полетом
Презентация к уроку Класс птицы. Особенности строения связанные с полетом Открытый урок по биологии 8 класс
Открытый урок по биологии 8 класс Бұршақтар, қалақайлар, шамшаттар тұқымдастарына сипаттама
Бұршақтар, қалақайлар, шамшаттар тұқымдастарына сипаттама Гигиена пищеварения. Предупреждение желудочно-кишечных заболеваний
Гигиена пищеварения. Предупреждение желудочно-кишечных заболеваний Приспособленность живых организмов к среде обитания
Приспособленность живых организмов к среде обитания Люпин белый. Особенности биологии и технологии возделывания
Люпин белый. Особенности биологии и технологии возделывания Донецкий ботанический сад— один из крупнейших ботанических садов Европы
Донецкий ботанический сад— один из крупнейших ботанических садов Европы Грунт для биологической очистки, для аквариумных растений, для естественной красоты
Грунт для биологической очистки, для аквариумных растений, для естественной красоты Живая природа осенью
Живая природа осенью Класс птицы
Класс птицы Мой домашний любимец
Мой домашний любимец Растительный и животный мир Раменского района
Растительный и животный мир Раменского района Анатомия и физиология женской половой системы
Анатомия и физиология женской половой системы Нормофлоры и пробиотики. Препараты на основе живых культур микроорганизмов. Культуры клеток и тканей растений
Нормофлоры и пробиотики. Препараты на основе живых культур микроорганизмов. Культуры клеток и тканей растений Цветок. Строение и функции цветка и его частей
Цветок. Строение и функции цветка и его частей Органические и неорганические вещества, входящие в состав клетки. Нуклеиновые кислоты и их роль в клетке
Органические и неорганические вещества, входящие в состав клетки. Нуклеиновые кислоты и их роль в клетке План-конспект урока по биологии
План-конспект урока по биологии Кость как орган
Кость как орган Изучение жизни муравьев-жнецов в условиях домашнего формикария
Изучение жизни муравьев-жнецов в условиях домашнего формикария Презентация к уроку биологии 5 класс Зачем живые организмы запасают питательные вещества?
Презентация к уроку биологии 5 класс Зачем живые организмы запасают питательные вещества? Закрепление материала по биологии для 5-ого класса
Закрепление материала по биологии для 5-ого класса Роль воды в жизни растений. Проращивание семян
Роль воды в жизни растений. Проращивание семян Фотосинтез
Фотосинтез Разнообразие земноводных
Разнообразие земноводных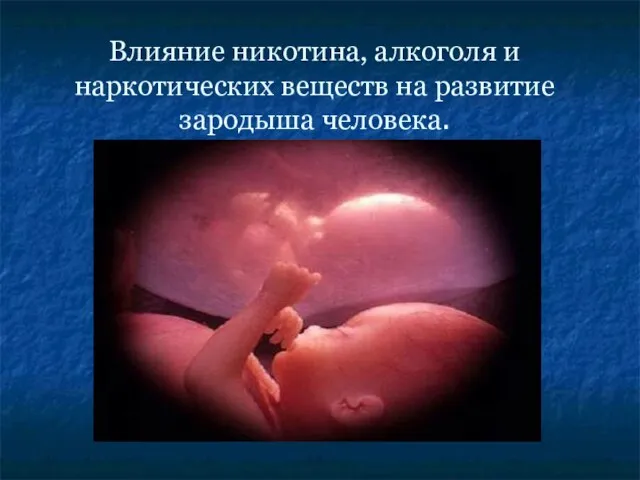 Влияние никотина, алкоголя и наркотических веществ на развитие зародыша человека
Влияние никотина, алкоголя и наркотических веществ на развитие зародыша человека Остеология, часть 1. Лекция 2
Остеология, часть 1. Лекция 2