Содержание
- 2. Краткий экскурс в историю становления биоинформатики Для биолога Для биоинформатика Ряд Нобелевских премий 1950-1970
- 3. Альфа-субъединица АТФ-синтетазы Лайнус Полинг Нобелевская премия по химии 1954 Вторичная структура – альфа-спираль и бета-лист Белки
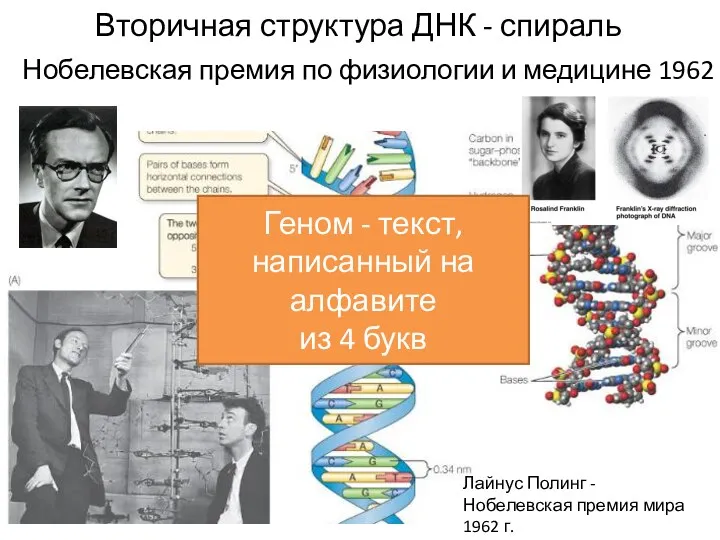
- 4. Нобелевская премия по физиологии и медицине 1962 Вторичная структура ДНК - спираль Лайнус Полинг - Нобелевская
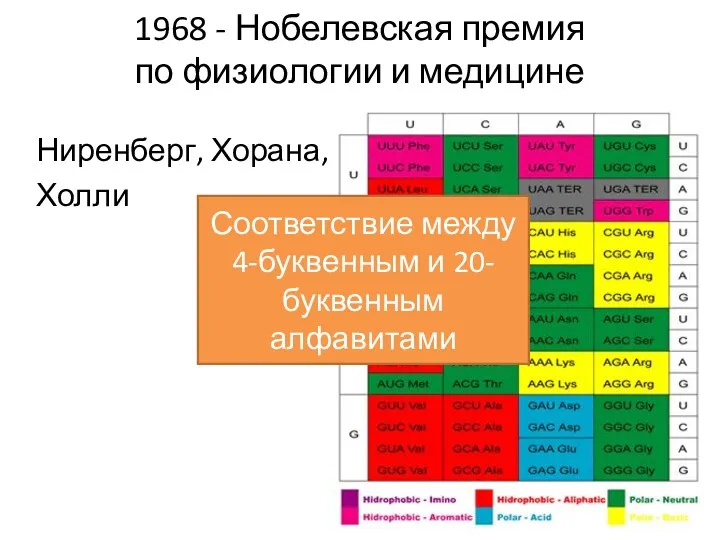
- 5. 1968 - Нобелевская премия по физиологии и медицине Ниренберг, Хорана, Холли Соответствие между 4-буквенным и 20-буквенным
- 6. Рождение технологий
- 7. 1977 год Нобелевская премия по химии 1980 г. Метод секвенирования Сэнгера The Wellcome Trust Sanger Institute,
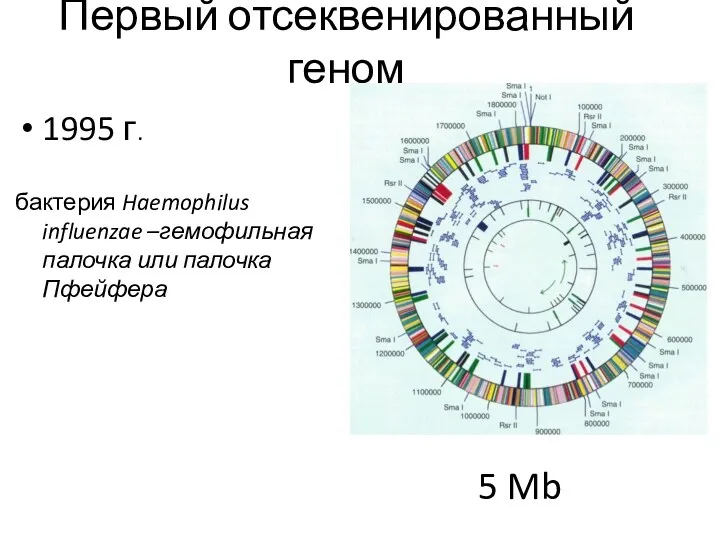
- 8. Первый отсеквенированный геном 1995 г. бактерия Haemophilus influenzae –гемофильная палочка или палочка Пфейфера 5 Mb
- 9. Human Genome Project Started in 1990 Finished 2001/2003 Sanger Sequencing 3 Gb
- 11. Next Generation Sequencing, or NextGenSeq, or NGS
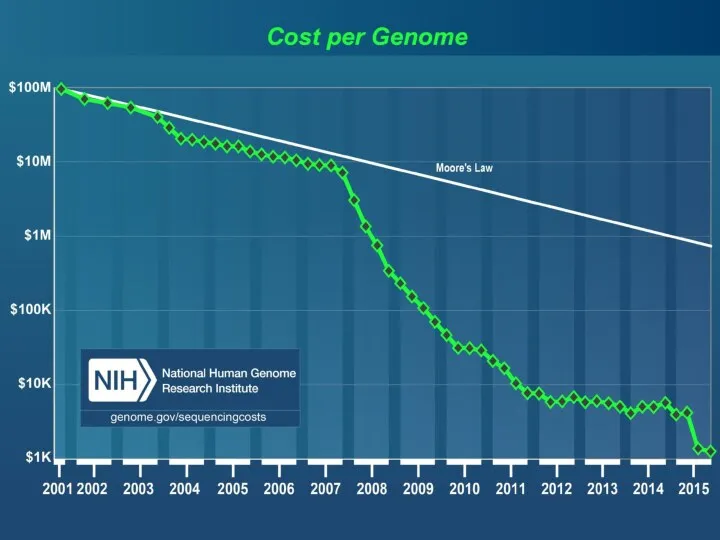
- 14. NextGen Sequencing Revolution Human genome – Volumes, double-sided, 4pt, ~ 43,000 chars per page To print
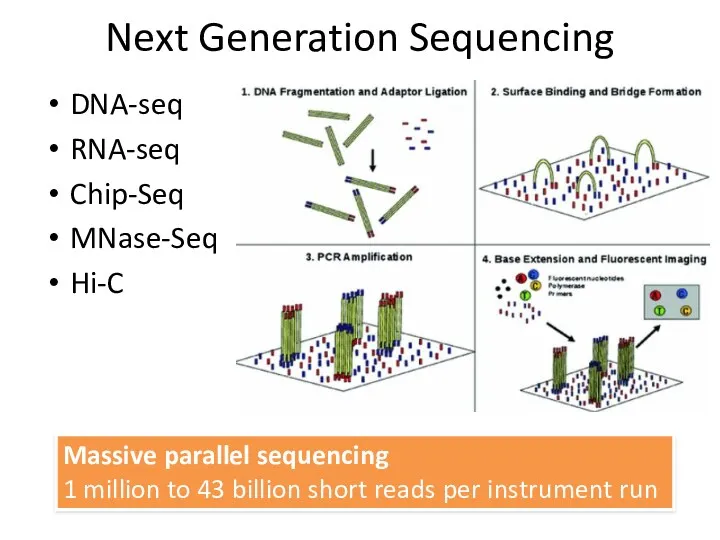
- 15. Next Generation Sequencing DNA-seq RNA-seq Chip-Seq MNase-Seq Hi-C Massive parallel sequencing 1 million to 43 billion
- 16. Data Accumulation
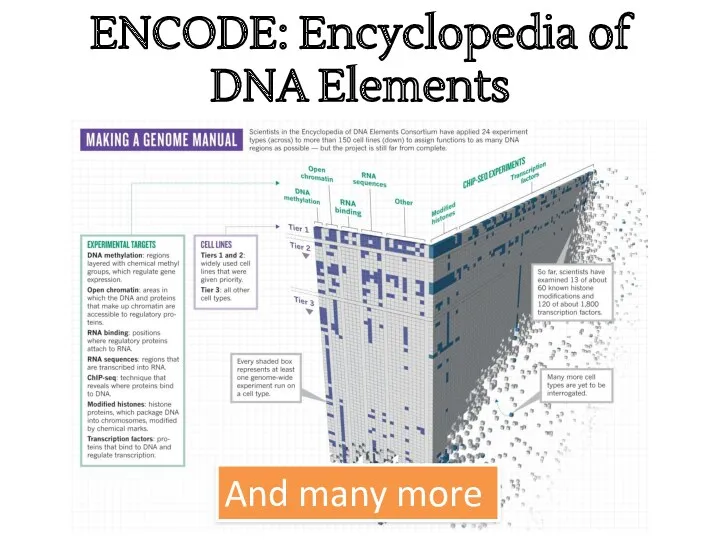
- 17. ENCODE: Encyclopedia of DNA Elements And many more
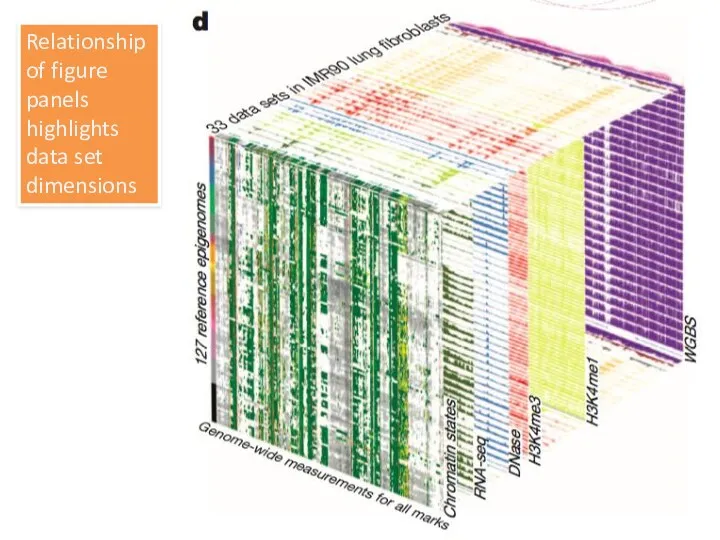
- 18. Relationship of figure panels highlights data set dimensions
- 19. Machine-Learning for cracking the codes
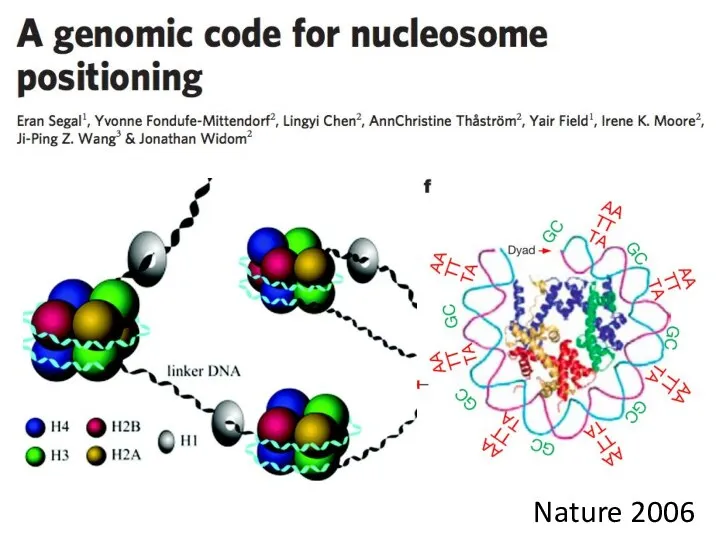
- 20. Nature 2006
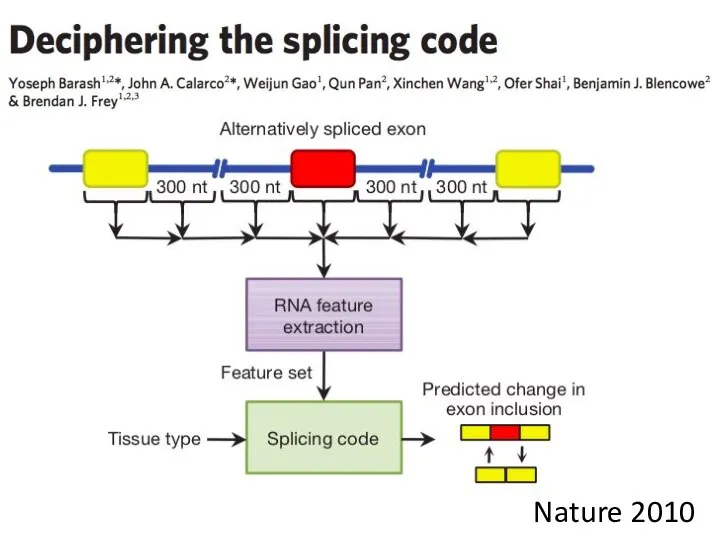
- 21. Nature 2010
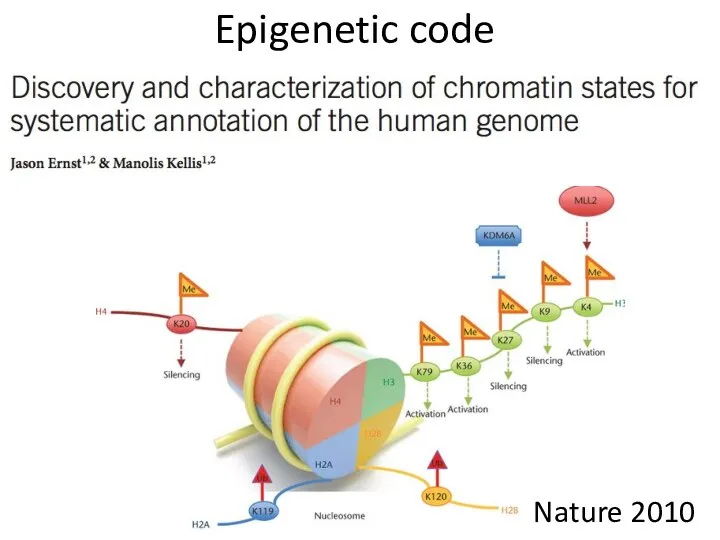
- 22. Epigenetic code Nature 2010
- 23. 2005-2015 много статей по распознаванию функциональных элементов генома методами “классического” машинного обучения
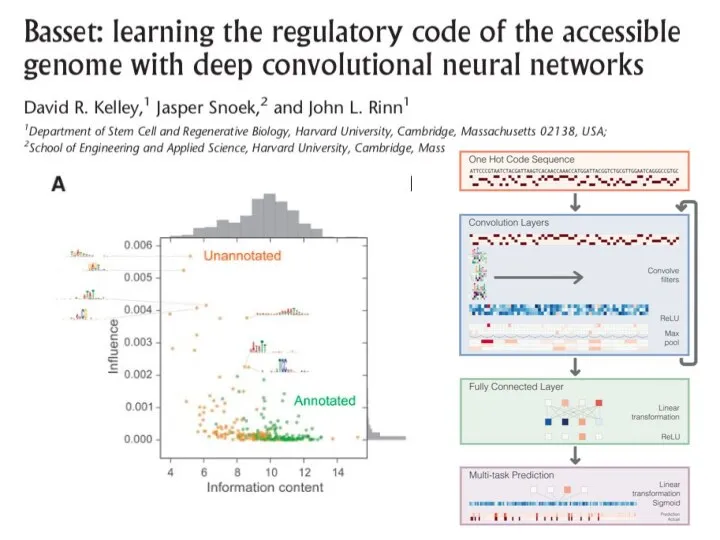
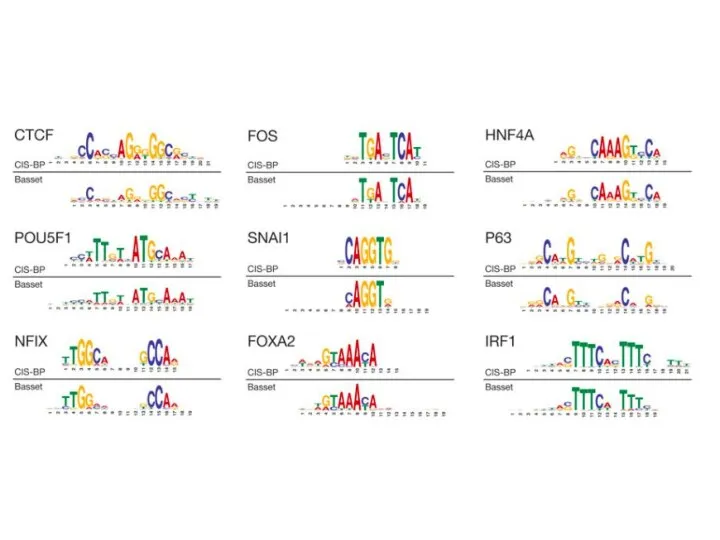
- 24. Deep Learning
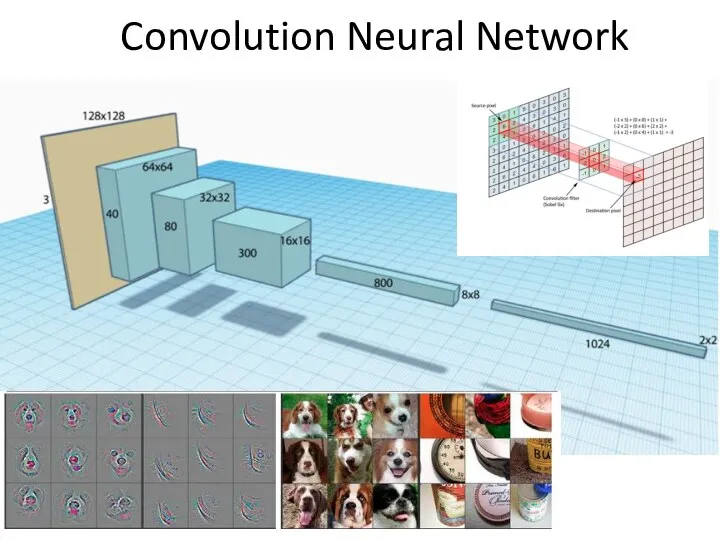
- 25. Convolution Neural Network
- 26. DNA as picture? Nature Methods volume 12, pages 931–934 (2015) Nature 2015
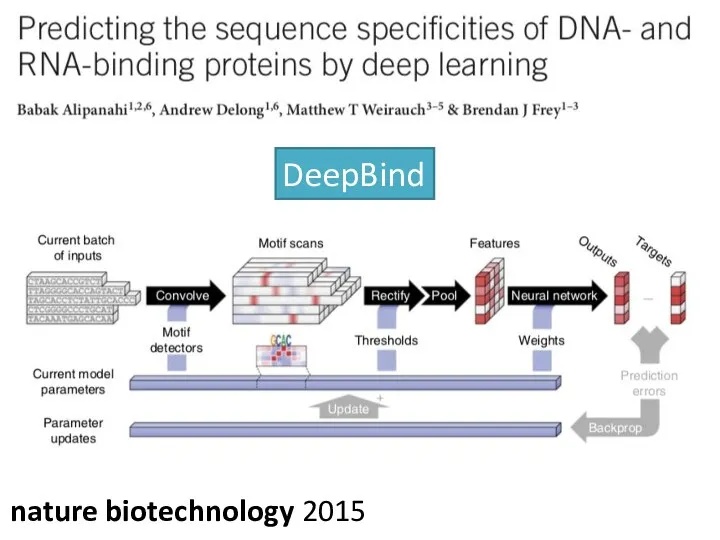
- 27. nature biotechnology 2015 DeepBind
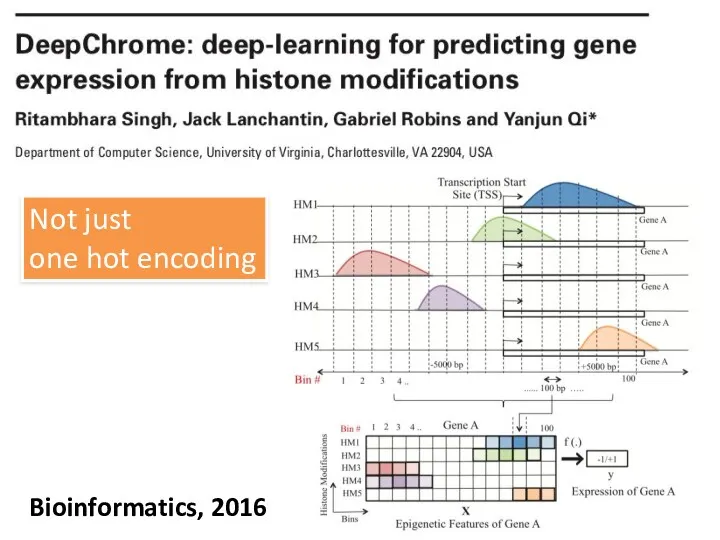
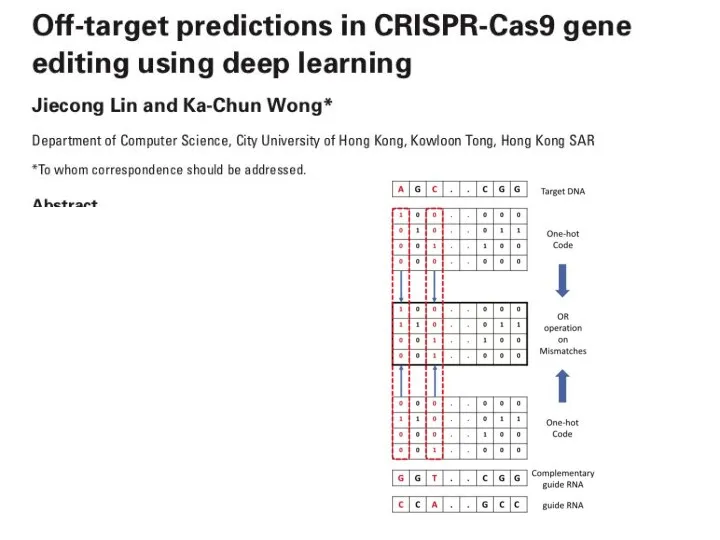
- 30. Bioinformatics, 2016 Not just one hot encoding
- 31. Bioinformatics, 2018, Plus structure information
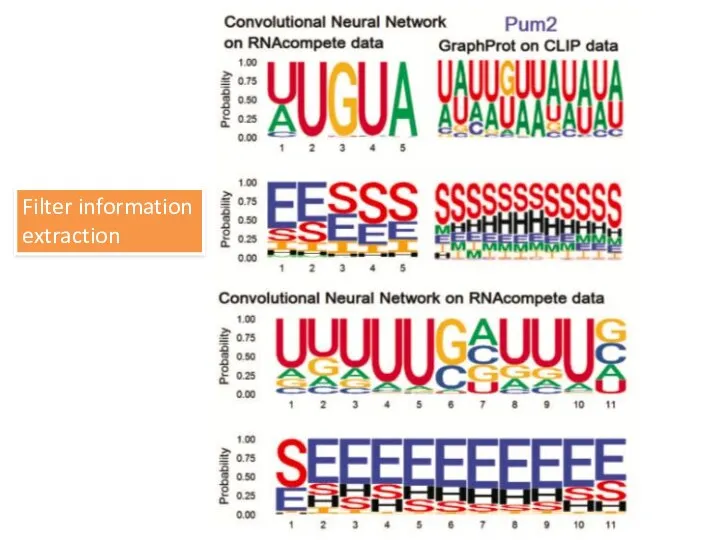
- 32. Filter information extraction
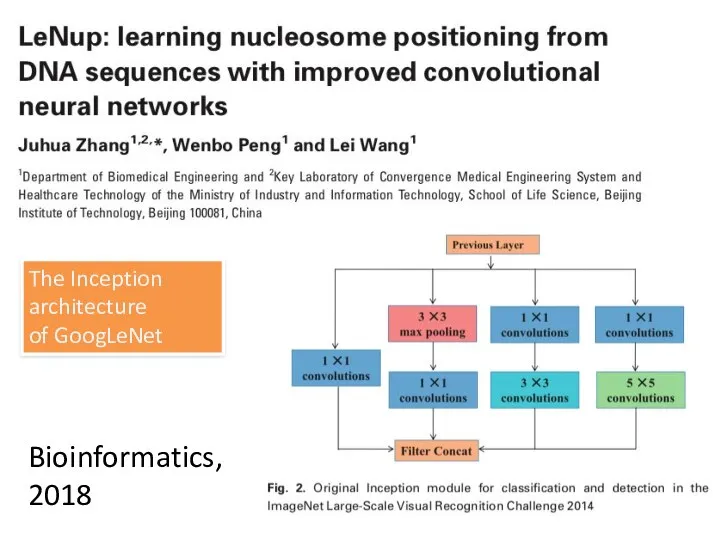
- 33. Bioinformatics, 2018 The Inception architecture of GoogLeNet
- 36. 2019 - 2024 Take the best solution from AI several years later it was introduced in
- 37. DNA Computing
- 38. Basic Idea: Perform molecular biology experiment to find solution to math problem.
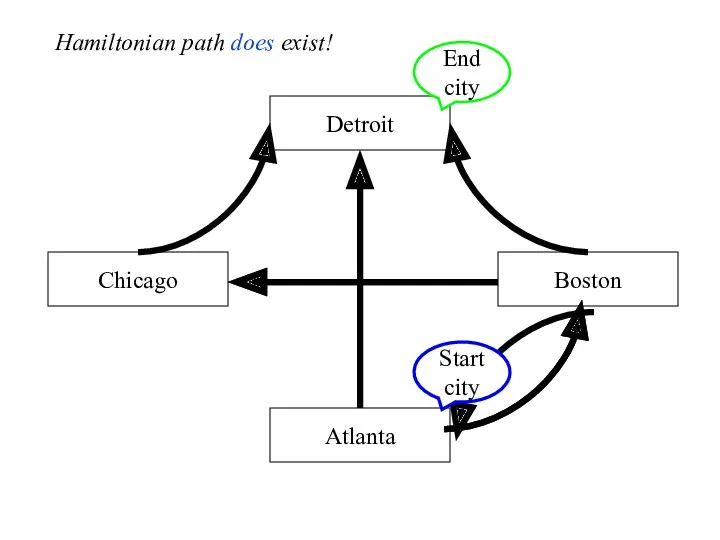
- 39. HAMILTONIAN PATH PROBLEM Given a network of nodes and directed connections between them, is there a
- 40. Detroit Boston Chicago Atlanta Start city End city Hamiltonian path does exist!
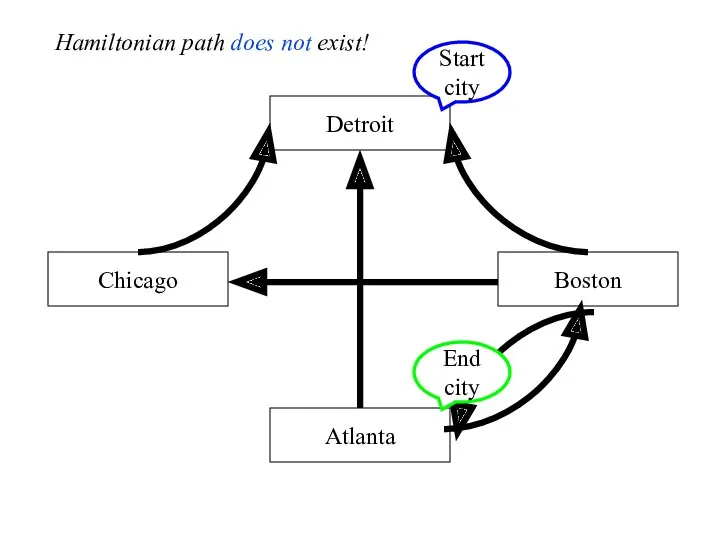
- 41. Detroit Boston Chicago Atlanta End city Start city Hamiltonian path does not exist!
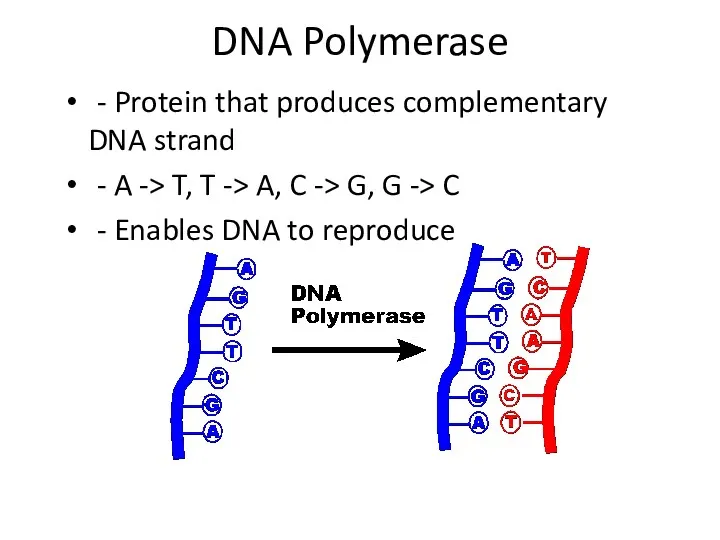
- 42. DNA Polymerase - Protein that produces complementary DNA strand - A -> T, T -> A,
- 43. Polymerase in Action The “bio” nano-machine: hops onto DNA strand slides along reads each base writes
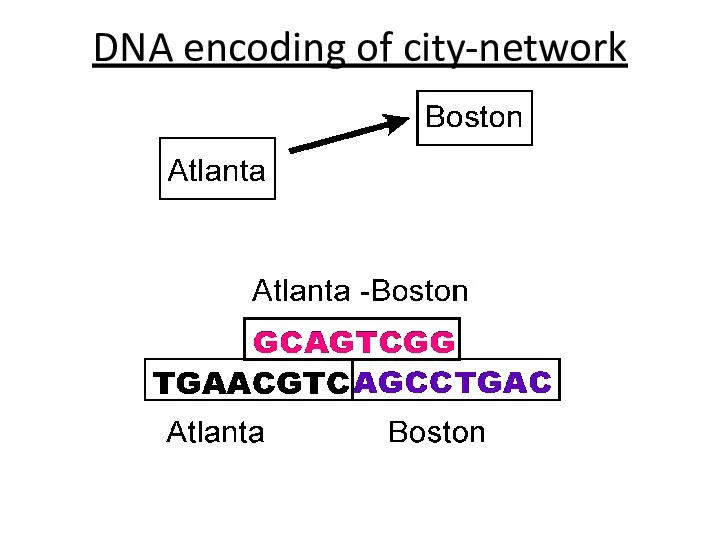
- 45. DNA encoding of city-network
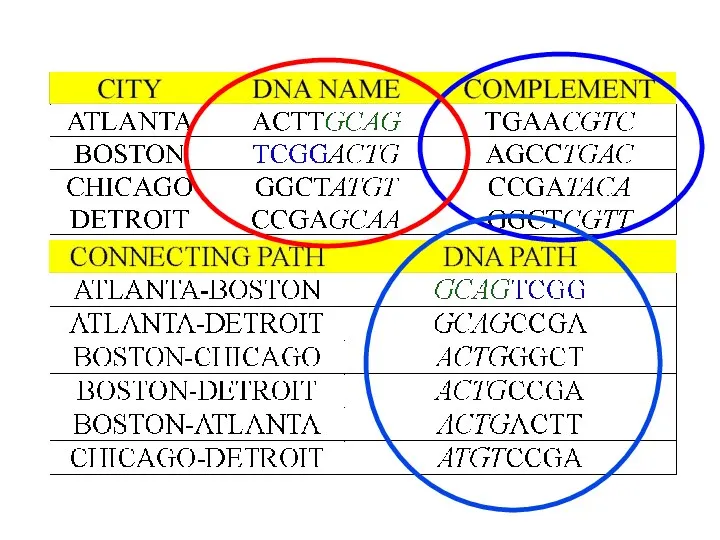
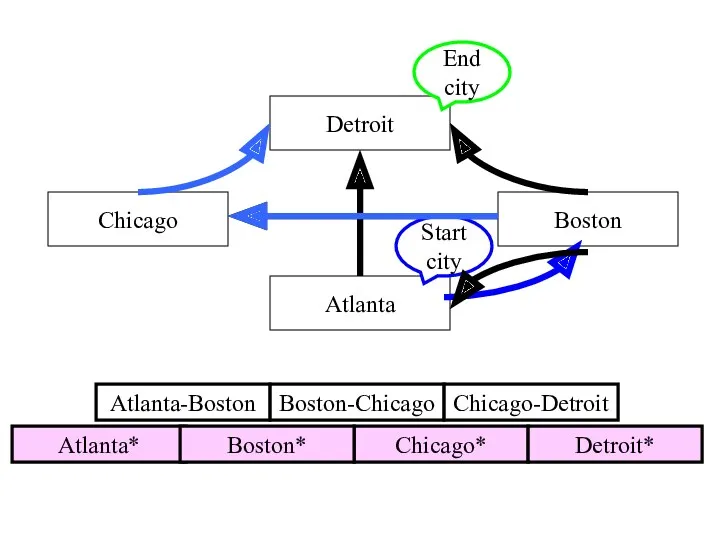
- 46. Detroit Boston Chicago Atlanta Start city End city Atlanta-Boston Boston-Chicago Chicago* Chicago-Detroit Detroit* Atlanta* Boston*
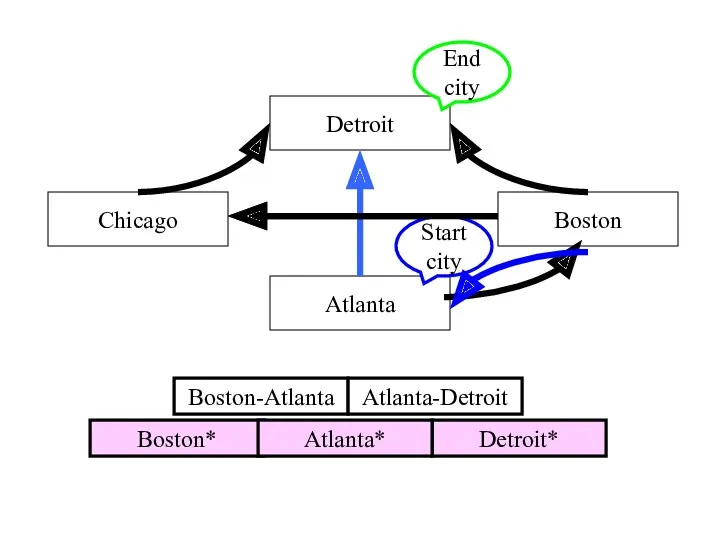
- 47. Detroit Boston Chicago Atlanta Start city End city Boston-Atlanta Atlanta-Detroit Detroit* Boston* Atlanta*
- 48. Ingredients and tools needed: - DNA strands that encode city names and connections between them -
- 49. Deepthi Bollu How Dense is the Information Storage? Nucleotides are spaced at 0.35 nm along DNA
- 50. Deepthi Bollu How enormous is the parallelism? A test tube of DNA can contain trillions of
- 51. Deepthi Bollu How extraordinary is the energy efficiency? Adleman figured his computer was running 2 x
- 52. Сomputer that ‘grows as it computes’ 2017
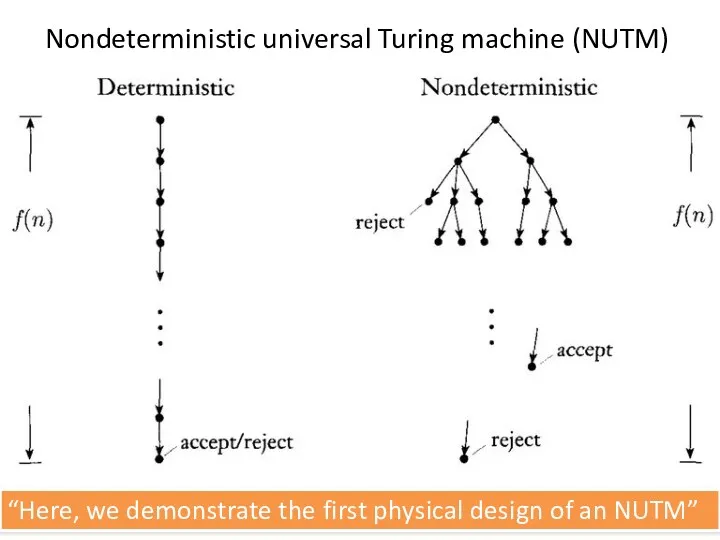
- 53. “Here, we demonstrate the first physical design of an NUTM” Nondeterministic universal Turing machine (NUTM)
- 54. Nondeterministic universal Turing machine (NUTM) Electronic computers need to choose which path to follow first. DNA
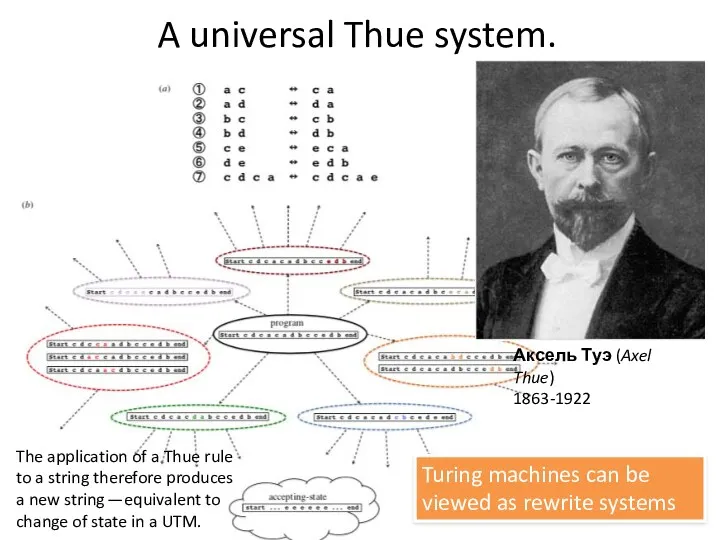
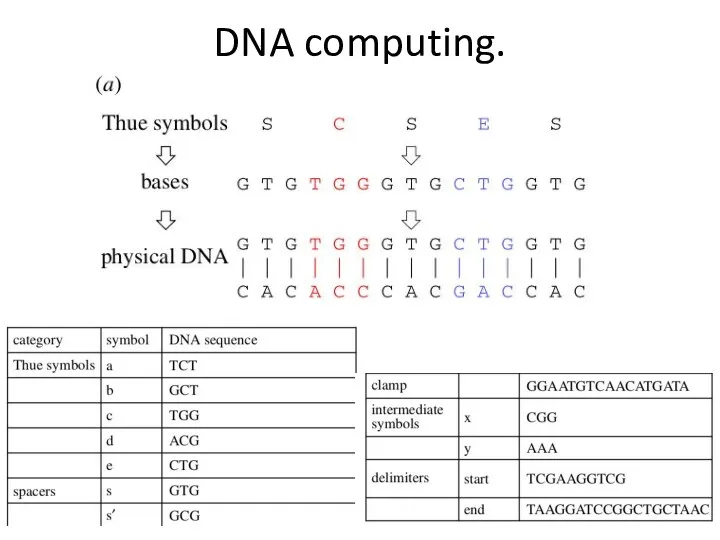
- 55. A universal Thue system. Аксель Туэ (Axel Thue) 1863-1922 Turing machines can be viewed as rewrite
- 56. DNA computing.
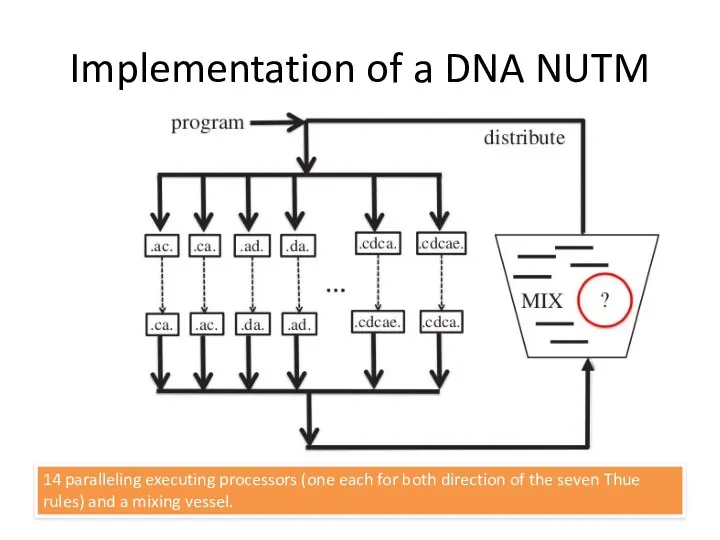
- 57. Implementation of a DNA NUTM 14 paralleling executing processors (one each for both direction of the
- 58. “As DNA molecules are very small a desktop computer could potentially utilize more processors than all
- 60. Human disease-gene networks
- 61. Рэй Курцвейл
- 62. 2020 – Персональные компьютеры достигнут вычислительной мощности, сравнимой с человеческим мозгом. … 2028 – Солнечная энергия
- 63. 2036 – Используя подход к биологии, как к программированию, человечеству впервые удастся запрограммировать клетки для лечения
- 64. Нобелевская премия по физиологии и медицине 203X год - за открытие алгоритмов работы генома 203X год
- 66. Скачать презентацию
































 Анатомия человека
Анатомия человека Строение волос
Строение волос Тип моллюски
Тип моллюски Надкласс II. Рыбы
Надкласс II. Рыбы Развитие органического мира
Развитие органического мира Омыртқалы жануарлардың қозғалуы
Омыртқалы жануарлардың қозғалуы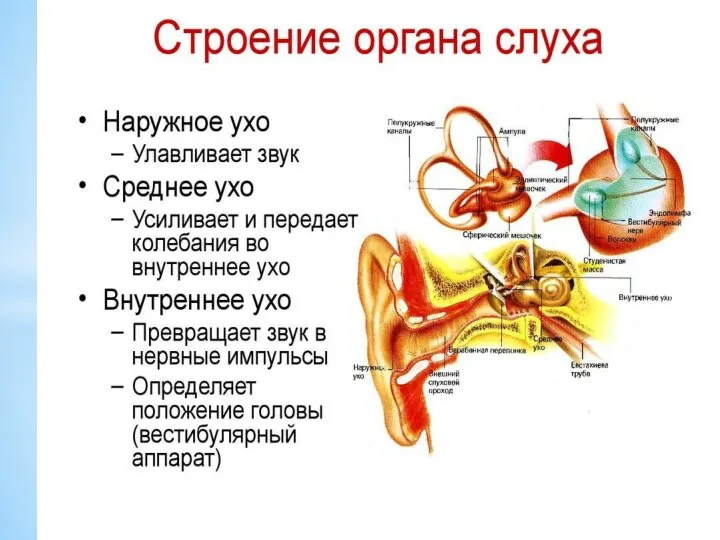 Строение органа слуха
Строение органа слуха Спинной мозг
Спинной мозг Сочные плоды
Сочные плоды Циклы развития растений (занятие по подготовке к ЕГЭ)
Циклы развития растений (занятие по подготовке к ЕГЭ) Половые гормоны
Половые гормоны Общий обзор головного мозга; его части как производные мозговых пузырей
Общий обзор головного мозга; его части как производные мозговых пузырей Разнообразие пород гусей
Разнообразие пород гусей Хордові. Підтип покривники, безчерепні
Хордові. Підтип покривники, безчерепні Вомбат – житель Австралии
Вомбат – житель Австралии Палеозойская эра
Палеозойская эра Сердечно-сосудистая система строение и физиология сердца
Сердечно-сосудистая система строение и физиология сердца Железы внутренней секреции
Железы внутренней секреции Строение и функции кожи (1)
Строение и функции кожи (1) 10 опасных животных на Земле
10 опасных животных на Земле Игра. Угадай животное
Игра. Угадай животное Протерозойская эра
Протерозойская эра Углеводы. Глюкоза
Углеводы. Глюкоза Технология секвенирования генома и сборка генома. Лекция 8
Технология секвенирования генома и сборка генома. Лекция 8 Введение в медицинскую биологию. Структурно-функциональная организация клетки. (Лекция 1)
Введение в медицинскую биологию. Структурно-функциональная организация клетки. (Лекция 1) Цветы для детского сада
Цветы для детского сада Съедобные и несъедобные грибы
Съедобные и несъедобные грибы Физиология размножения животных
Физиология размножения животных