Содержание
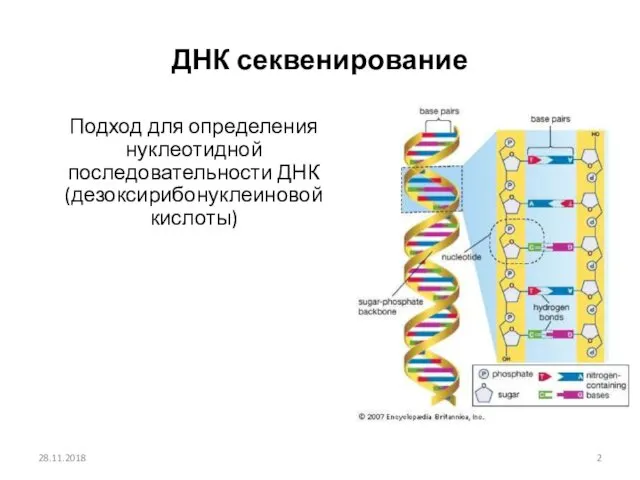
- 2. ДНК секвенирование Подход для определения нуклеотидной последовательности ДНК (дезоксирибонуклеиновой кислоты) 28.11.2018
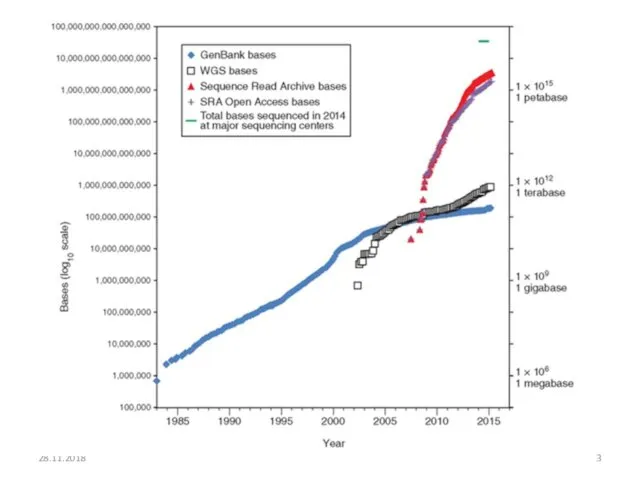
- 3. 28.11.2018
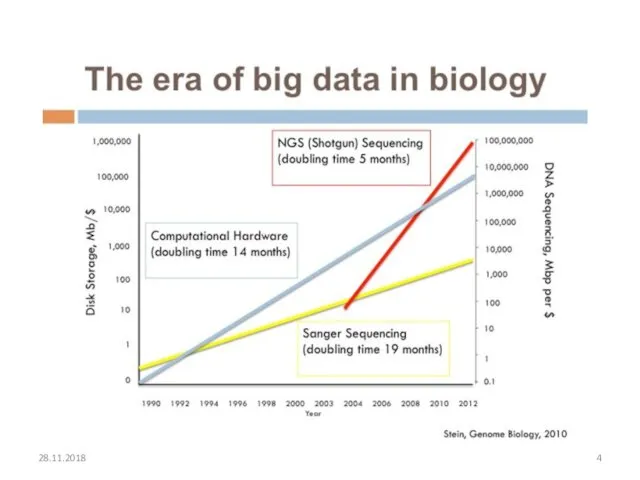
- 4. 28.11.2018
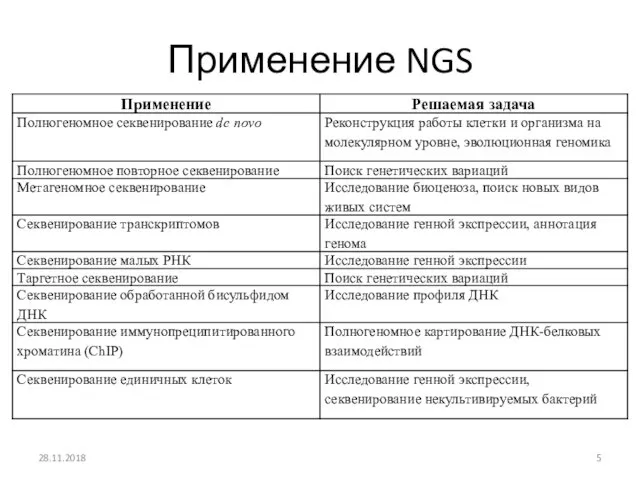
- 5. Применение NGS 28.11.2018
- 6. Основные термины Геномные библиотеки - это коллекция геномной ДНК полученная от одного организма и подготовленная для
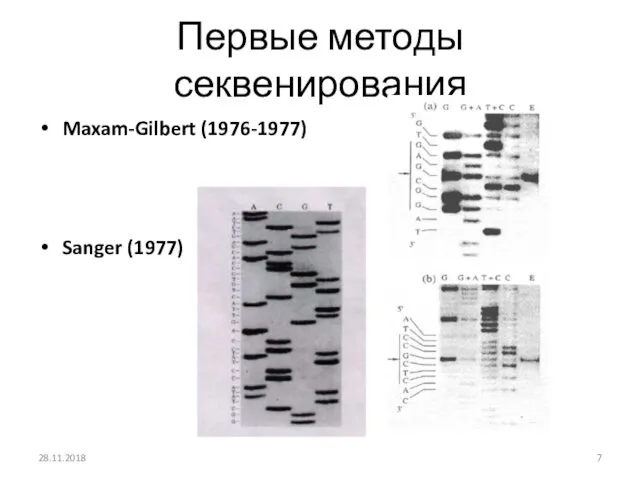
- 7. Первые методы секвенирования Maxam-Gilbert (1976-1977) Sanger (1977) 28.11.2018
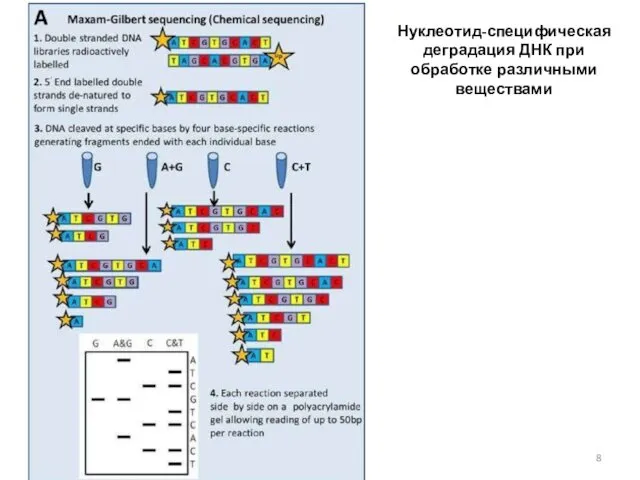
- 8. 28.11.2018 Нуклеотид-специфическая деградация ДНК при обработке различными веществами
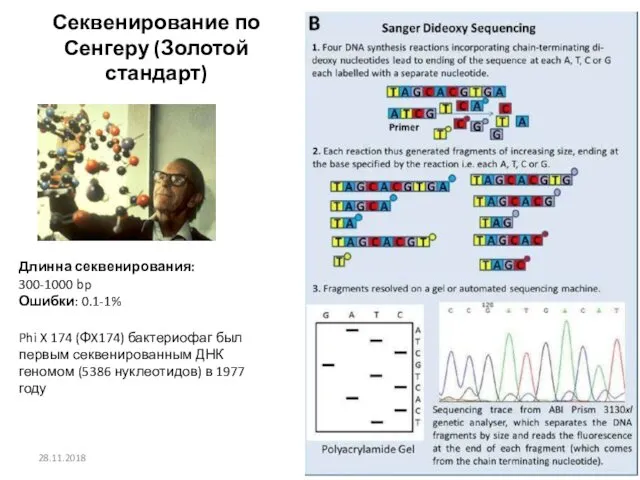
- 9. Секвенирование по Сенгеру (Золотой стандарт) 28.11.2018 Phi X 174 (ΦX174) бактериофаг был первым секвенированным ДНК геномом
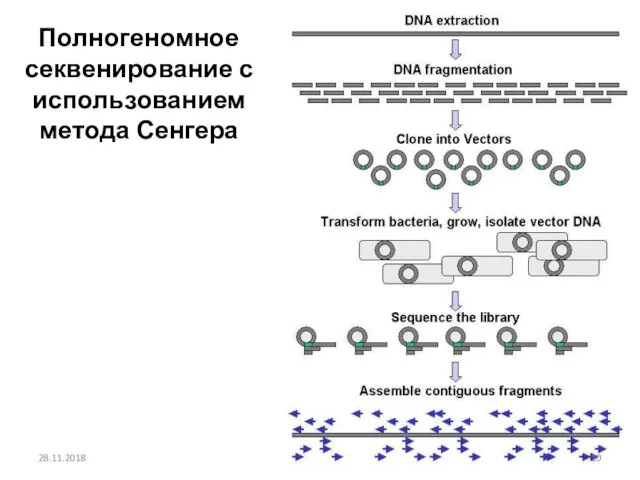
- 10. Полногеномное секвенирование с использованием метода Сенгера 28.11.2018
- 11. Проект геном человека Размер генома – 3.2 Гб Длительность – 10 лет 1990 – 2000 Цена
- 12. Секвенирование по Сенгеру Плюсы: Относительно низкий уровень ошибок Удобное и дешевое секвенирование небольших фрагментов генома (16S
- 13. 28.11.2018
- 14. New Generation Sequencing 28.11.2018 Плюсы: Простая подготовка ДНК библиотек (пробоподготовка) Высокая производительность Низкая стоимость секвенирования Минусы:
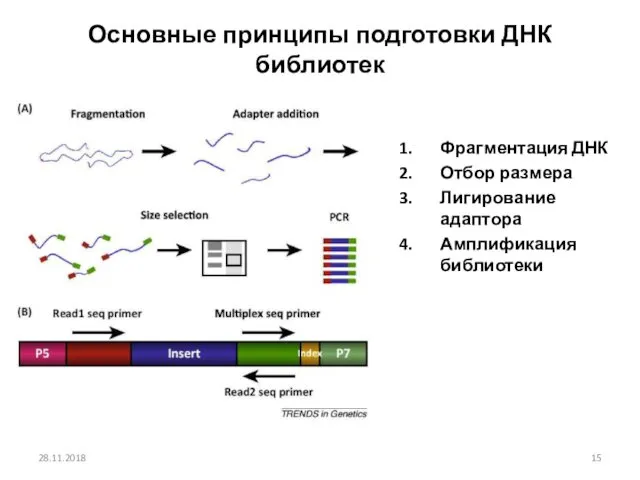
- 15. Основные принципы подготовки ДНК библиотек Фрагментация ДНК Отбор размера Лигирование адаптора Амплификация библиотеки 28.11.2018
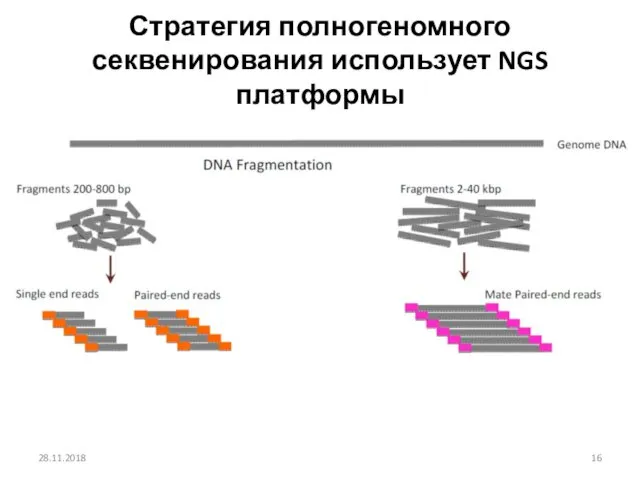
- 16. Стратегия полногеномного секвенирования использует NGS платформы 28.11.2018
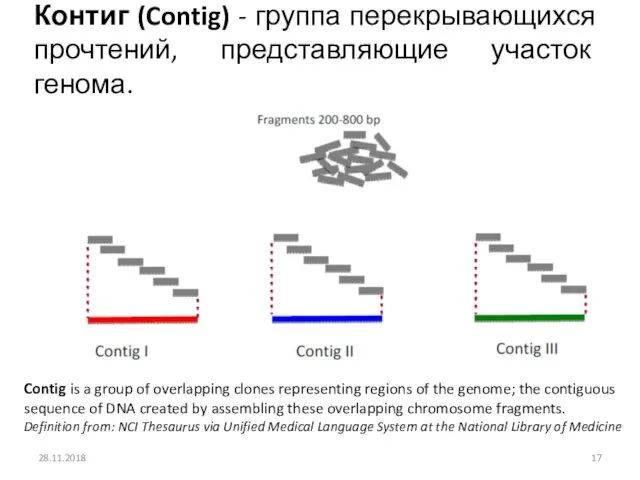
- 17. Контиг (Contig) - группа перекрывающихся прочтений, представляющие участок генома. 28.11.2018 Contig is a group of overlapping
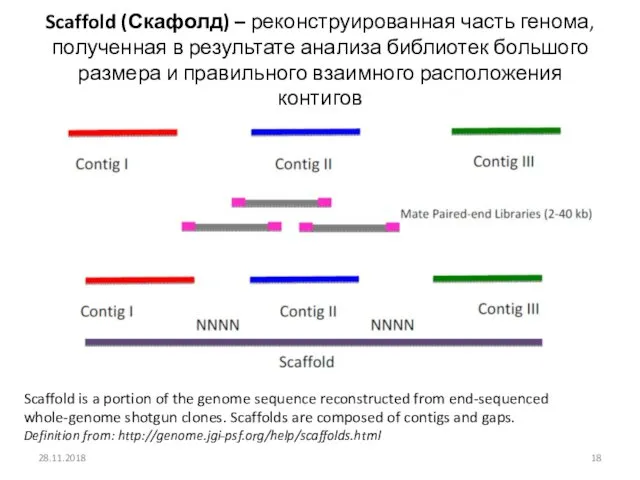
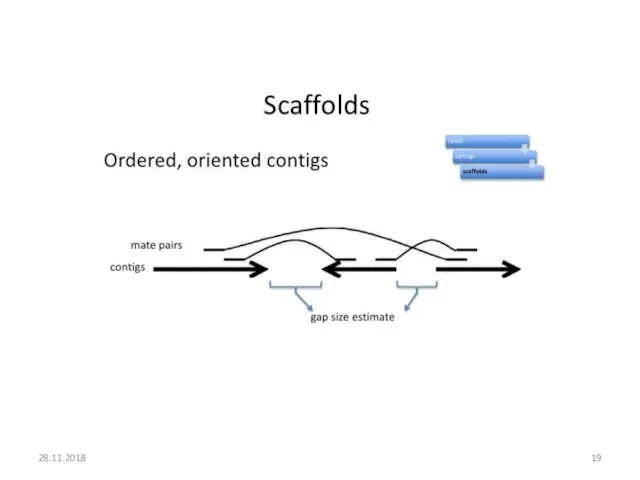
- 18. Scaffold (Скафолд) – реконструированная часть генома, полученная в результате анализа библиотек большого размера и правильного взаимного
- 19. 28.11.2018
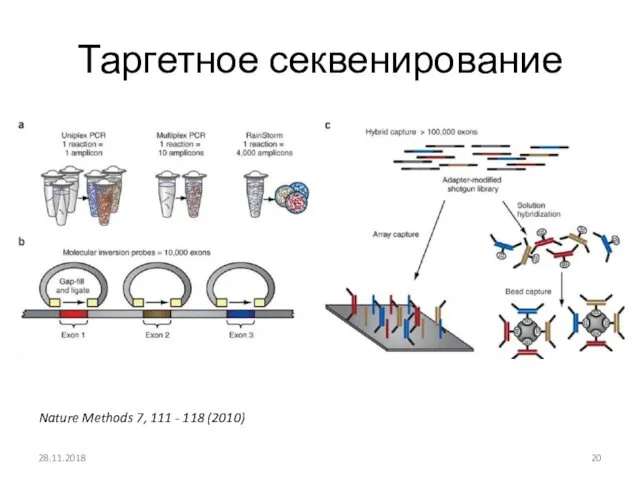
- 20. Таргетное секвенирование 28.11.2018 Nature Methods 7, 111 - 118 (2010)
- 21. Индексирование (Баркодинг) Можно за один запуск секвенатора прочитать несколько геномов или геномных участков Индексы – короткие
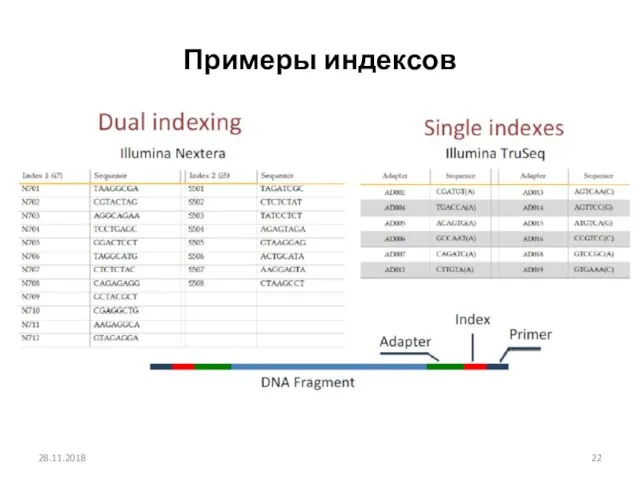
- 22. Примеры индексов 28.11.2018
- 23. Платформы 28.11.2018
- 24. 28.11.2018
- 25. 454 Sequencing Technology 28.11.2018 Фрагментация ДНК Подготовка библиотеки Пришивание адапторов к молекулам ДНК с двух концов.
- 26. 28.11.2018 Один фрагмент = одна бусина (bead) Библиотека фрагментов ДНК прикрепляется к бусинам после денатурации ДНК.
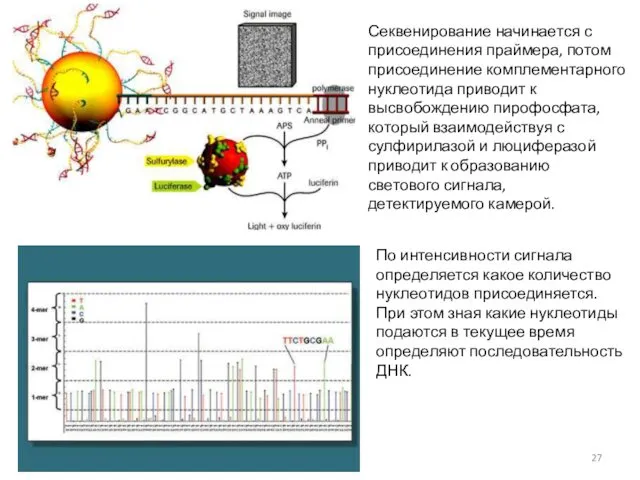
- 27. 28.11.2018 Секвенирование начинается с присоединения праймера, потом присоединение комплементарного нуклеотида приводит к высвобождению пирофосфата, который взаимодействуя
- 28. 28.11.2018
- 29. Ion Torrent Подготовка библиотеки похожа на Roche 454 • фрагментация ДНК • Прикрепление адаптера • Эмульсионная
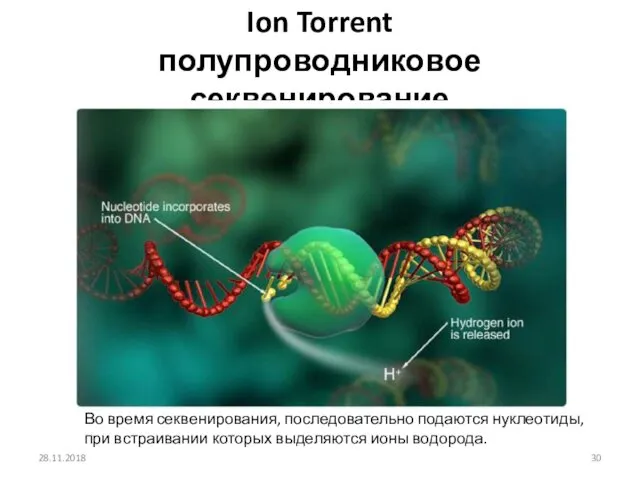
- 30. Ion Torrent полупроводниковое секвенирование 28.11.2018 Во время секвенирования, последовательно подаются нуклеотиды, при встраивании которых выделяются ионы
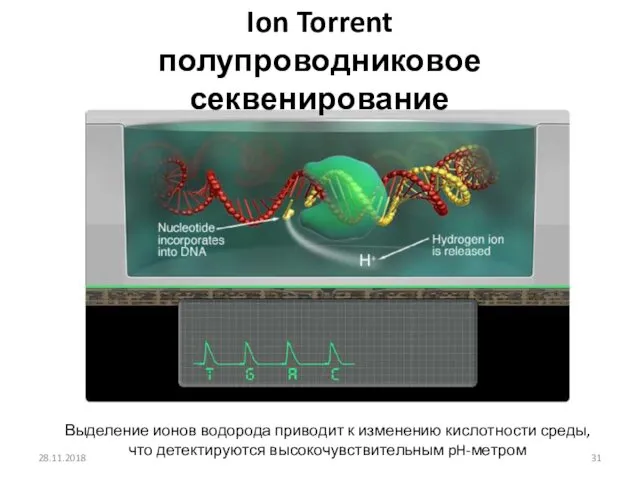
- 31. 28.11.2018 Ion Torrent полупроводниковое секвенирование Выделение ионов водорода приводит к изменению кислотности среды, что детектируются высокочувствительным
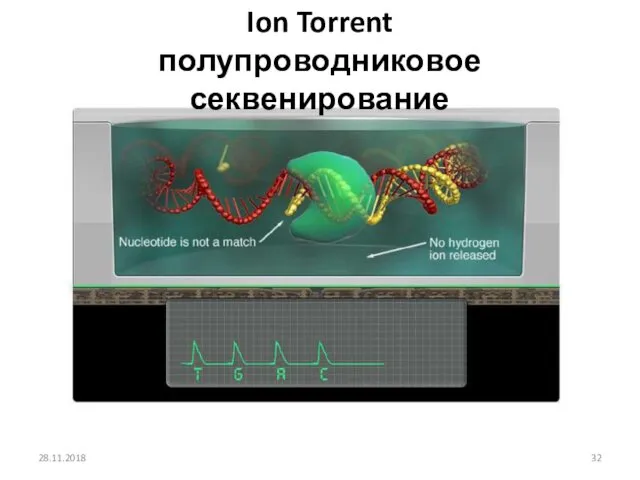
- 32. 28.11.2018 Ion Torrent полупроводниковое секвенирование
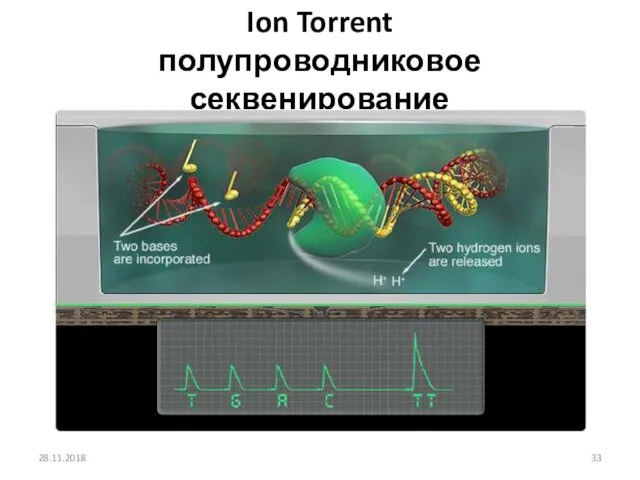
- 33. 28.11.2018 Ion Torrent полупроводниковое секвенирование
- 34. 28.11.2018
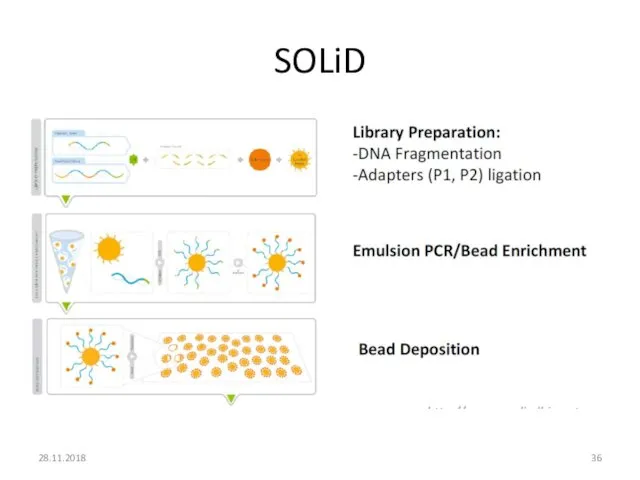
- 35. SOLiD 28.11.2018 Подготовка библиотеки похожа на Roche 454 • фрагментация ДНК • Прикрепление адаптера • Эмульсионная
- 36. SOLiD 28.11.2018
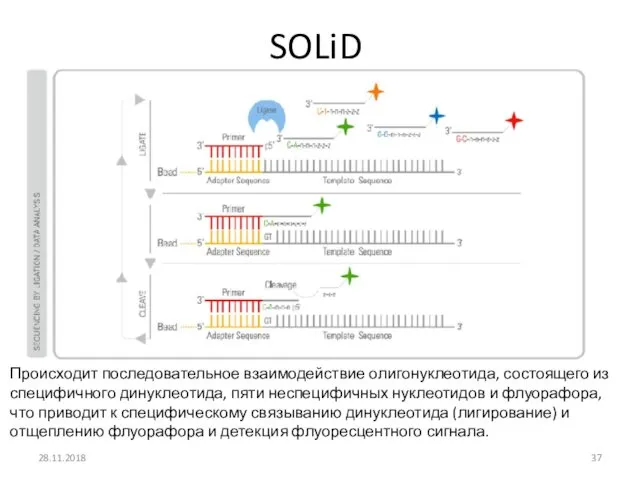
- 37. SOLiD 28.11.2018 Происходит последовательное взаимодействие олигонуклеотида, состоящего из специфичного динуклеотида, пяти неспецифичных нуклеотидов и флуорафора, что
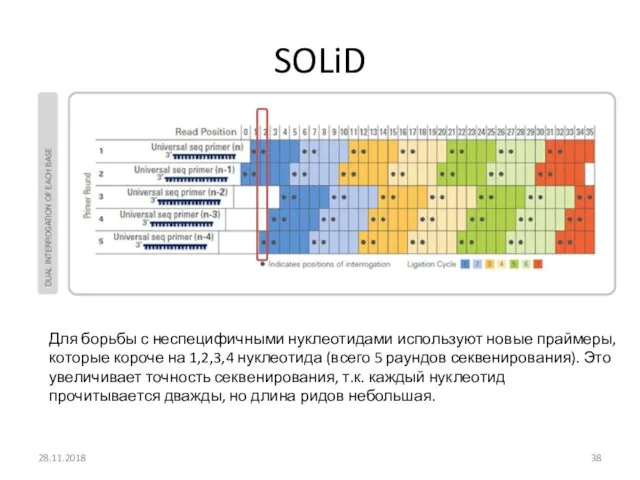
- 38. SOLiD 28.11.2018 Для борьбы с неспецифичными нуклеотидами используют новые праймеры, которые короче на 1,2,3,4 нуклеотида (всего
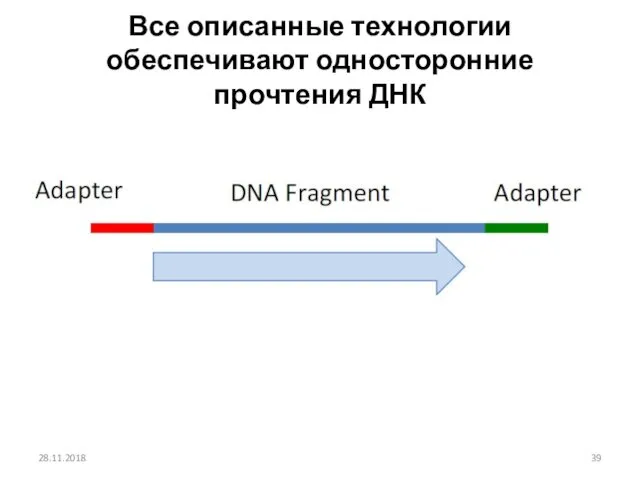
- 39. Все описанные технологии обеспечивают односторонние прочтения ДНК 28.11.2018
- 40. 28.11.2018
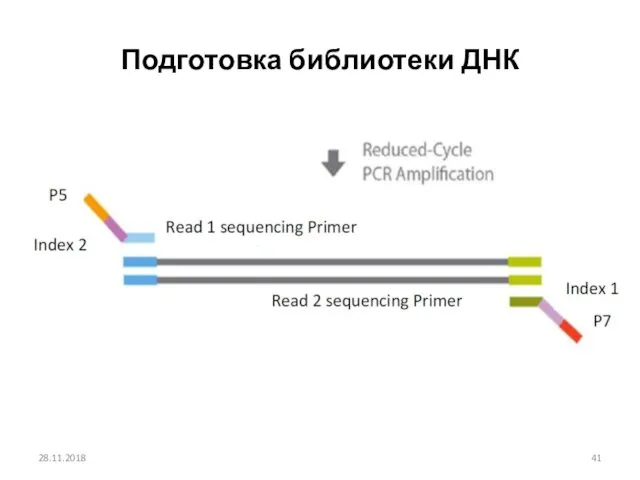
- 41. Подготовка библиотеки ДНК 28.11.2018
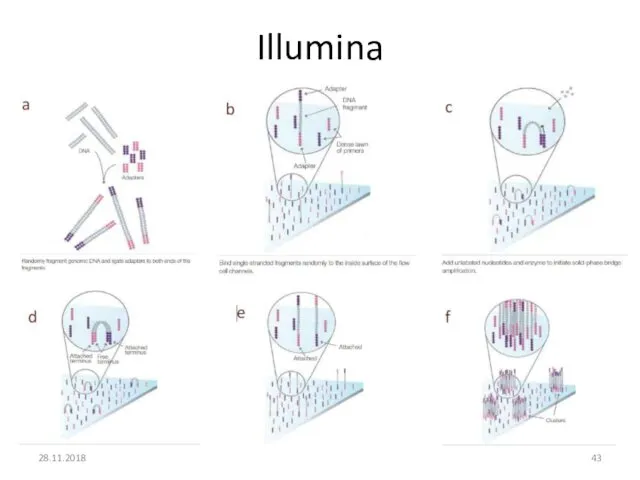
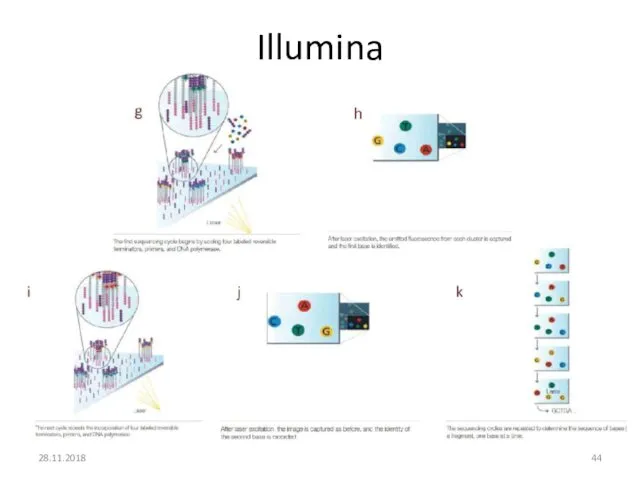
- 42. Illumina Гибридизация ДНК-библиотек Генерация кластеров (ПЦР) Секвенирование синтезом 28.11.2018 http://www.youtube.com/watch?v=HMyCqWhwB8E
- 43. 28.11.2018 Illumina
- 44. 28.11.2018 Illumina
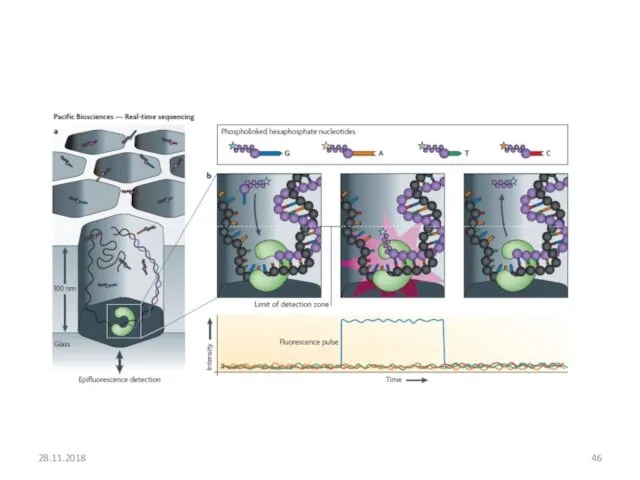
- 45. Pacific Biosciences single molecule real-time (SMRT) sequencing Одномолекулярное секвенирование в реальном времени Секвенировании без амплификации Очень
- 46. 28.11.2018
- 47. 28.11.2018
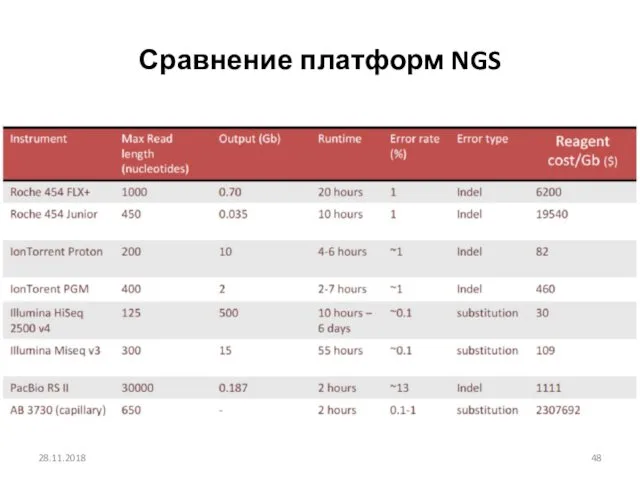
- 48. Сравнение платформ NGS 28.11.2018
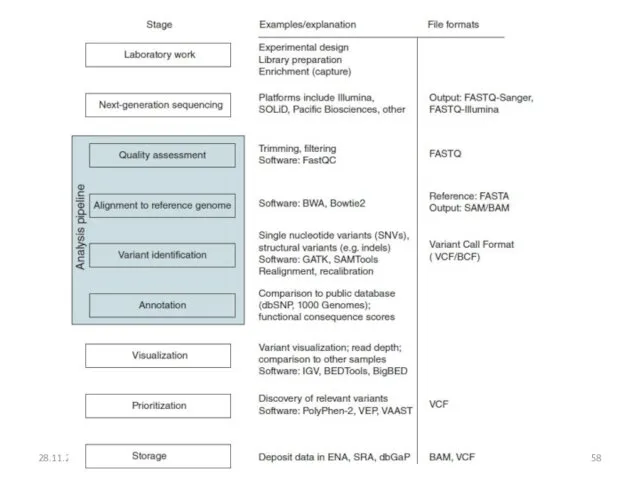
- 49. Контроль качества данных 28.11.2018
- 50. 28.11.2018
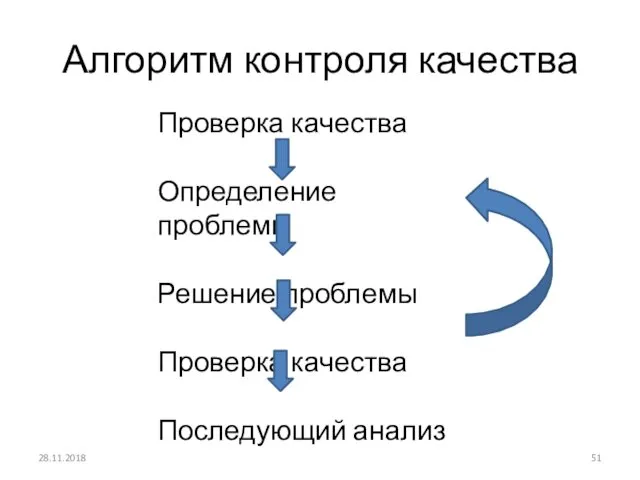
- 51. Алгоритм контроля качества 28.11.2018 Проверка качества Определение проблемы Решение проблемы Проверка качества Последующий анализ
- 52. Зачем чистить данные? 28.11.2018 • Риды низкого качества • Контаминация (примесь ДНК другого организма) • Служебные
- 53. FASTA и FASTQ форматы 28.11.2018 FASTA FASTQ Линия начинающаяся с @ содержит идентификатор последовательности Последовательность Линия
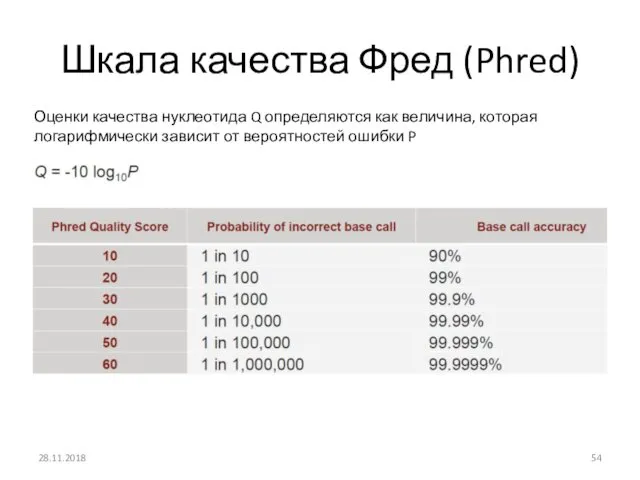
- 54. Шкала качества Фред (Phred) 28.11.2018 Оценки качества нуклеотида Q определяются как величина, которая логарифмически зависит от
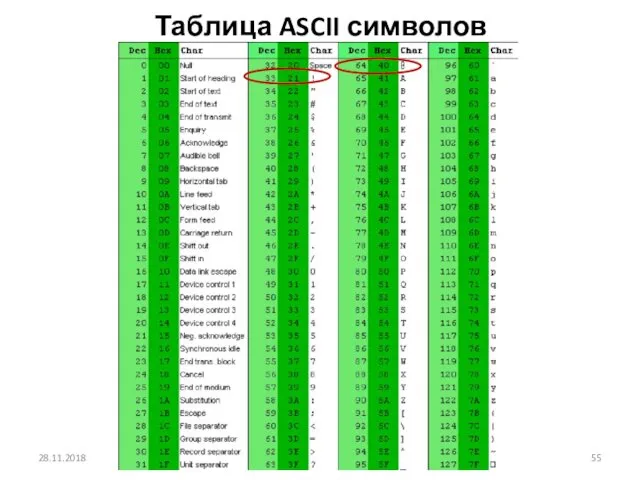
- 55. Таблица ASCII символов 28.11.2018
- 56. Разные Phred шкалы 28.11.2018
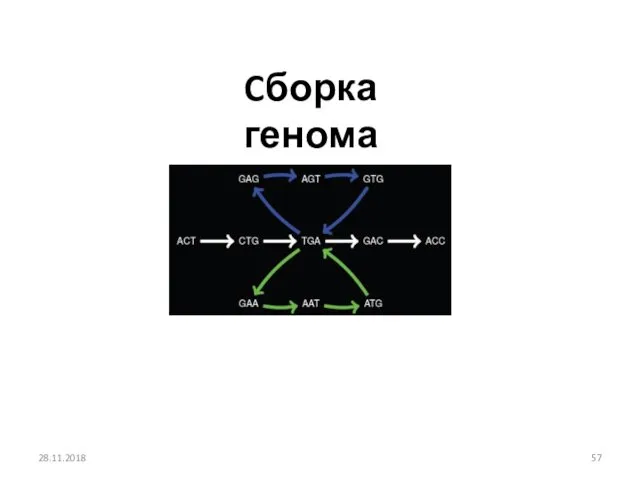
- 57. Cборка генома 28.11.2018
- 58. 28.11.2018
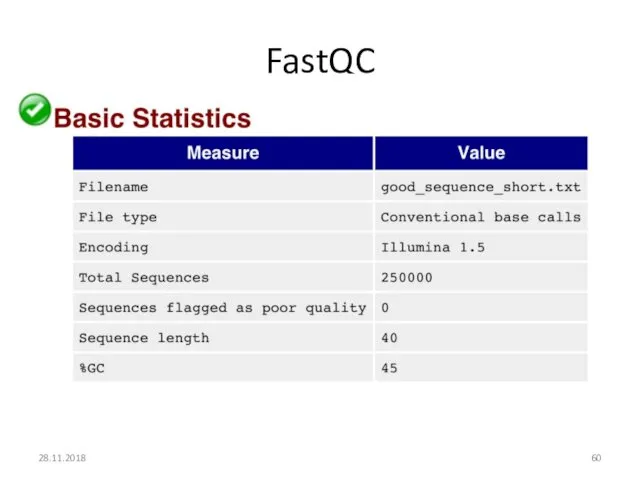
- 59. FastQC – инструмент для контроля качества данных На вход – исходные данные с секвенатора HTML отчет
- 60. FastQC 28.11.2018
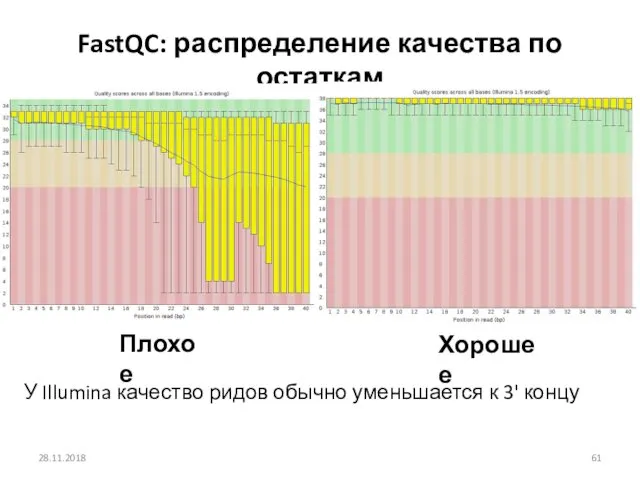
- 61. FastQC: распределение качества по остаткам 28.11.2018 Плохое Хорошее У Illumina качество ридов обычно уменьшается к 3'
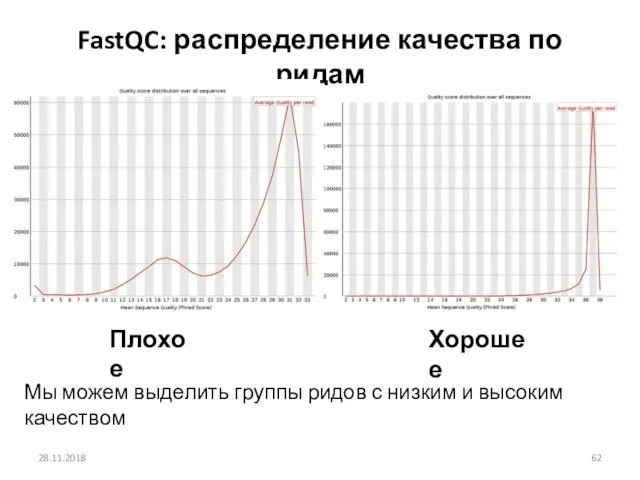
- 62. 28.11.2018 FastQC: распределение качества по ридам Плохое Хорошее Мы можем выделить группы ридов с низким и
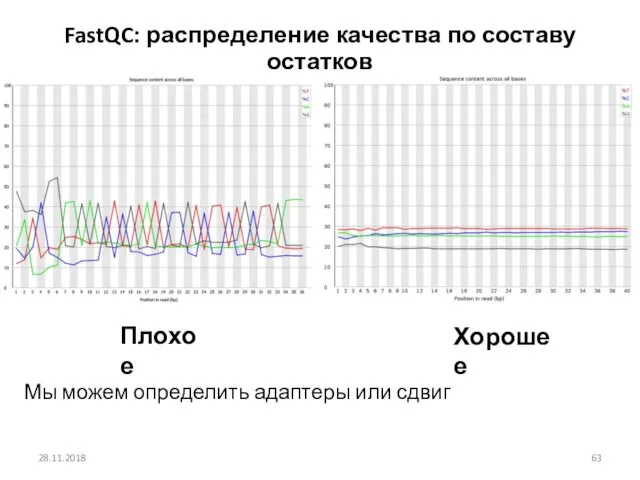
- 63. 28.11.2018 FastQC: распределение качества по составу остатков Плохое Хорошее Мы можем определить адаптеры или сдвиг
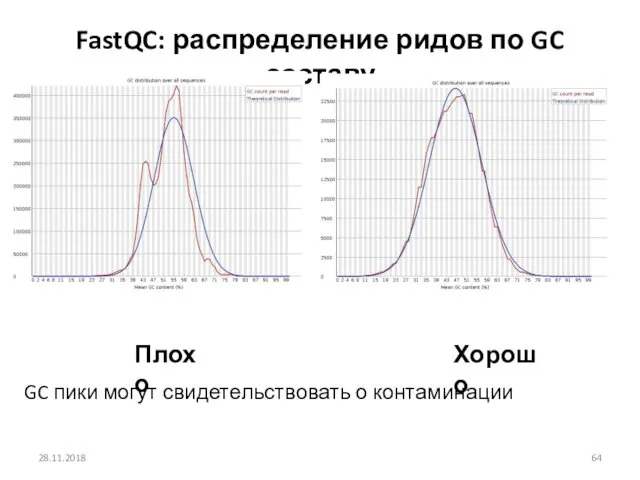
- 64. 28.11.2018 FastQC: распределение ридов по GC составу Плохо Хорошо GC пики могут свидетельствовать о контаминации
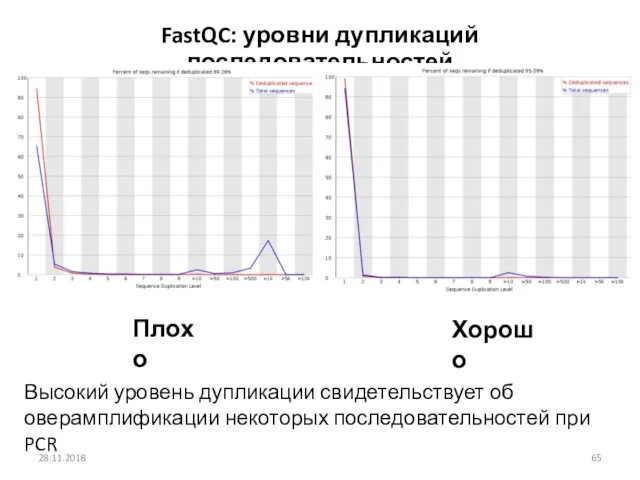
- 65. 28.11.2018 FastQC: уровни дупликаций последовательностей Плохо Хорошо Высокий уровень дупликации свидетельствует об оверамплификации некоторых последовательностей при
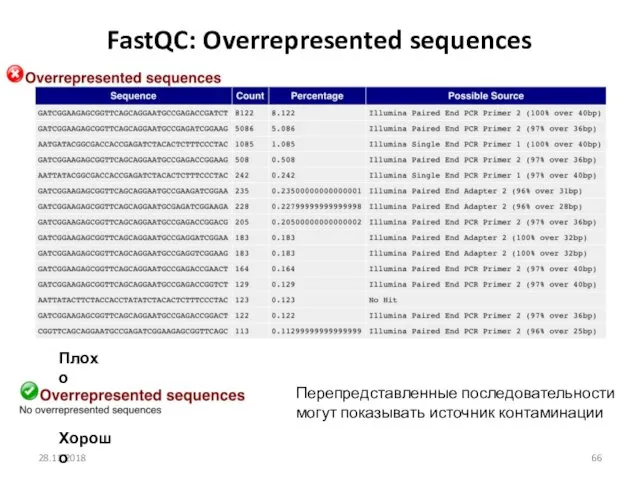
- 66. 28.11.2018 FastQC: Overrepresented sequences Плохо Хорошо Перепредставленные последовательности могут показывать источник контаминации
- 67. 28.11.2018 FastQC: Качество ячеек Плохо Хорошо У Illumina можно определить проблемы с ячейками
- 68. Шаги препроцессинга Фильтрация данных по качеству Удаление ридов, качество которых ниже определенного порога; Обрезание части ридов,
- 69. У нас есть очищенные данные. Что дальше? Сборка de novo Сборка по референсному геному Выравнивание с
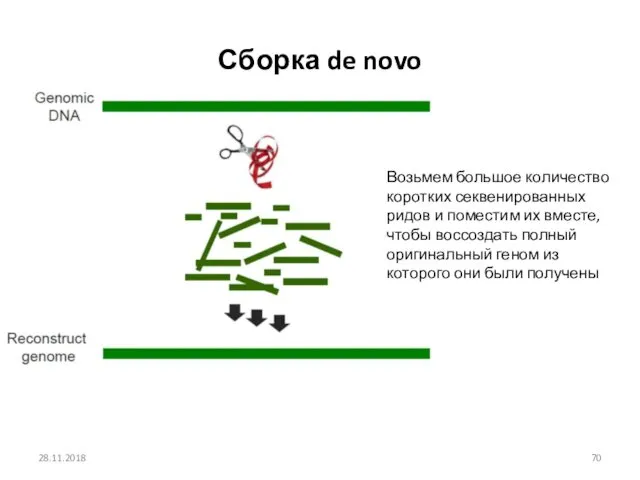
- 70. Сборка de novo 28.11.2018 Возьмем большое количество коротких секвенированных ридов и поместим их вместе, чтобы воссоздать
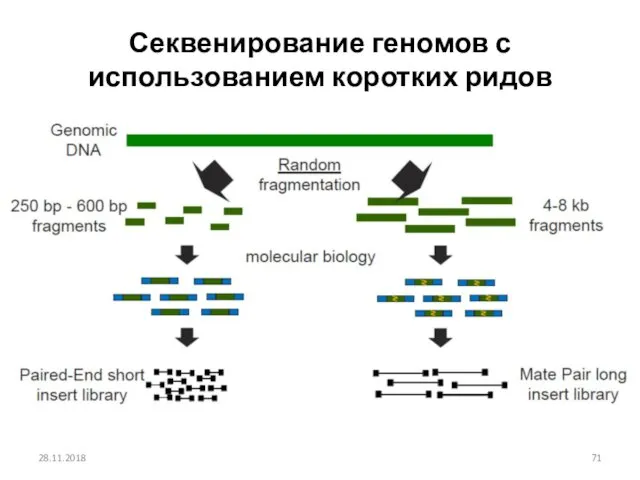
- 71. Секвенирование геномов с использованием коротких ридов 28.11.2018
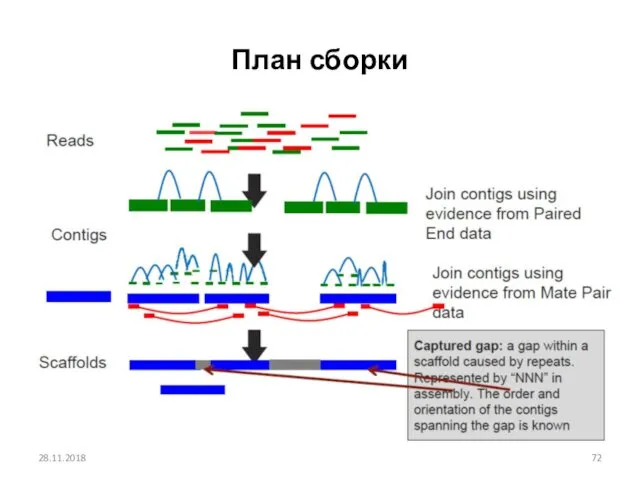
- 72. План сборки 28.11.2018
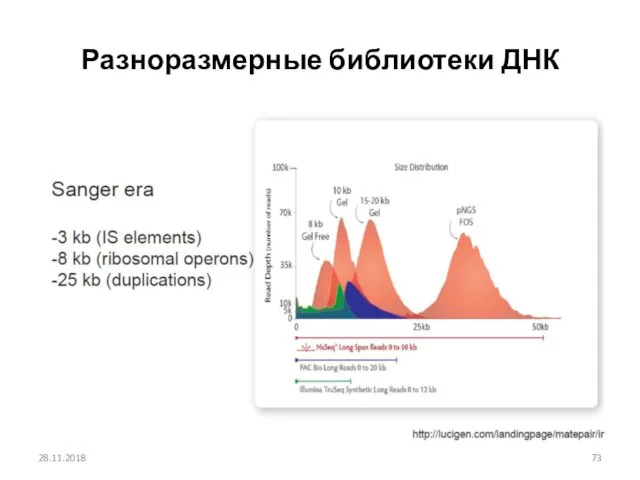
- 73. Разноразмерные библиотеки ДНК 28.11.2018
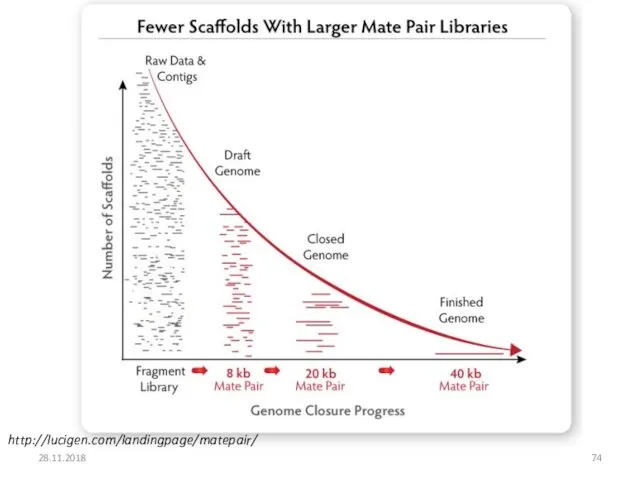
- 74. 28.11.2018 http://lucigen.com/landingpage/matepair/
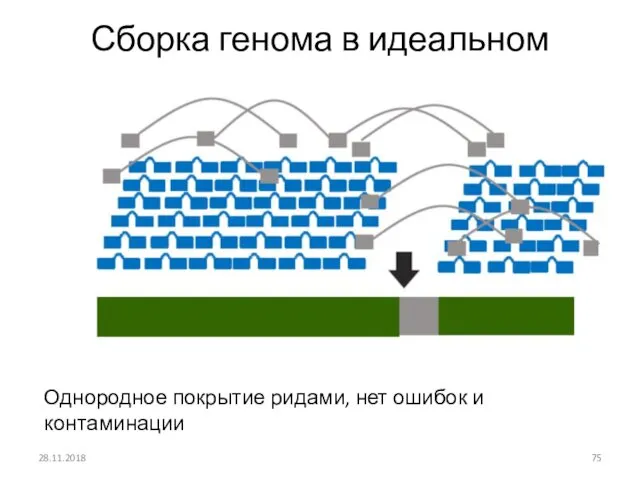
- 75. Сборка генома в идеальном случае 28.11.2018 Однородное покрытие ридами, нет ошибок и контаминации
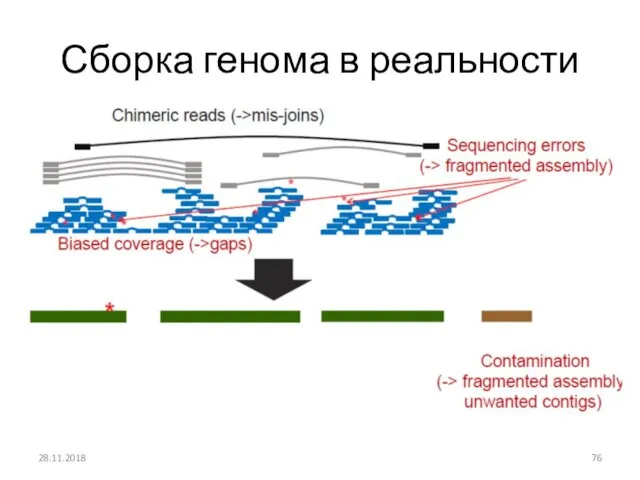
- 76. Сборка генома в реальности 28.11.2018
- 77. 28.11.2018 Кафедра биоинформатики МБФ РНИМУ
- 78. Выбор правильной программы - сборщика геномов (ассемблер) На сколько большой геном? Существуют ли известные особенности этого
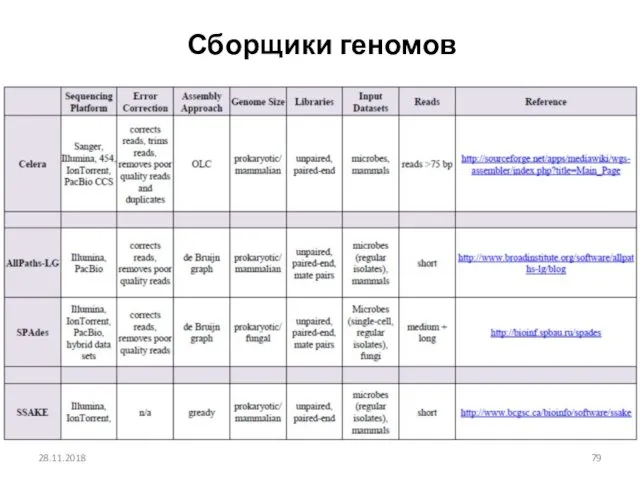
- 79. Сборщики геномов 28.11.2018
- 80. Оценка качества сборки генома Количество контигов Общая длинна всех контигов Длинна наибольшего контига Количество неправильно собранных
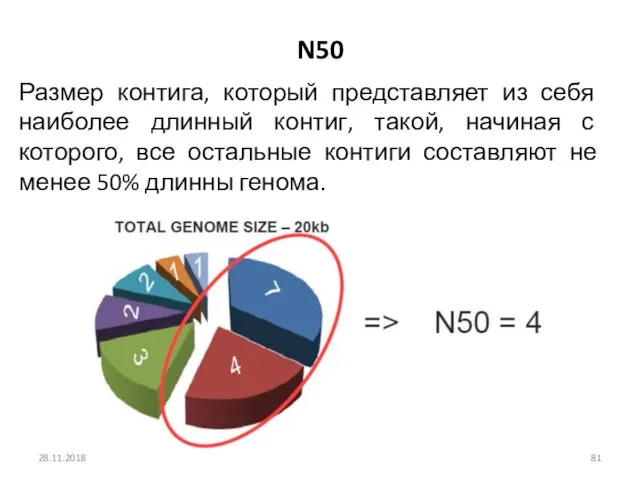
- 81. N50 Размер контига, который представляет из себя наиболее длинный контиг, такой, начиная с которого, все остальные
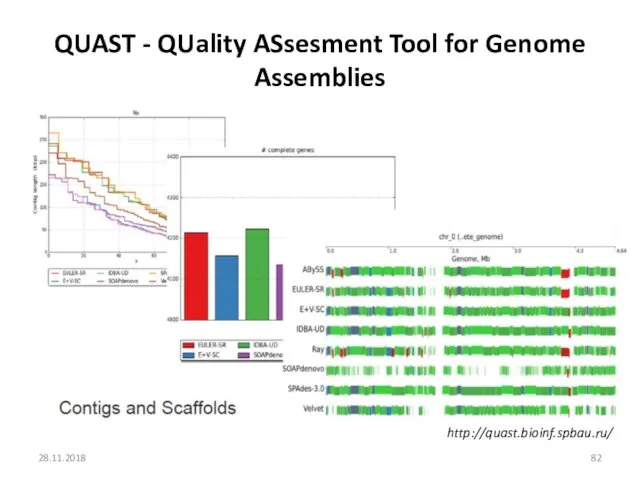
- 82. QUAST - QUality ASsesment Tool for Genome Assemblies 28.11.2018 http://quast.bioinf.spbau.ru/
- 83. 28.11.2018
- 84. Реальные графы де Брюйна 28.11.2018
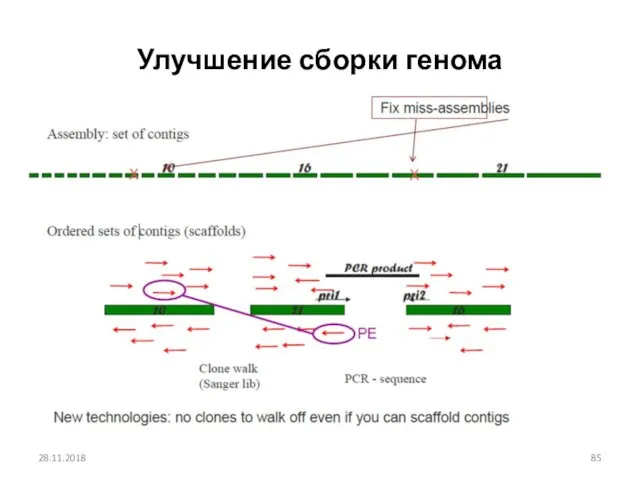
- 85. Улучшение сборки генома 28.11.2018
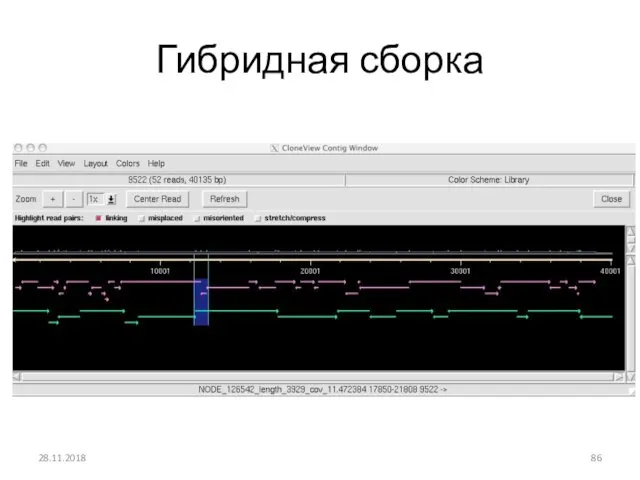
- 86. Гибридная сборка 28.11.2018
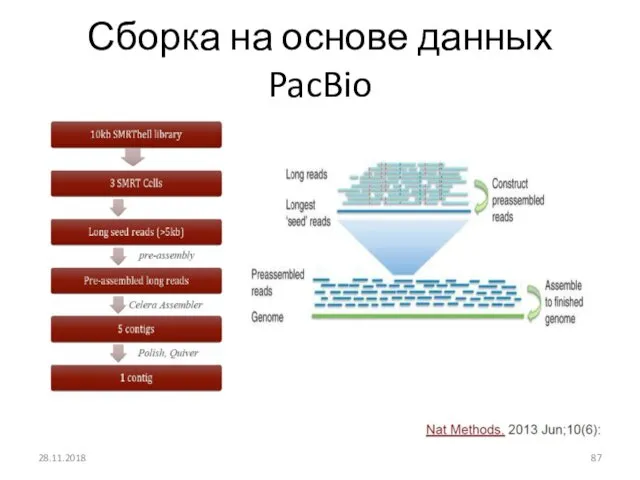
- 87. Сборка на основе данных PacBio 28.11.2018
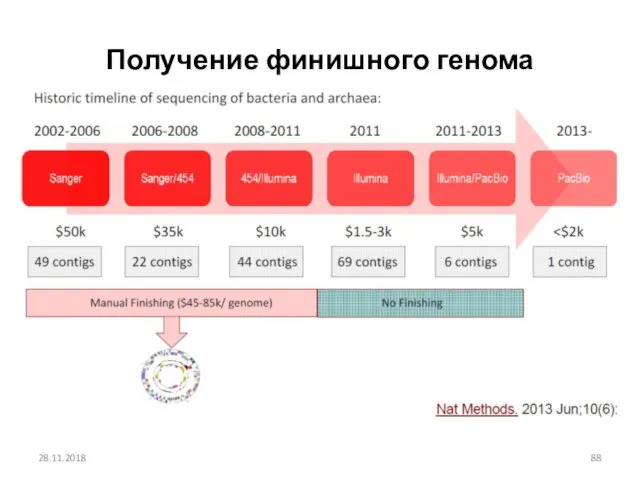
- 88. Получение финишного генома 28.11.2018
- 89. Зачем нужны финишные геномы? Функциональные геномные исследования требуют высококачественной, полной последовательности генома в качестве отправной точки
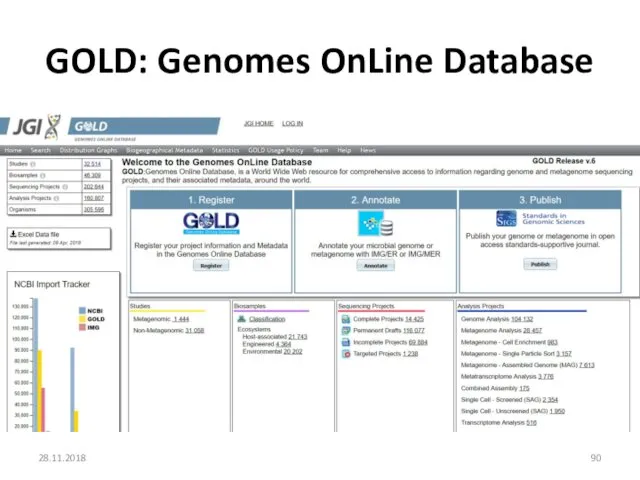
- 90. GOLD: Genomes OnLine Database 28.11.2018
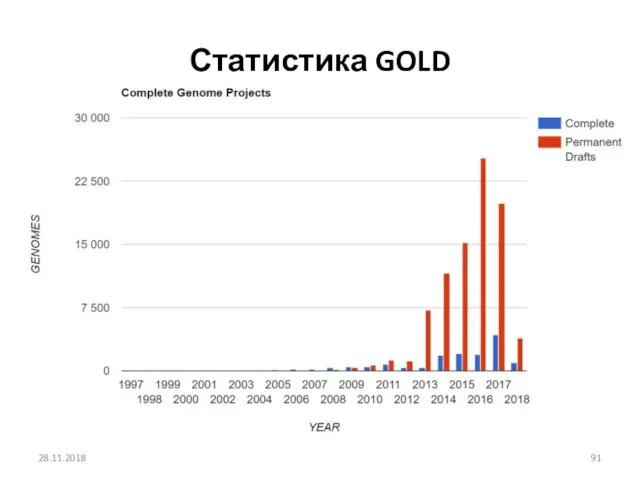
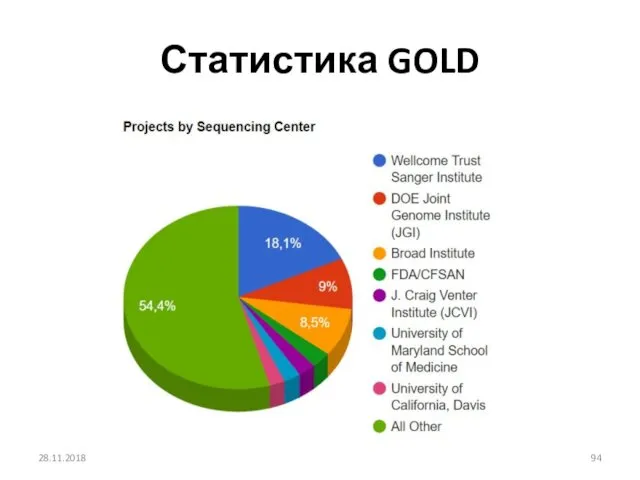
- 91. Статистика GOLD 28.11.2018
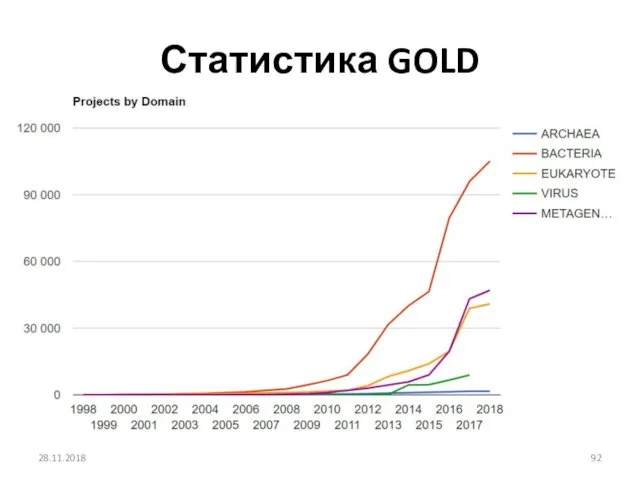
- 92. Статистика GOLD 28.11.2018
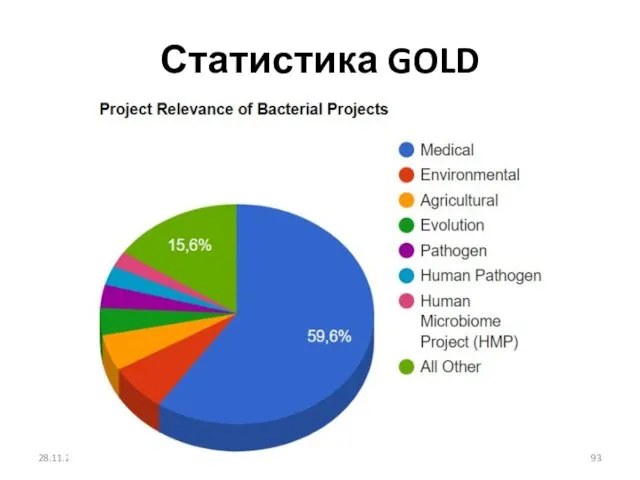
- 93. Статистика GOLD 28.11.2018
- 94. Статистика GOLD 28.11.2018
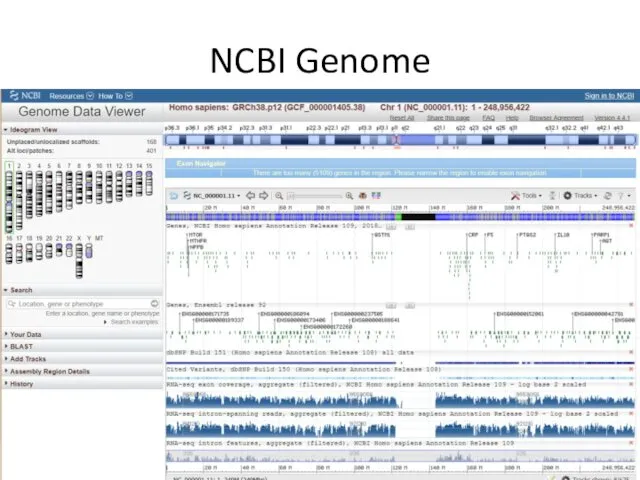
- 95. NCBI Genome 28.11.2018
- 96. NCBI Genome 28.11.2018
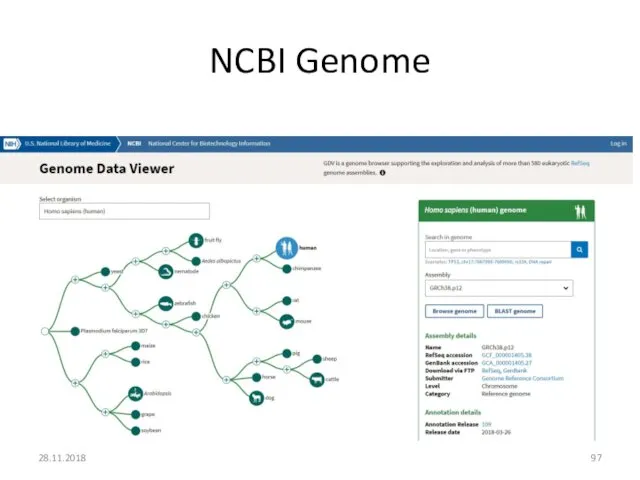
- 97. NCBI Genome 28.11.2018
- 98. NCBI Genome 28.11.2018
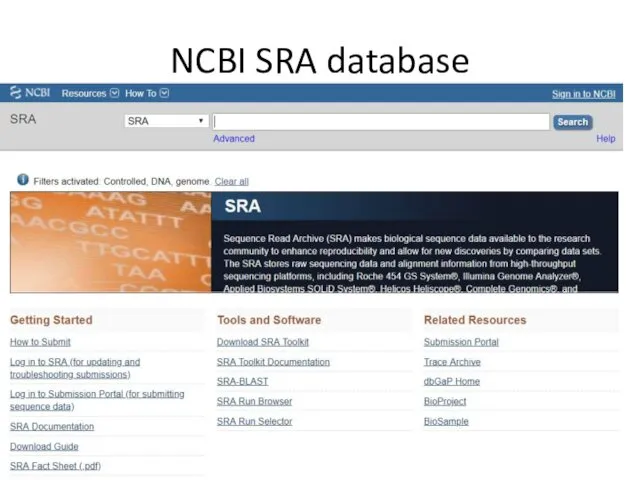
- 99. NCBI SRA database 28.11.2018
- 101. Скачать презентацию
































 Все о картофеле
Все о картофеле Орган слуха
Орган слуха Жилкування листків
Жилкування листків Кисломолочные бактерии. 5 класс
Кисломолочные бактерии. 5 класс Мимические мышцы лица
Мимические мышцы лица Интересные факты про бобра
Интересные факты про бобра Огород без химии, или бюджетные удобрения
Огород без химии, или бюджетные удобрения Матричные биосинтезы
Матричные биосинтезы Класс Пресмыкающиеся, или Рептилии. Отряд Чешуйчатые
Класс Пресмыкающиеся, или Рептилии. Отряд Чешуйчатые Класс Паукообразные
Класс Паукообразные Эпизоотологическое и эпидемиологическое значение грызунов. Дератизация
Эпизоотологическое и эпидемиологическое значение грызунов. Дератизация Биохимия нервной ткани
Биохимия нервной ткани Хищные птицы Республики Коми
Хищные птицы Республики Коми Открытый урок. Генетика как наука
Открытый урок. Генетика как наука В1 витамині
В1 витамині Визначення типу шкіри на різних ділянках обличчя та складання правил догляду за власною шкірою
Визначення типу шкіри на різних ділянках обличчя та складання правил догляду за власною шкірою Ауыз қуысы. Ауыз қуысы мүшелері және оның паталогиясы
Ауыз қуысы. Ауыз қуысы мүшелері және оның паталогиясы Тестовая работа по теме Корень. Строение корня, 6 класс.
Тестовая работа по теме Корень. Строение корня, 6 класс. Теории происхождения человека
Теории происхождения человека Цветковые (покрытосемянные) растения
Цветковые (покрытосемянные) растения Пагін і його будова
Пагін і його будова Презентация Технология Развитие критического мышления через чтение и письмо - способ формирования системы универсальных учебных действий на уроках биологии.
Презентация Технология Развитие критического мышления через чтение и письмо - способ формирования системы универсальных учебных действий на уроках биологии. Бактерії і актиноміцети- як збудники хвороб рослин. Лекція №4
Бактерії і актиноміцети- як збудники хвороб рослин. Лекція №4 Клетка – основная структурная единица организма
Клетка – основная структурная единица организма Законы Менделя
Законы Менделя Височная кость. Каналы височной кости
Височная кость. Каналы височной кости Хвойные растения
Хвойные растения презентация на тему Плоды
презентация на тему Плоды