Содержание
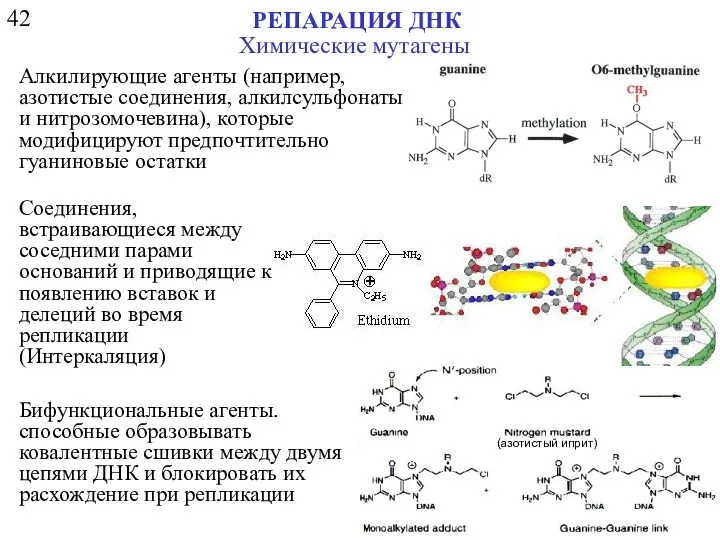
- 3. 42 РЕПАРАЦИЯ ДНК Химические мутагены Алкилирующие агенты (например, азотистые соединения, алкилсульфонаты и нитрозомочевина), которые модифицируют предпочтительно
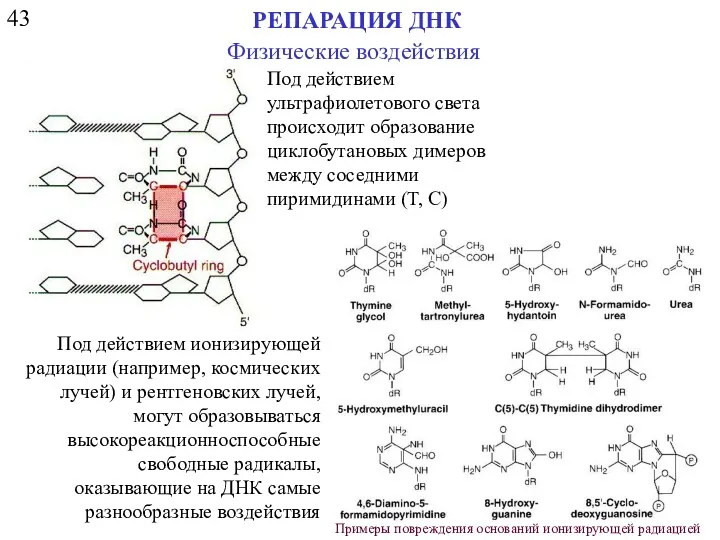
- 4. 43 РЕПАРАЦИЯ ДНК Физические воздействия Под действием ионизирующей радиации (например, космических лучей) и рентгеновских лучей, могут
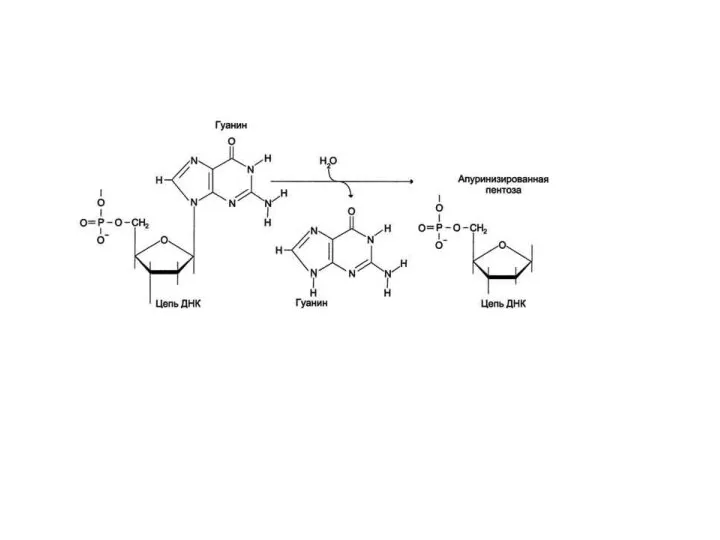
- 5. 44 ОСНОВНЫЕ ТИПЫ РЕПАРАЦИОННЫХ ПРОЦЕССОВ 1) Непосредственное исправление модификаций или неправильных спариваний, не требующее репликации для
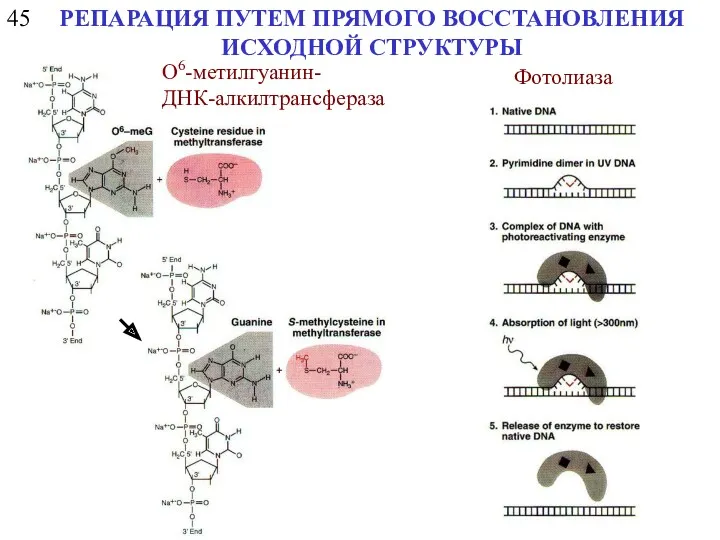
- 6. 45 РЕПАРАЦИЯ ПУТЕМ ПРЯМОГО ВОССТАНОВЛЕНИЯ ИСХОДНОЙ СТРУКТУРЫ O6-метилгуанин- ДНК-алкилтрансфераза Фотолиаза
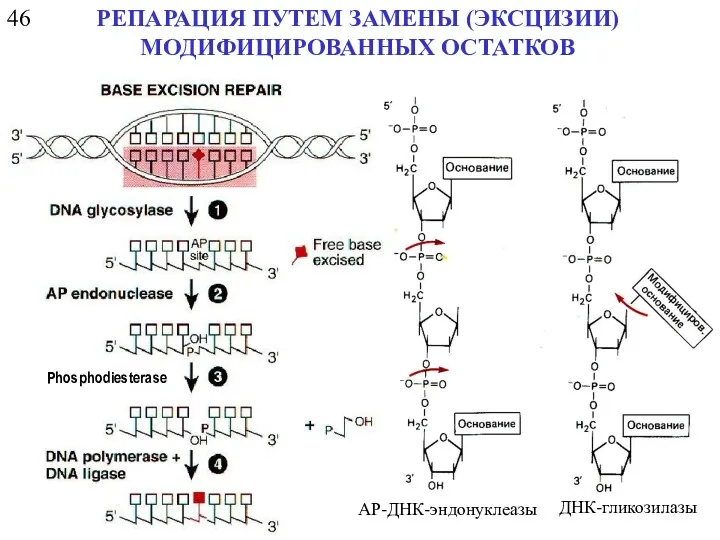
- 7. 46 РЕПАРАЦИЯ ПУТЕМ ЗАМЕНЫ (ЭКСЦИЗИИ) МОДИФИЦИРОВАННЫХ ОСТАТКОВ ДНК-гликозилазы AP-ДНК-эндонуклеазы Phosphodiesterase
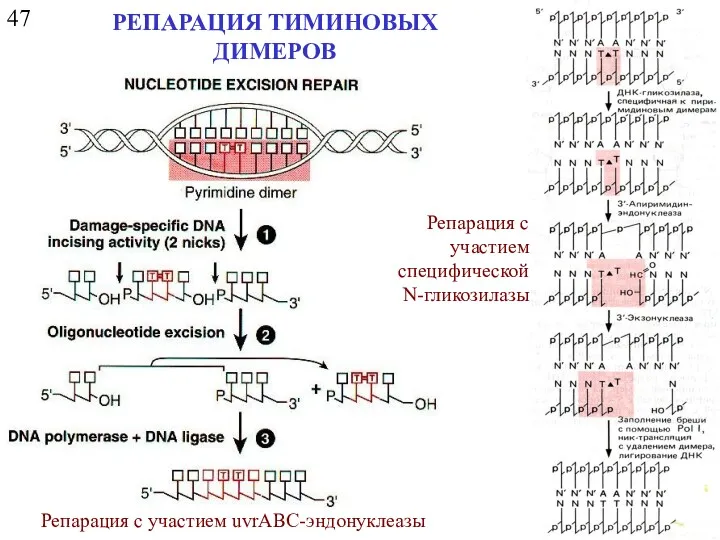
- 8. 47 РЕПАРАЦИЯ ТИМИНОВЫХ ДИМЕРОВ Репарация с участием uvrABC-эндонуклеазы Репарация с участием специфической N-гликозилазы
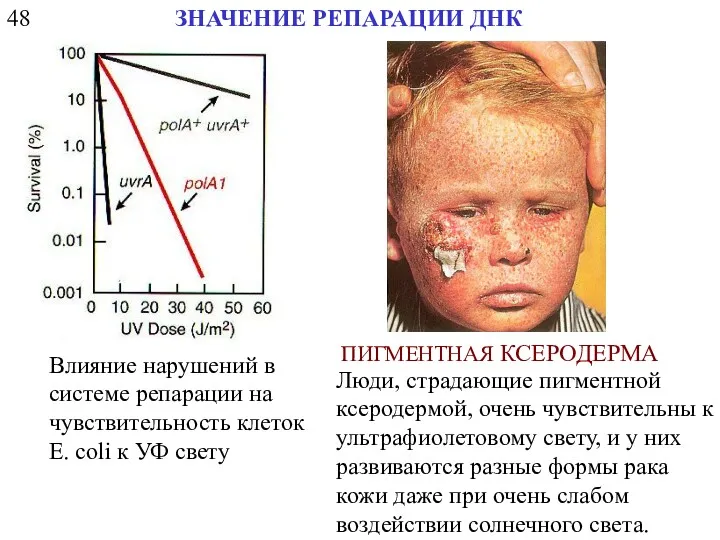
- 9. 48 ЗНАЧЕНИЕ РЕПАРАЦИИ ДНК ПИГМЕНТНАЯ КСЕРОДЕРМА Люди, страдающие пигментной ксеродермой, очень чувствительны к ультрафиолетовому свету, и
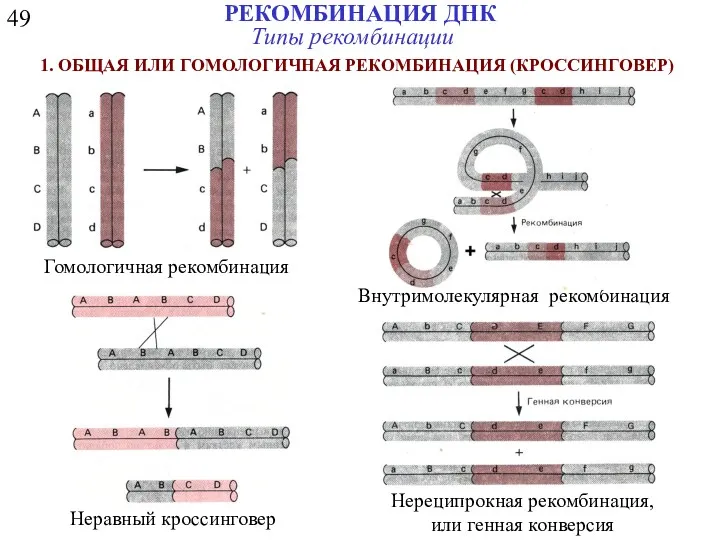
- 10. 49 РЕКОМБИНАЦИЯ ДНК Типы рекомбинации 1. ОБЩАЯ ИЛИ ГОМОЛОГИЧНАЯ РЕКОМБИНАЦИЯ (КРОССИНГОВЕР) Гомологичная рекомбинация Неравный кроссинговер Внутримолекулярная
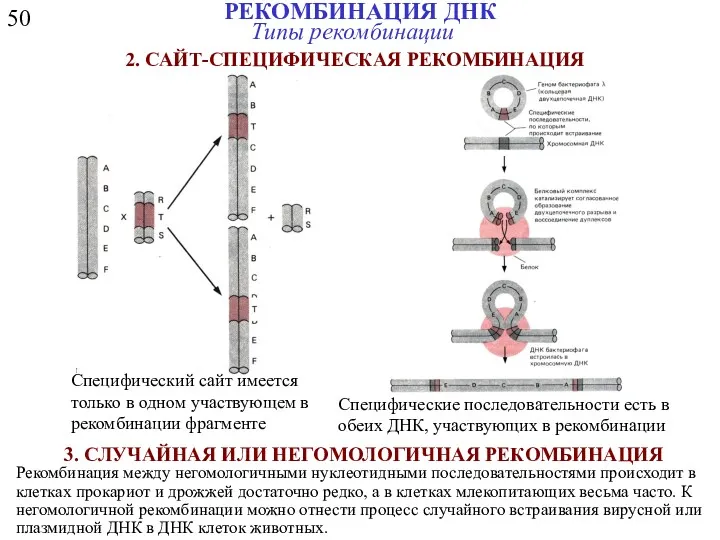
- 11. 50 РЕКОМБИНАЦИЯ ДНК Типы рекомбинации 3. СЛУЧАЙНАЯ ИЛИ НЕГОМОЛОГИЧНАЯ РЕКОМБИНАЦИЯ Специфический сайт имеется только в одном
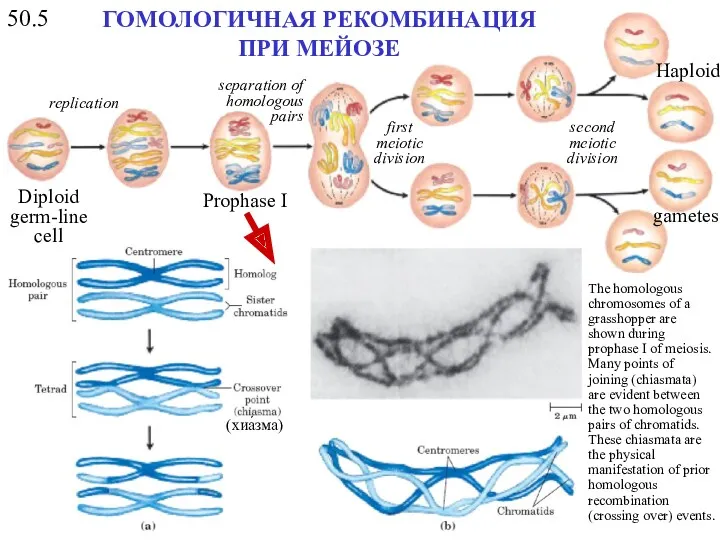
- 12. 50.5 The homologous chromosomes of a grasshopper are shown during prophase I of meiosis. Many points
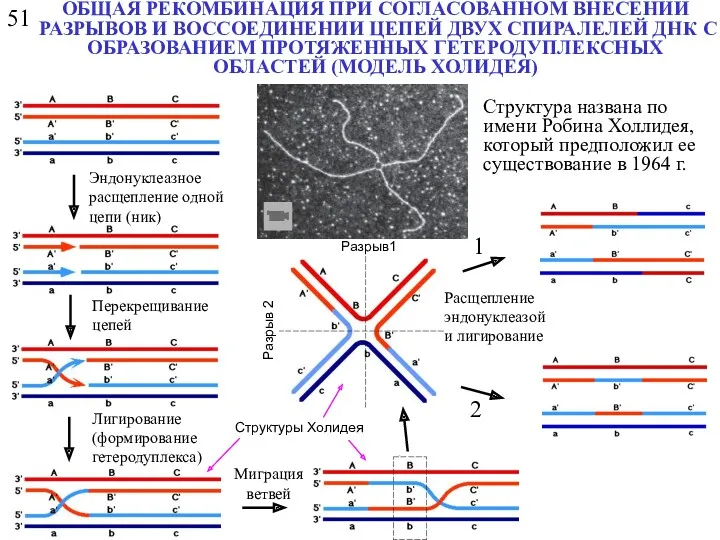
- 13. 51 Разрыв1 Разрыв 2 1 2 Эндонуклеазное расщепление одной цепи (ник) Перекрещивание цепей Лигирование (формирование гетеродуплекса)
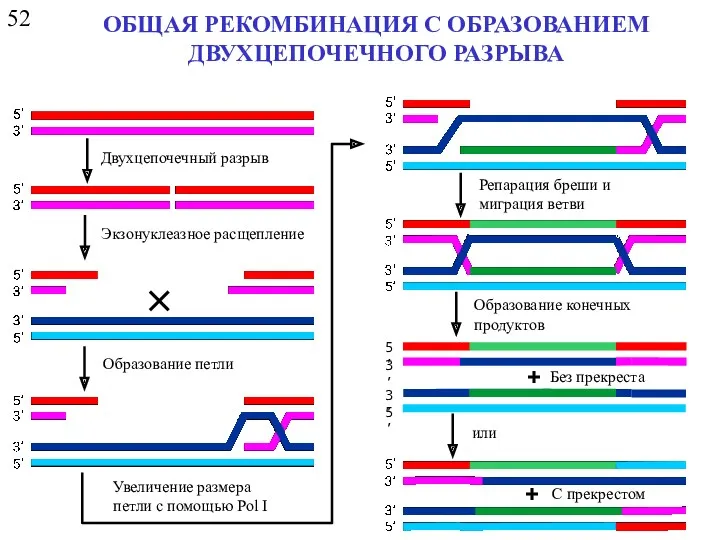
- 14. 52 ОБЩАЯ РЕКОМБИНАЦИЯ С ОБРАЗОВАНИЕМ ДВУХЦЕПОЧЕЧНОГО РАЗРЫВА Двухцепочечный разрыв Экзонуклеазное расщепление Образование петли Увеличение размера петли
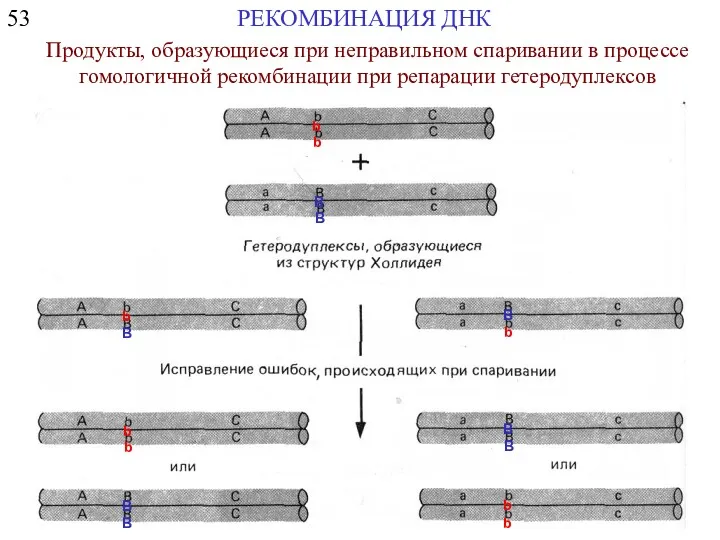
- 15. 53 РЕКОМБИНАЦИЯ ДНК Продукты, образующиеся при неправильном спаривании в процессе гомологичной рекомбинации при репарации гетеродуплексов b
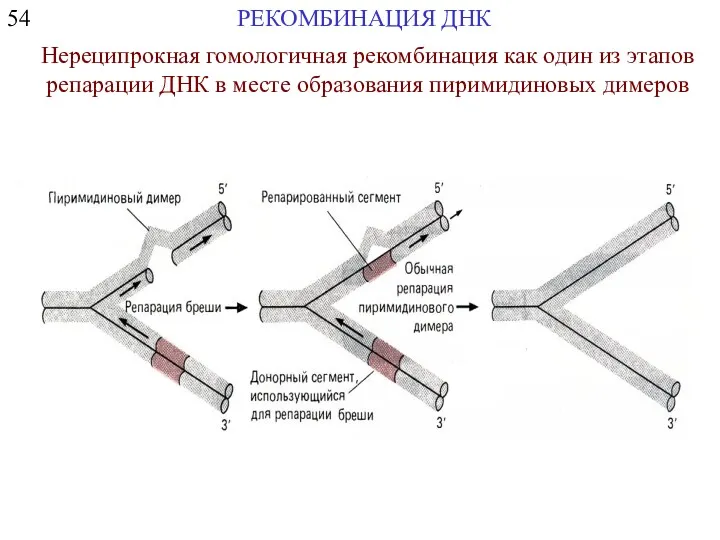
- 16. 54 РЕКОМБИНАЦИЯ ДНК Нереципрокная гомологичная рекомбинация как один из этапов репарации ДНК в месте образования пиримидиновых
- 17. ФЕРМЕНТЫ, УЧАСТВУЮЩИЕ В РЕКОМБИНАЦИИ ДНК 54.5 RесА-белок катализирует обмен одиночными цепями, используя энергию гидролиза АТР до
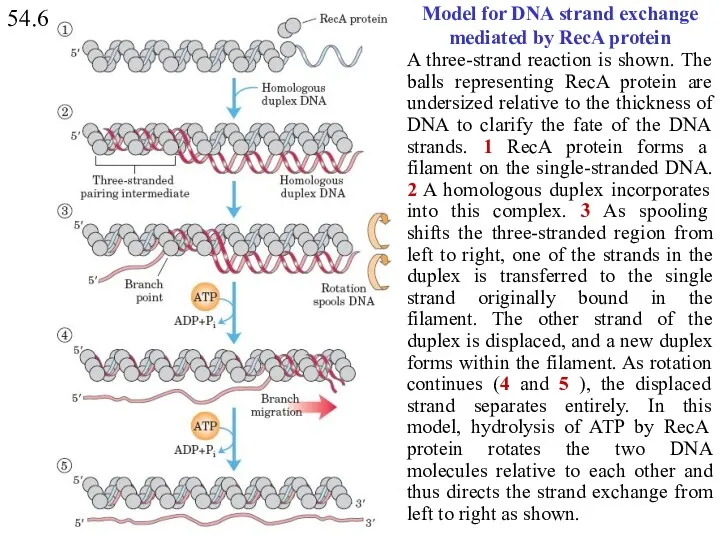
- 18. 54.6 Model for DNA strand exchange mediated by RecA protein A three-strand reaction is shown. The
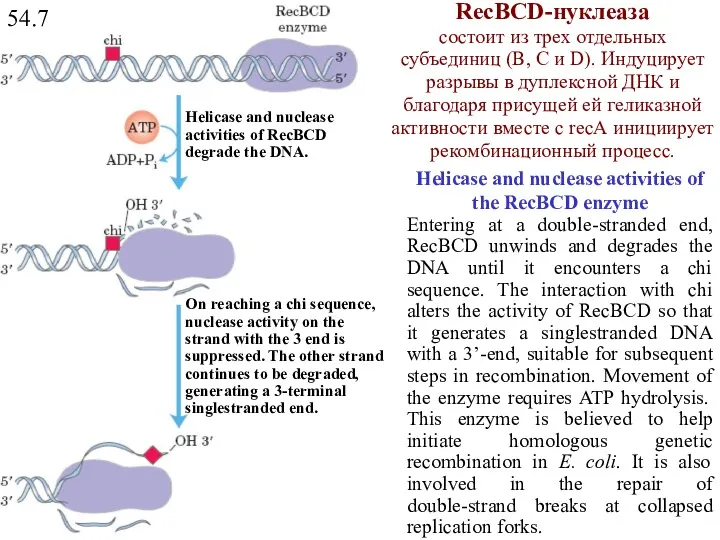
- 19. 54.7 Helicase and nuclease activities of the RecBCD enzyme Entering at a double-stranded end, RecBCD unwinds
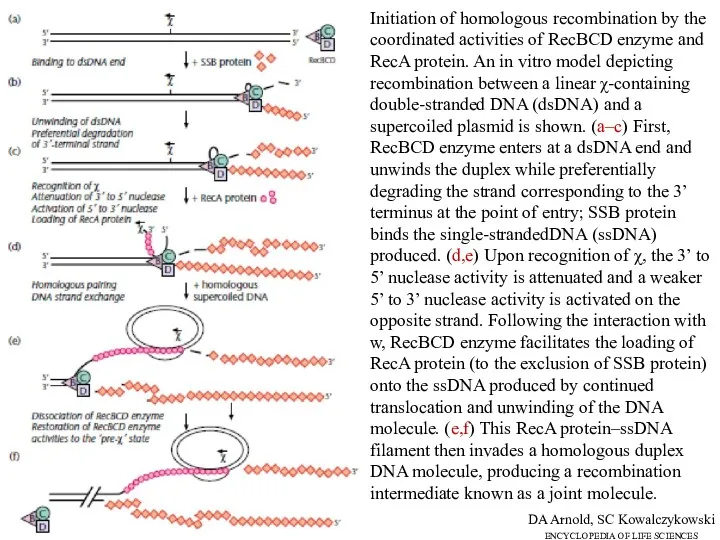
- 20. Initiation of homologous recombination by the coordinated activities of RecBCD enzyme and RecA protein. An in
- 21. The sequence of the Chi site is unique to each group of closely related organisms; in
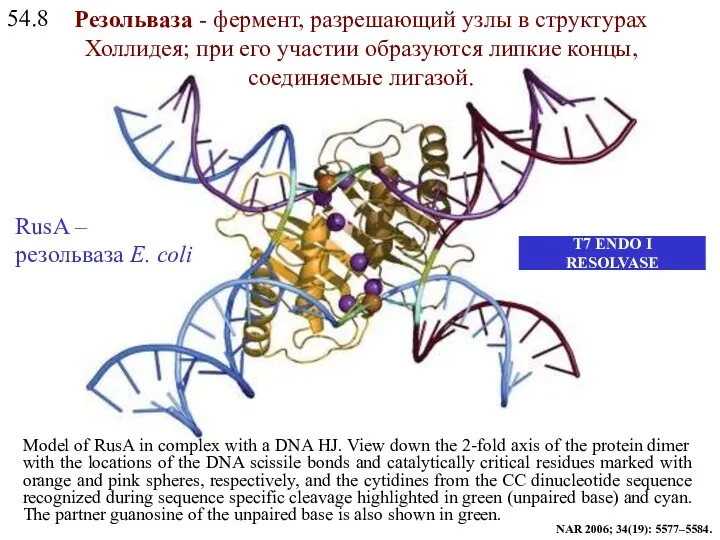
- 22. 54.8 T7 ENDO I RESOLVASE Model of RusA in complex with a DNA HJ. View down
- 24. Скачать презентацию



 Дикорослі та культурні рослини
Дикорослі та культурні рослини Мышцы головы человека, их строение и функции
Мышцы головы человека, их строение и функции Составление формул и диаграмм цветков
Составление формул и диаграмм цветков Птицы. Отряд Совообразные
Птицы. Отряд Совообразные Вред курения.
Вред курения. Тип Членистоногие. Подтип Хелицеровые. Эволюция хелицеровых
Тип Членистоногие. Подтип Хелицеровые. Эволюция хелицеровых Презентация к уроку биологии 7 класс
Презентация к уроку биологии 7 класс разработка урока в 5 классе по теме: Грибы
разработка урока в 5 классе по теме: Грибы Продукты пчеловодства и использование их в жизни человека
Продукты пчеловодства и использование их в жизни человека Общая биология. Половой диморфизм
Общая биология. Половой диморфизм Гладкие мышцы. Особенности строения, сократимости и возбудимости
Гладкие мышцы. Особенности строения, сократимости и возбудимости Водросли. Место водорослей среди растений
Водросли. Место водорослей среди растений Чудесное превращение. Появление комара
Чудесное превращение. Появление комара Анатомическое строение корня
Анатомическое строение корня Ферменттеге жалпы сипаттама
Ферменттеге жалпы сипаттама Класс млекопитающие, отряд китообразные
Класс млекопитающие, отряд китообразные Вскрытие трупов животных
Вскрытие трупов животных Растениеводство. Пропашные культуры
Растениеводство. Пропашные культуры Ягоды
Ягоды Тыквенные овощи
Тыквенные овощи Пауки и паутина. Рисование паутины пауков
Пауки и паутина. Рисование паутины пауков Царство животные
Царство животные Як спілкуються тварини. 7 клас
Як спілкуються тварини. 7 клас Эволюционная теория. Вид, его критерии. Популяция как структурная единица вида и эволюции
Эволюционная теория. Вид, его критерии. Популяция как структурная единица вида и эволюции Грудная клетка. Лекция № 10
Грудная клетка. Лекция № 10 Класс земноводные (амфибии)
Класс земноводные (амфибии) Червона книга України
Червона книга України Дүниесі: Жануарлар
Дүниесі: Жануарлар