Содержание
- 2. Научные программы, реализующие метод молекулярной динамики (только примеры, а не полный список) Коммерческие коды: Materials Explorer
- 3. Программа XMD Автор: Джон Рифкин (John Rifkin), Университет штата Коннектикут (США) http://xmd.sourceforge.net/about.html Используемые потенциалы: метод погруженного
- 4. Синтаксис использования Командное окно Windows (можно использовать FAR manager) > xmd xmd.in xmd.in – файл, содержащий
- 5. Ввод основных параметров моделирования MASS 58.7 (масса атомов в а.е.м.) DTIME 5.e-15 (шаг времени в сек)
- 6. Ввод начальных координат и расчетной ячейки READ coord.in # Файл coord.in содержит следующую информацию: POSITION 3
- 7. Таблица потенциалов МПА eunit eV potential set eam 1 potential embed 1 500 0.000000 0.250000 0.000000000000000E+00
- 8. Параметры, определяющие условия моделирования ITEMP temp # задание исходной температуры (генерация распределения скоростей, соответствующего температуре temp)
- 9. Команды проведения МД расчетов 1. QUENCH nstep [nquench=1,2] # Релаксация (минимизация энергии) при Т=0 К. Осуществляется
- 10. Использование команд цикла repeat K …… end # Используется для K- кратного повторения всех команд, заключенных
- 11. Команды вывода BSAVE nskip file # сохранение размеров расчетной ячейки ESAVE nskip file # сохранение средней
- 13. Скачать презентацию
 Шкала електромагнітних випромінювань. (11 клас)
Шкала електромагнітних випромінювань. (11 клас) Механическая работа. Физика 7 класс
Механическая работа. Физика 7 класс Кинематическая схема станка СТД-120 м
Кинематическая схема станка СТД-120 м Решение задач по теме Электромагнитное поле
Решение задач по теме Электромагнитное поле Механизмнің құрылымдығы және кинематикалық жұптарды жіктеу
Механизмнің құрылымдығы және кинематикалық жұптарды жіктеу Температураны өлшеу. Термоэлектрлі термометрлер
Температураны өлшеу. Термоэлектрлі термометрлер Медиаобразование, как средство активизации познавательной деятельности учащихся
Медиаобразование, как средство активизации познавательной деятельности учащихся Работа и мощность электрического тока
Работа и мощность электрического тока Ультразвук к в химической технологии. Лекция 5
Ультразвук к в химической технологии. Лекция 5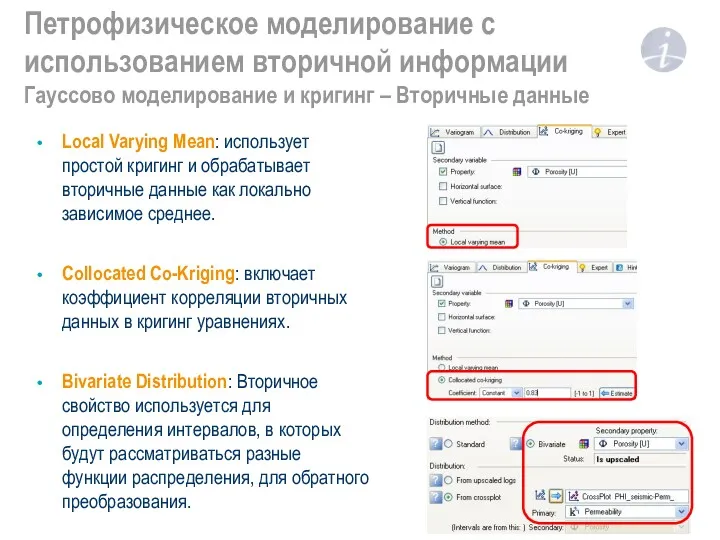 Петрофизическое моделирование с использованием вторичной информации. Гауссово моделирование и кригинг – Вторичные данные
Петрофизическое моделирование с использованием вторичной информации. Гауссово моделирование и кригинг – Вторичные данные Электрический ток в полупроводниках
Электрический ток в полупроводниках Свободные и вынужденные электромагнитные колебания. Колебательный контур. Превращение энергии при электромагнитных колебаниях
Свободные и вынужденные электромагнитные колебания. Колебательный контур. Превращение энергии при электромагнитных колебаниях Использование информационно-коммуникационных технологий в процессе обучения физике как средство повышения качества знаний
Использование информационно-коммуникационных технологий в процессе обучения физике как средство повышения качества знаний Energia Solar Fotovoltaica e Indústria de Rochas Ornamentais
Energia Solar Fotovoltaica e Indústria de Rochas Ornamentais Геометрическая оптика
Геометрическая оптика Электрооборудование автомобилей. Системы автоматического управления тормозами. (Урок 14)
Электрооборудование автомобилей. Системы автоматического управления тормозами. (Урок 14) Расчетный напор НС-2 при расположении башни в начале сети
Расчетный напор НС-2 при расположении башни в начале сети Теплообменные аппараты
Теплообменные аппараты Изопроцессы в газах, решение графических задач
Изопроцессы в газах, решение графических задач Элементы квантовой физики. Лекция №6
Элементы квантовой физики. Лекция №6 Механическая работа
Механическая работа Расчет статически неопределимых систем по допускаемым нагрузкам
Расчет статически неопределимых систем по допускаемым нагрузкам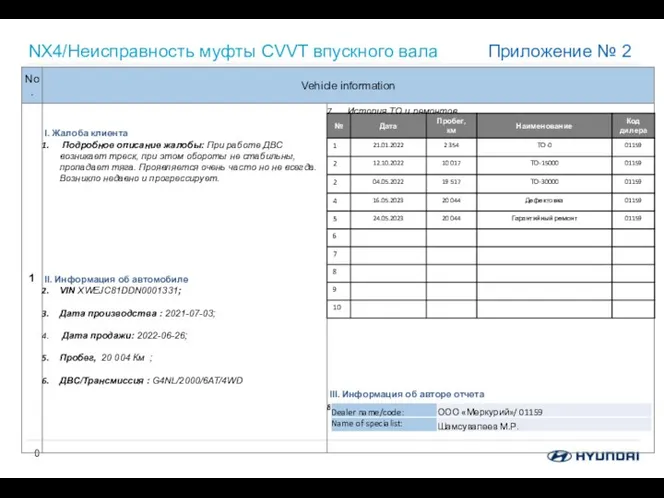 NX4. Неисправность муфты CVVT впускного вала (приложение 2)
NX4. Неисправность муфты CVVT впускного вала (приложение 2) Домашняя лабораторная работа. Определение плотности куска хозяйственного мыла
Домашняя лабораторная работа. Определение плотности куска хозяйственного мыла Конические зубчатые передачи. Расчет конических зубчатых передач
Конические зубчатые передачи. Расчет конических зубчатых передач Синтез нанокомпозитных материалов на основе субоксида кремния и наноструктур благородных металлов
Синтез нанокомпозитных материалов на основе субоксида кремния и наноструктур благородных металлов Рубка металла
Рубка металла Коэффициент полезного действия (КПД) механизма
Коэффициент полезного действия (КПД) механизма