Содержание
- 2. Форматы файлов, используемых в биоинформатике FASTA >roa1_drome Rea guano receptor type III >> 0.1 MVNSNQNQNGNSNGHDDDFPQDSITEPEHMRKLFIGGLDYRTTDENLKAHEKWGNIVDVVVMKDPRTKRSRGFGFITYSHSSMIDEAQKSRPHKIDGRVEPKRAVPRQDIDSPNAGATVKKLFVGALKDDHDEQSIRDYFQHFGNIVDNIVIDKETGKKRGFAFVEFDDYDPVDKVVLQKQHQLNGKMVDVKKALPKNDQQGGGGGRGGPGGRAGGNRGNMGGGNYGNQNGGGNWNNGGNNWGNNRGNDNWGNNSFGGGGGGGGGYGGGNNSWGNNNPWDNGNGGGNFGGGGNNWNGGNDFGGYQQNYGGGPQRGGGNFNNNRMQPYQGGGGFKAGGGNQGNYGNNQGFNNGGNNRRY >roa2_drome
- 3. ClustalW Очень известная и широко распространённая программа: UNIX, Internet, Windows. Выполняет MSA; может строить филогенетические деревья.
- 4. ClustalW ToTo To fasta @ list >IPNS_STRJU P18286 MPILMPSAEVPTIDISPLSGDDAKAKQRVAQEINKAARGSGFFYASNHGVDVQLLQDVVN EFHRNMSDQEKHDLAINAYNKDNPHVRNGYYKAIKGKKAVESFCYLNPSFSDDHPMIKSE TPMHEVNLWPDEEKHPRFRPFCEDYYRQLLRLSTVIMRGYALALGRREDFFDEALAEADT LSSVSLIRYPYLEEYPPVKTGADGTKLSFEDHLDVSMITVLYQTEVQNLQVETVDGWQDI PRSDEDFLVNCGTYMGHITHDYFPAPNHRVKFINAERLSLPFFLNAGHNSVIEPFVPEGA AGTVKNPTTSYGEYLQHGLRALIVKNGQT >IPNS_STRCL P10621
- 5. ClustalW CLUSTAL W (1.7) multiple sequence alignment IPNS_STRJU -MPILMPSAEVPTIDISPLSGDDAKAKQRVAQEINKAARGSGFFYASNHGVDVQLLQDVV IPNS_STRGR -MPIPMLPAHVPTIDISPLSGGDADDKKRVAQEINKACRESGFFYASHHGIDVQLLKDVV IPNS_FLASS ----MNRHADVPVIDISGLSGNDMDVKKDIAARIDRACRGSGFFYAANHGVDLAALQKFT IPNS_PENCH --MASTPKANVPKIDVSPLFGDNMEEKMKVARAIDAASRDTGFFYAVNHGVDVKRLSNKT IPNS_CEPAC
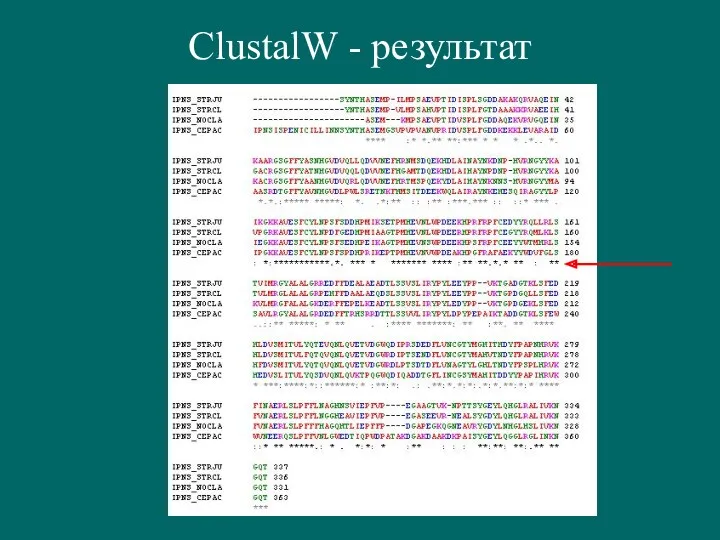
- 6. ClustalW - результат
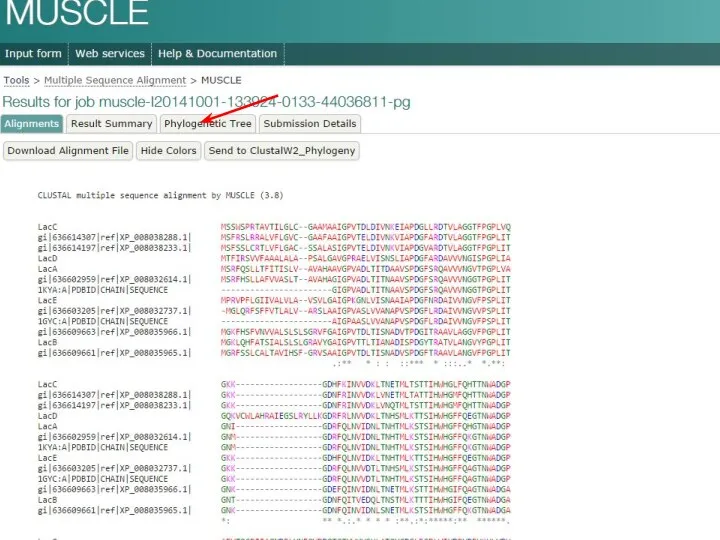
- 7. http://www.ebi.ac.uk/Tools/msa/muscle/
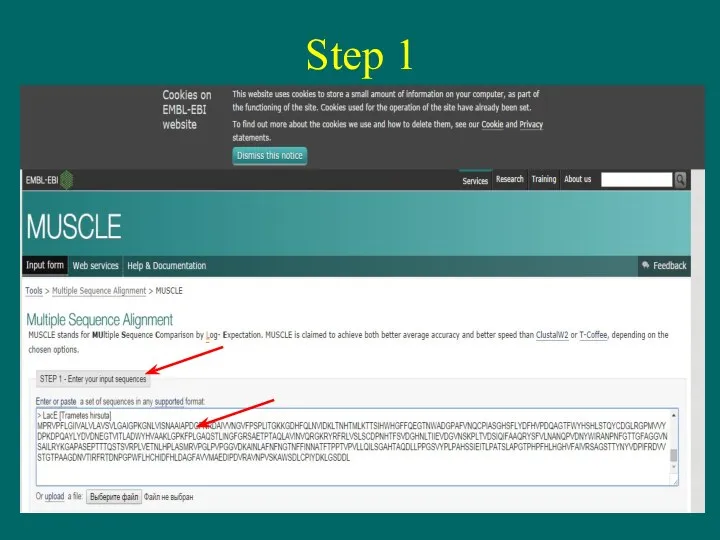
- 8. Step 1
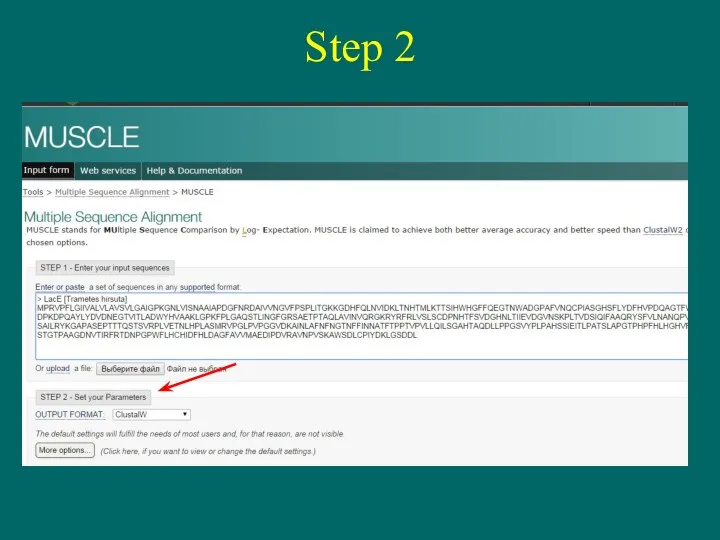
- 9. Step 2
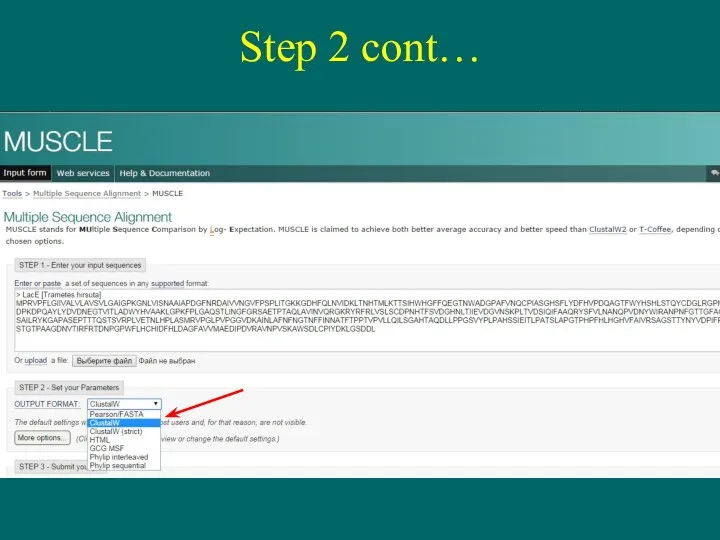
- 10. Step 2 cont…
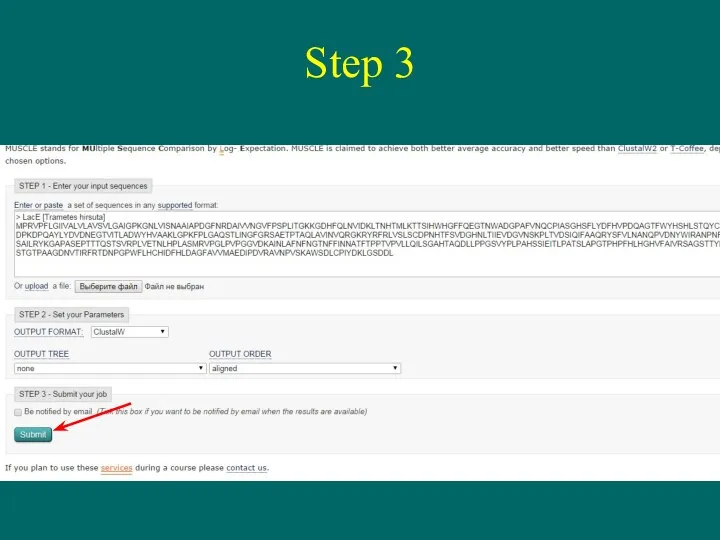
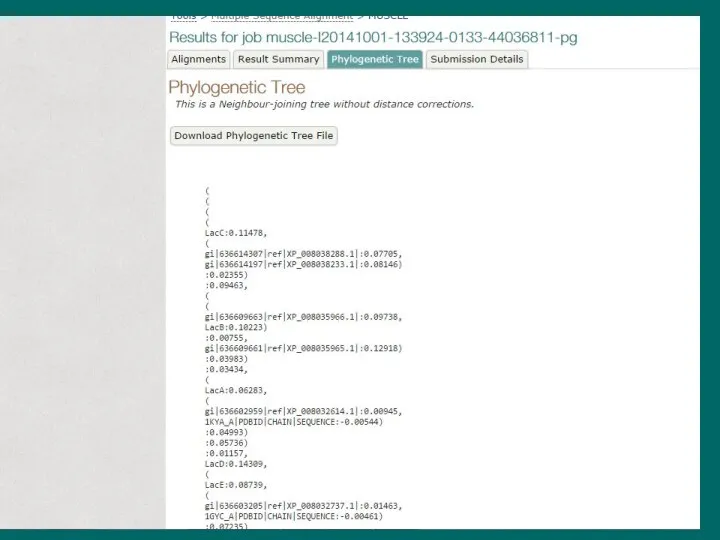
- 11. Step 3
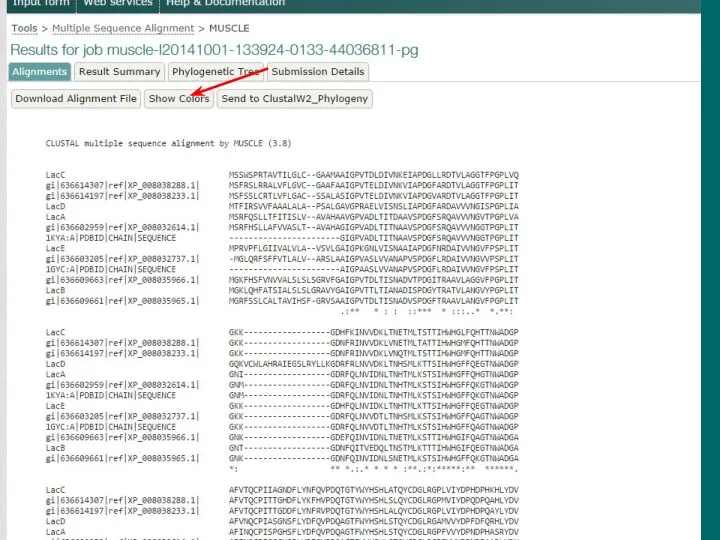
- 17. Статистика выравнивания
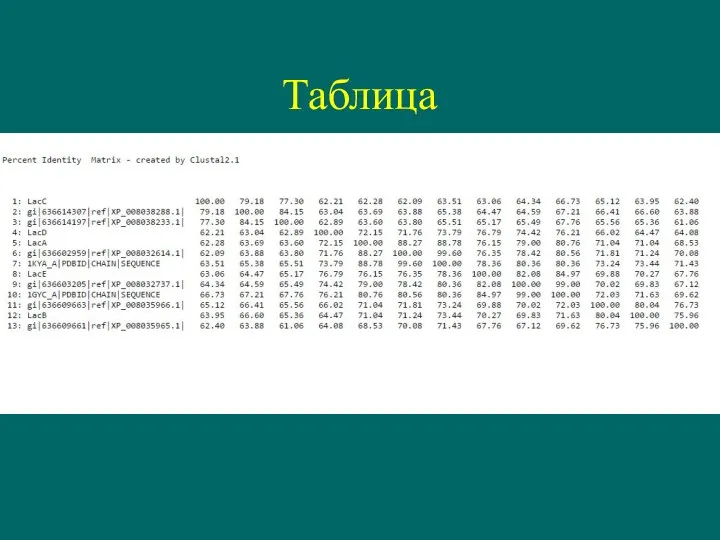
- 18. Таблица
- 19. Задание на дом Провести выравнивание последовательностей из файла (Muscle) Дать статистику выравнивания: описать, какие белки имеют
- 20. Поиск консервативных участков глазами и руками☺ «*» - строго одна и та же ак у всех
- 21. Система оценки - белки
- 22. Задание на дом Провести выравнивание последовательностей из файла (Muscle) Дать статистику выравнивания: Найти участки консервативные (глазами
- 23. Задание на дом Провести выравнивание последовательностей из файла с использованием других программ: T-coffee ClustalW ProbCons Сравнить
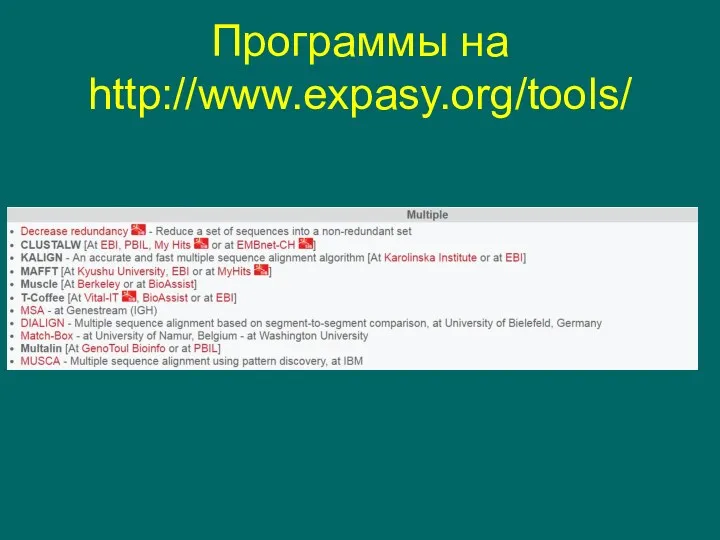
- 24. Программы на http://www.expasy.org/tools/
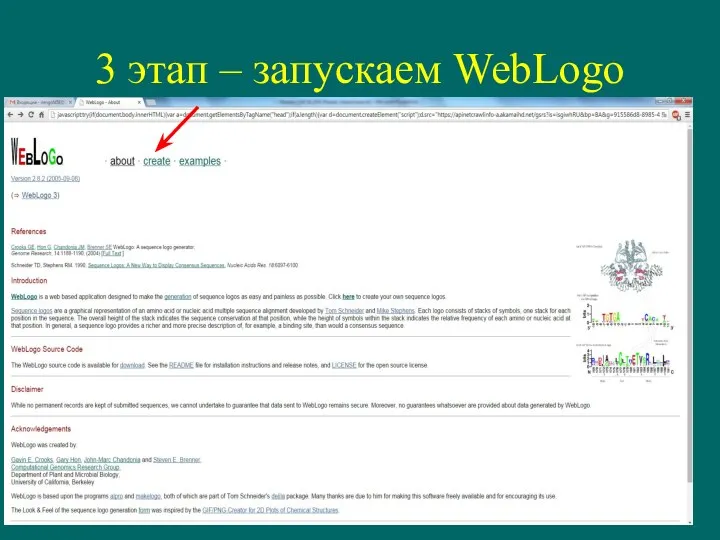
- 25. Построение Logo Weblogo – http://weblogo.berkeley.edu/
- 26. 1 этап – определяем консервативный участок
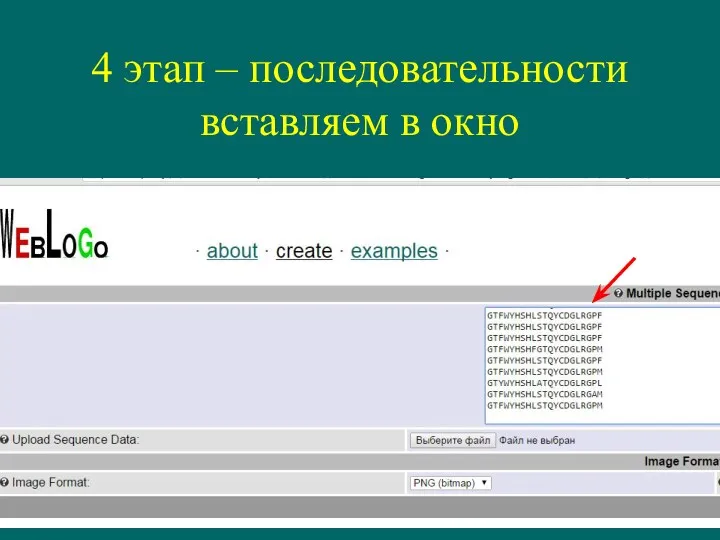
- 27. 2 этап – выбираем общее выравнивание этого участка GTFWYHSHLSTQYCDGLRGPL GTFWYHSHLSTQYCDGLRGPI GTFWYHSHLSTQYCDGLRGPF GTFWYHSHLSTQYCDGLRGPF GTFWYHSHLSTQYCDGLRGPF GTFWYHSHFGTQYCDGLRGPM GTFWYHSHLSTQYCDGLRGPF GTFWYHSHLSTQYCDGLRGPM
- 28. 3 этап – запускаем WebLogo
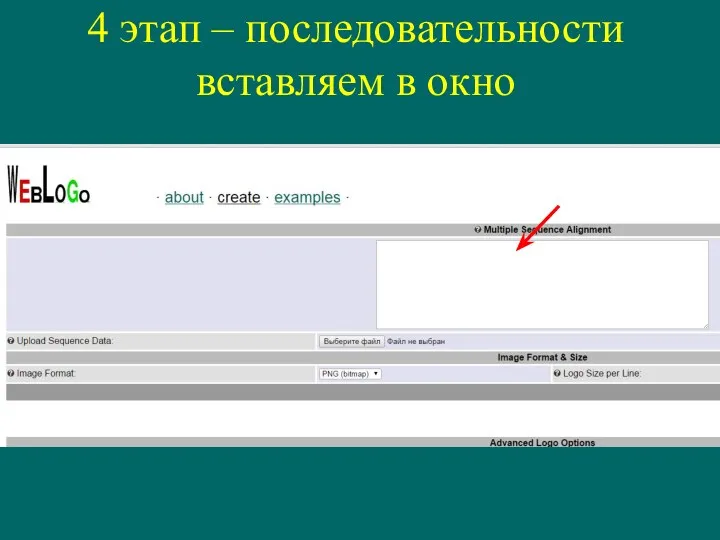
- 29. 4 этап – последовательности вставляем в окно
- 30. 4 этап – последовательности вставляем в окно
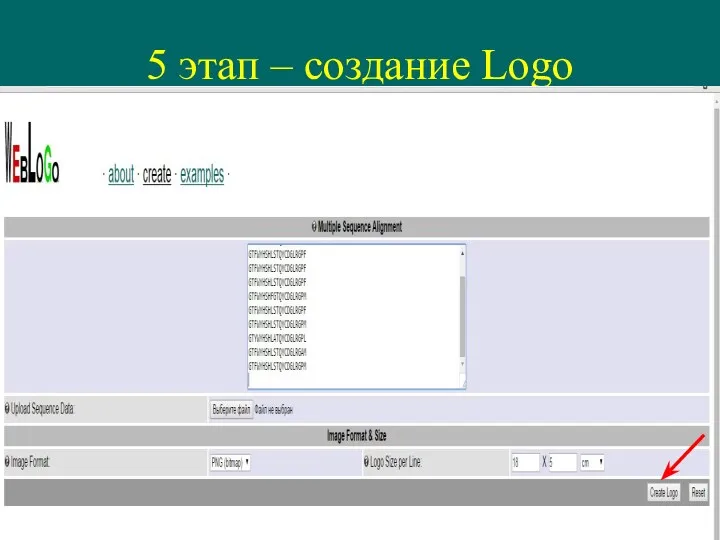
- 31. 5 этап – создание Logo
- 32. 6 этап – готовый Logo
- 33. Можно поиграть с настройками (при желании☺)
- 35. Скачать презентацию















 Karmarkar Algorithm
Karmarkar Algorithm ActionScript 3.0 негізінде қолданба құру әдістемесі
ActionScript 3.0 негізінде қолданба құру әдістемесі Решение задач на обработку массивов
Решение задач на обработку массивов Телекоммуникационные сети
Телекоммуникационные сети Компьютерная игра (или видео-игра)
Компьютерная игра (или видео-игра) Контейнеры в C++
Контейнеры в C++ Лекция №7 Мультимедиа технологии
Лекция №7 Мультимедиа технологии Орта мектептерде ұйымдастыру жұмыстарын жүргізуде деректер қорын пайдалану
Орта мектептерде ұйымдастыру жұмыстарын жүргізуде деректер қорын пайдалану Б. Бернстайн и его теория языковых кодов
Б. Бернстайн и его теория языковых кодов Creating Functions and Debugging Subprograms
Creating Functions and Debugging Subprograms Типы алгоритмических структур с примерами
Типы алгоритмических структур с примерами Проблемы и современные методы управления мехатронными модулями и системами
Проблемы и современные методы управления мехатронными модулями и системами Хранение, отбор и сортировка информации в базах данных. Лекция 19
Хранение, отбор и сортировка информации в базах данных. Лекция 19 Радио России
Радио России Базовые понятия языка Си
Базовые понятия языка Си Пиши код правильно
Пиши код правильно Создание буклетов в приложении MS Publisher
Создание буклетов в приложении MS Publisher Теоретический лицей. Образец сайта
Теоретический лицей. Образец сайта Программирование на языке высокого уровня. (Лекции 1, 2)
Программирование на языке высокого уровня. (Лекции 1, 2) Сравнительный анализ UML-диаграмм
Сравнительный анализ UML-диаграмм Логические функции MS Excel
Логические функции MS Excel Кодирование звуковой информации
Кодирование звуковой информации Обмін даними між графічним редактором, тестовим і табличним процесором
Обмін даними між графічним редактором, тестовим і табличним процесором Технология XPath
Технология XPath 10 советов, которые помогут вам использовать PowerPoint на пользу
10 советов, которые помогут вам использовать PowerPoint на пользу Нейронные сети
Нейронные сети Система автоматизации библиотек ИРБИС
Система автоматизации библиотек ИРБИС Программирование на Java
Программирование на Java