Содержание
- 2. Механизмы репарации ДНК
- 3. Объекты для изучения репарации Escherichia coli Saccharomyces cerevisiae Caenorhabditis elegans Arabidopsis Thaliana Мыши, крысы Клеточные культуры
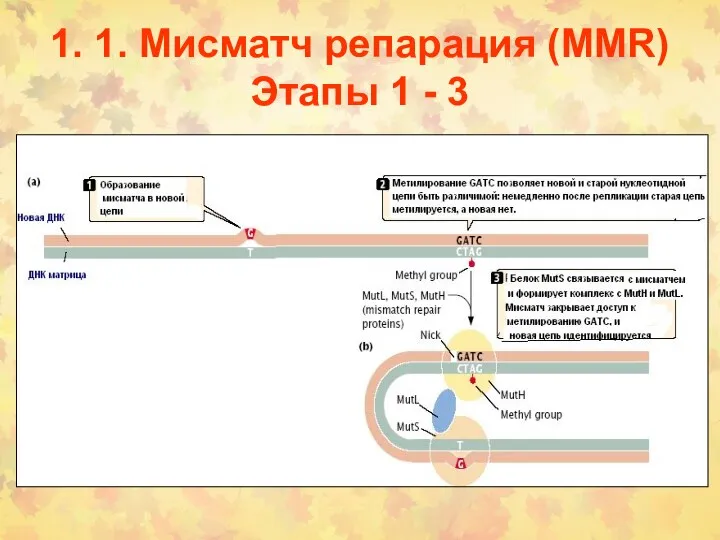
- 4. 1. 1. Мисматч репарация (MMR) Этапы 1 - 3
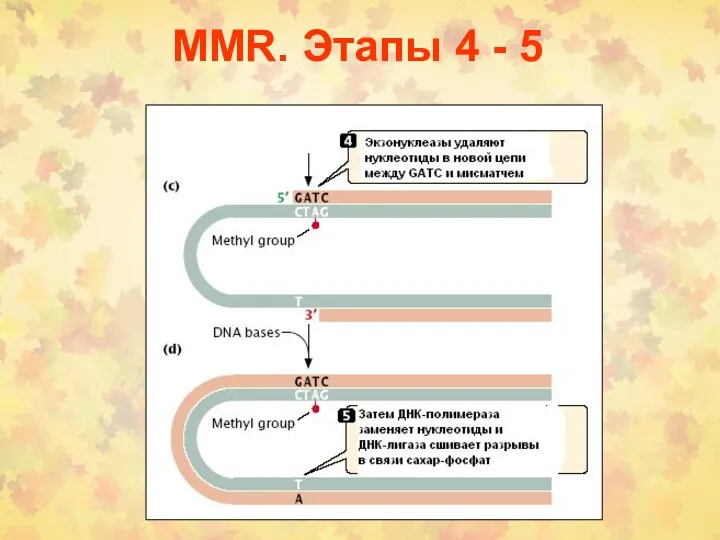
- 5. MMR. Этапы 4 - 5
- 6. А. Репарация мисматчей у бактерий VSP- very short patch repair Short patch repair Long patch repair
- 7. VSP- very short patch repair – 1 В основном удаляется Т из мисматчей G/T и C/T.
- 8. VSP- very short patch repair – 2 MutY заменяет А из мисматчей C/A и G/A. Это
- 9. MMR млекопитающих 9 генов: MLH1, MLH3, PMS1-2, MSH2-6 MSH – гомолог MutS MLH – гомолог MutL
- 10. MMR человека На примере болезни HNPCC (heredity non-polyposis colorectal cancer) в 1993-1994 гг. У человека найдено
- 11. MMR человека На примере болезни HNPCC (heredity non-polyposis colorectal cancer) в 1993-1994 гг. Пациенты с HNPCC
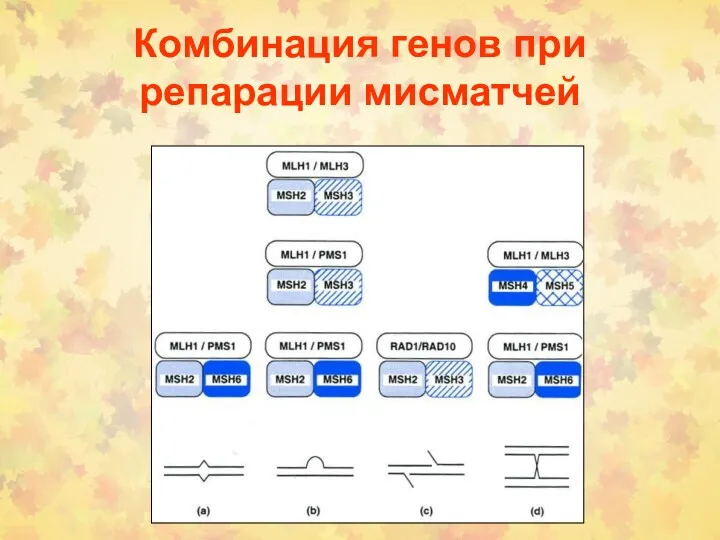
- 12. Комбинация генов при репарации мисматчей
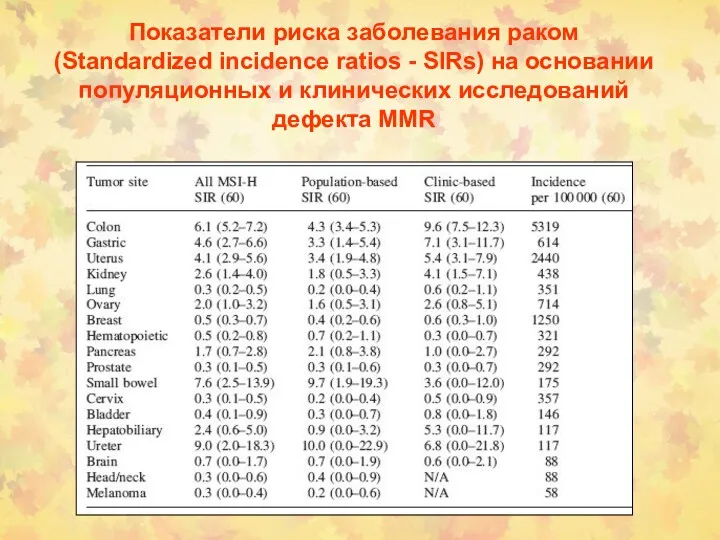
- 13. Показатели риска заболевания раком (Standardized incidence ratios - SIRs) на основании популяционных и клинических исследований дефекта
- 14. Механизмы, осуществляющие вклад в специфичность клеточных типов, чувствительных к дефициту MMR
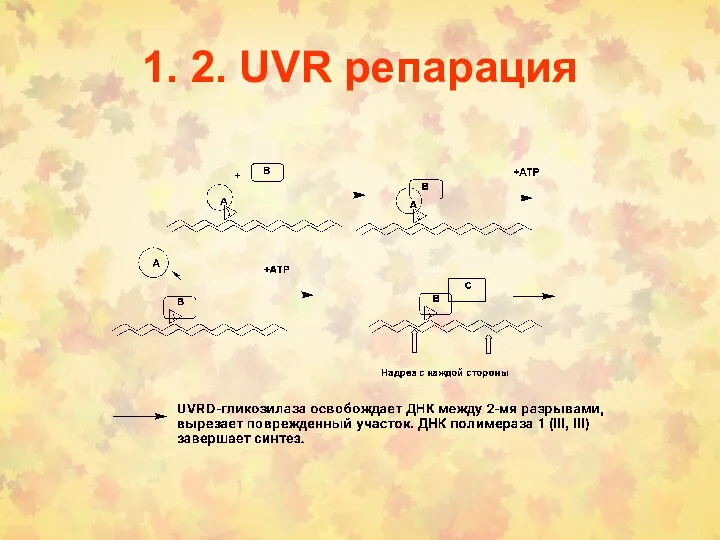
- 15. 1. 2. UVR репарация
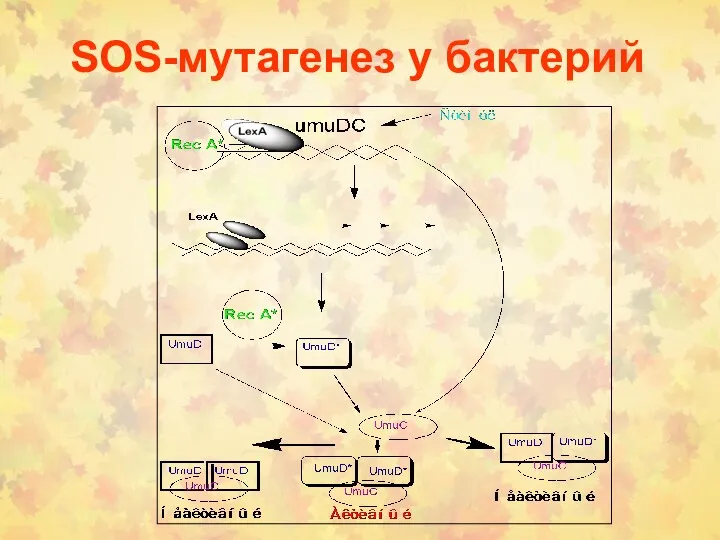
- 16. SOS-мутагенез у бактерий
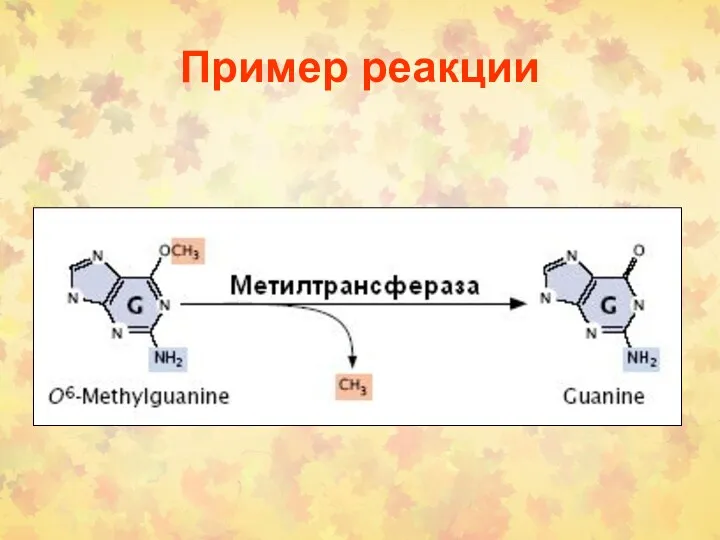
- 17. 2. Прямая репарация Репарируются О6-метилгуанин и О4-метилгуанин ферментом МТаза (MGMT). У Е. coli 2 фермента (гены
- 18. Пример реакции
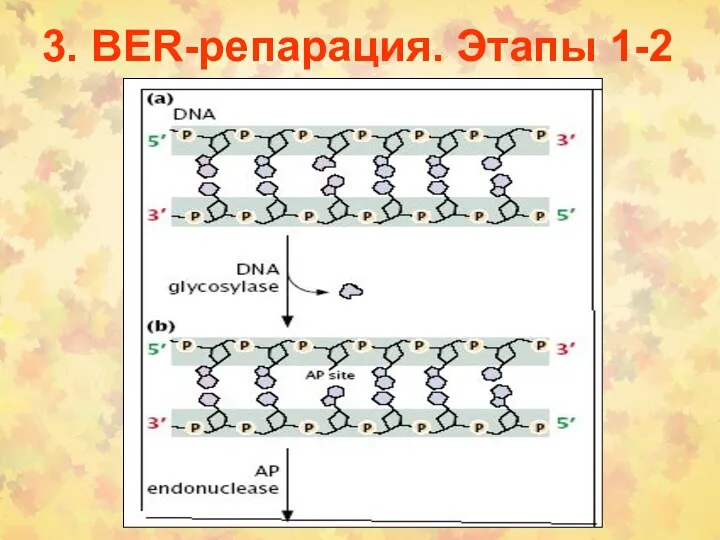
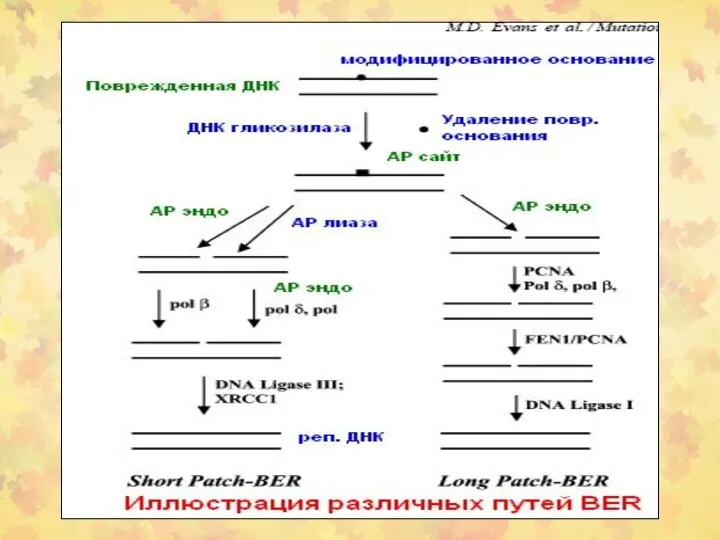
- 19. 3. BER-репарация. Этапы 1-2
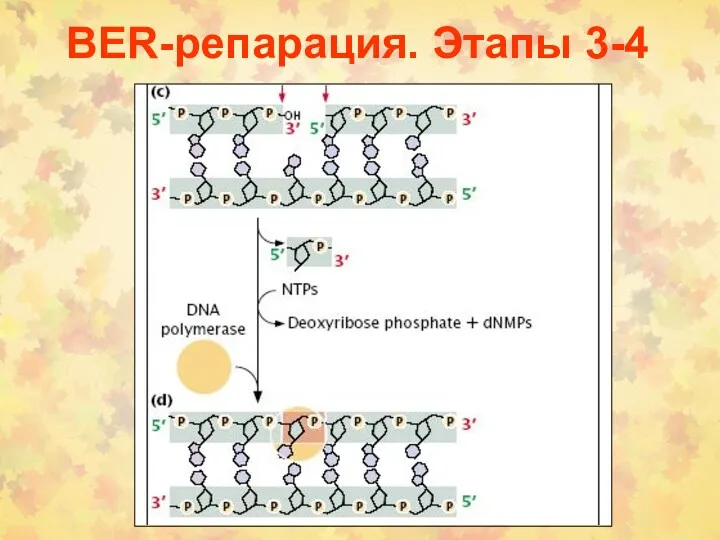
- 20. BER-репарация. Этапы 3-4
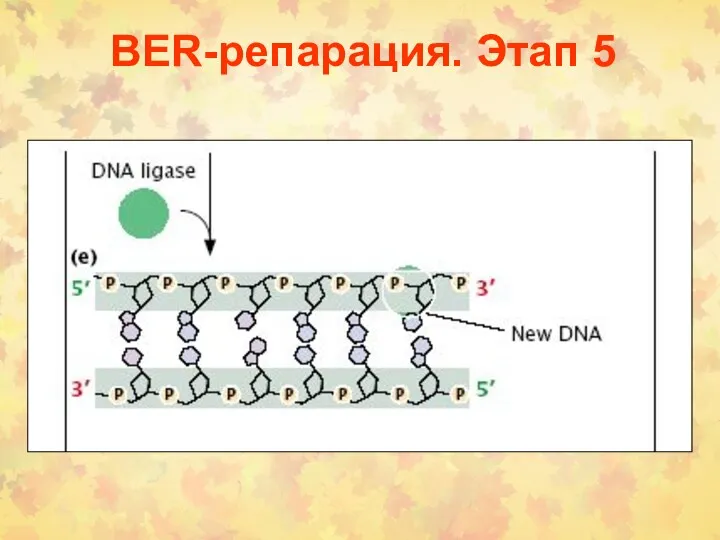
- 21. BER-репарация. Этап 5
- 23. 4. NER-репарация 1. TCR – transcription coupled repair (преимущественная репарация траснкрибируемых цепей гена) 2. GGR –
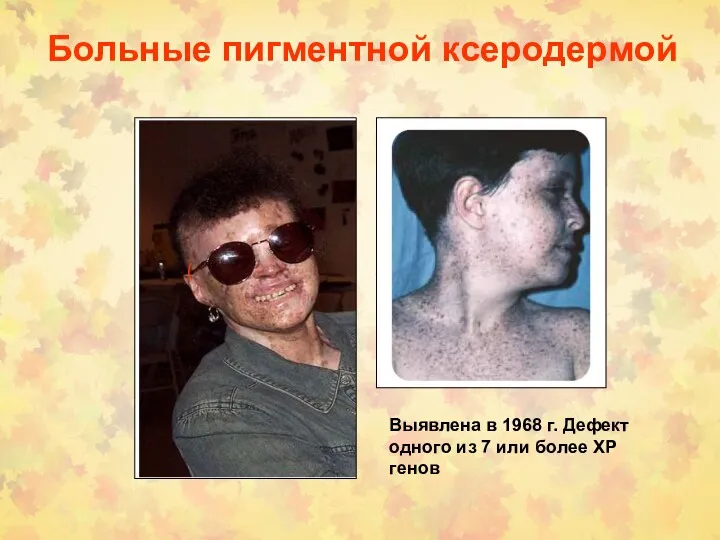
- 24. Больные пигментной ксеродермой ( Выявлена в 1968 г. Дефект одного из 7 или более XP генов
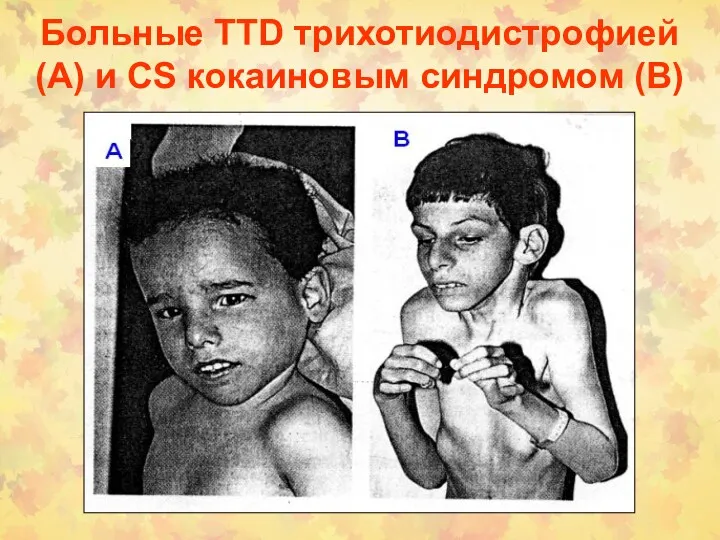
- 25. Больные TTD трихотиодистрофией (А) и CS кокаиновым синдромом (В)
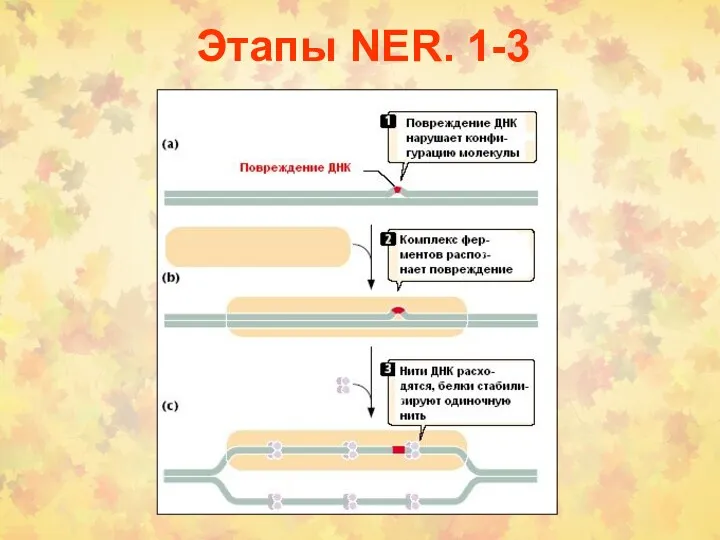
- 26. Этапы NER. 1-3
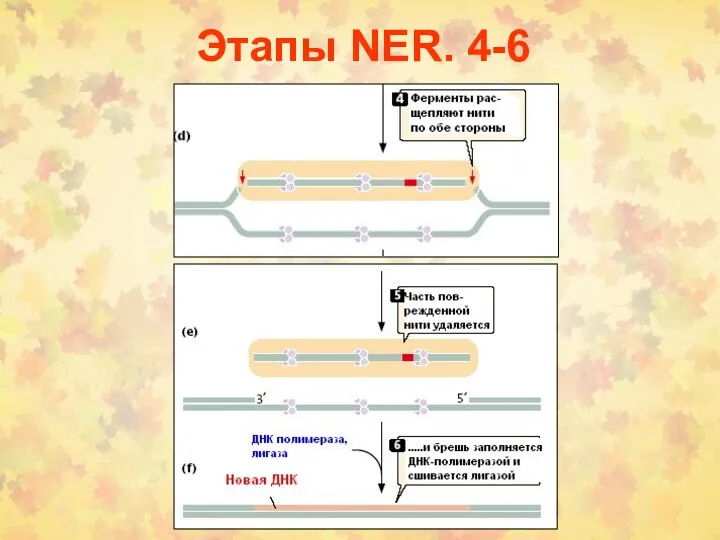
- 27. Этапы NER. 4-6
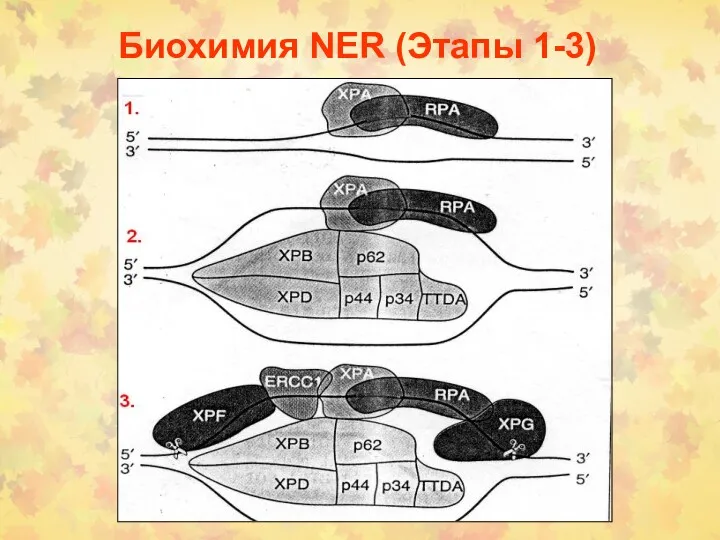
- 28. Биохимия NER (Этапы 1-3)
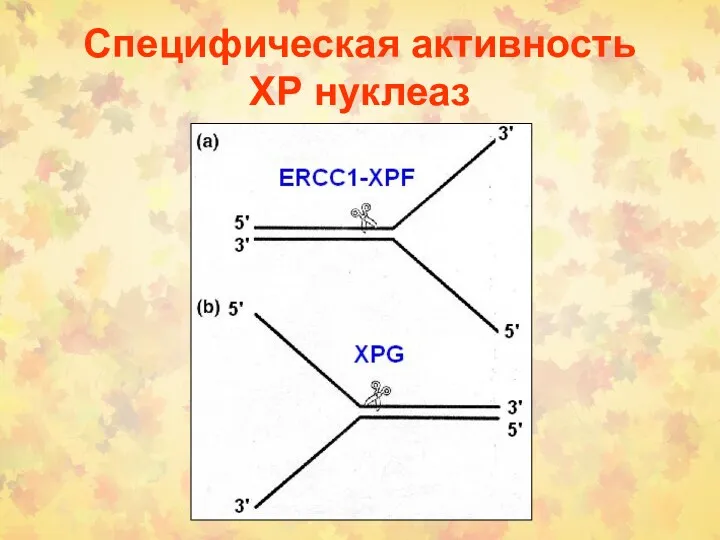
- 29. Специфическая активность ХР нуклеаз
- 30. Биохимия NER (Этапы 4-5)
- 31. Повреждение ХР при болезнях ХР – мутации в генах XP A-D,F,G TTD – серо-дефицитные хрупкие волосы,
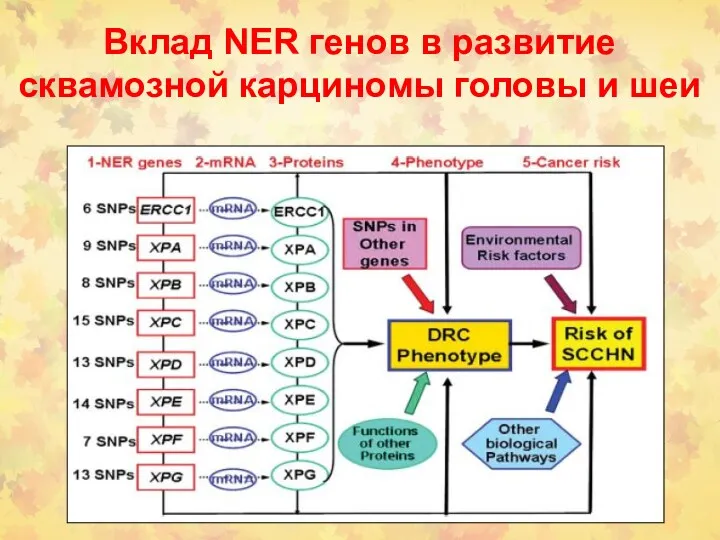
- 32. Вклад NER генов в развитие сквамозной карциномы головы и шеи
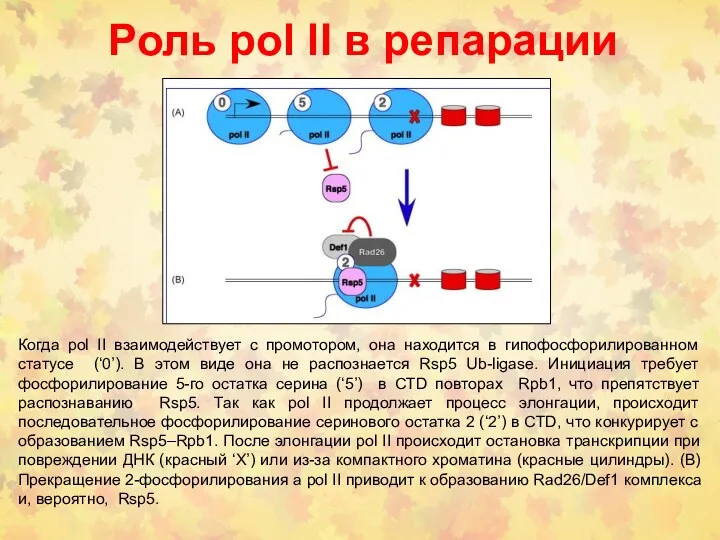
- 33. Роль pol lI в репарации Когда pol II взаимодействует с промотором, она находится в гипофосфорилированном статусе
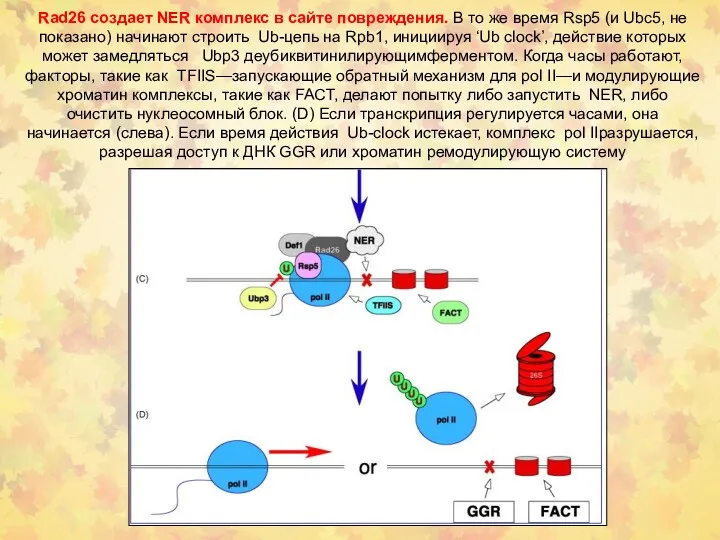
- 34. Rad26 создает NER комплекс в сайте повреждения. В то же время Rsp5 (и Ubc5, не показано)
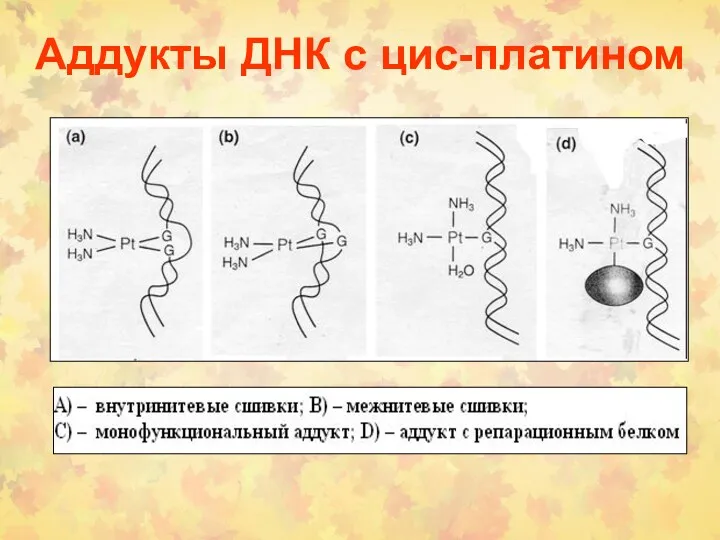
- 35. Аддукты ДНК с цис-платином
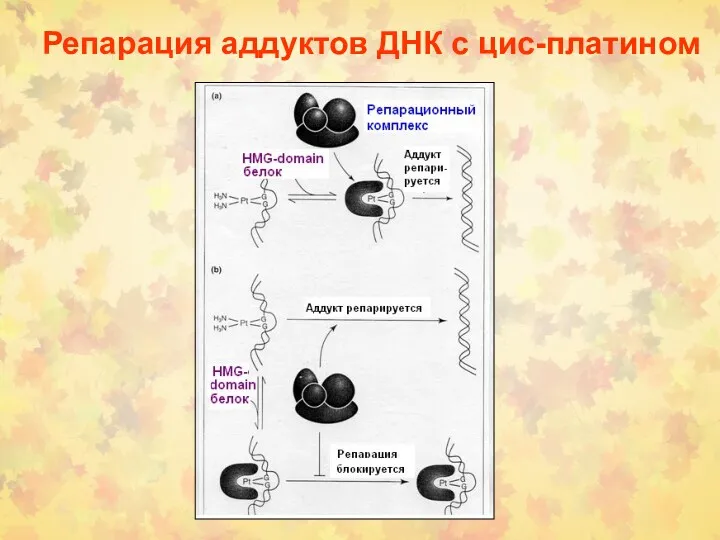
- 36. Репарация аддуктов ДНК с цис-платином
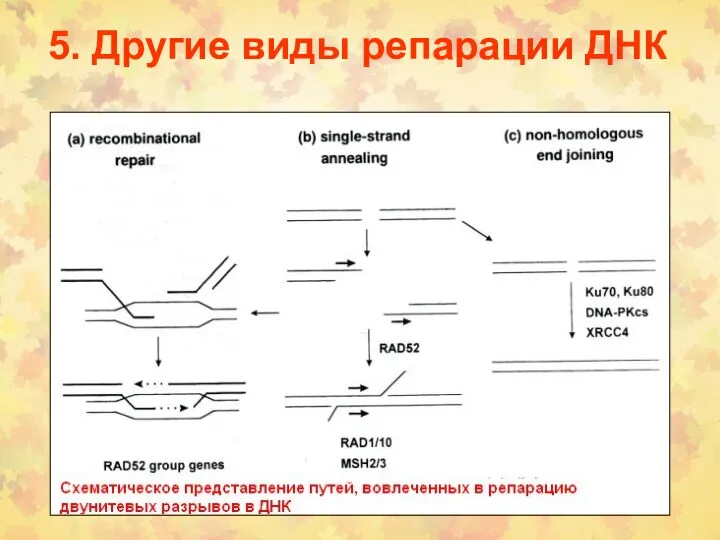
- 37. 5. Другие виды репарации ДНК
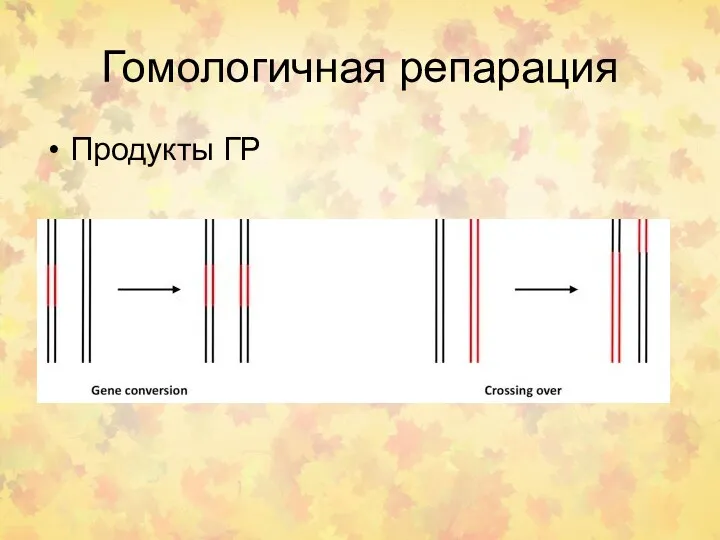
- 38. Гомологичная репарация Продукты ГР
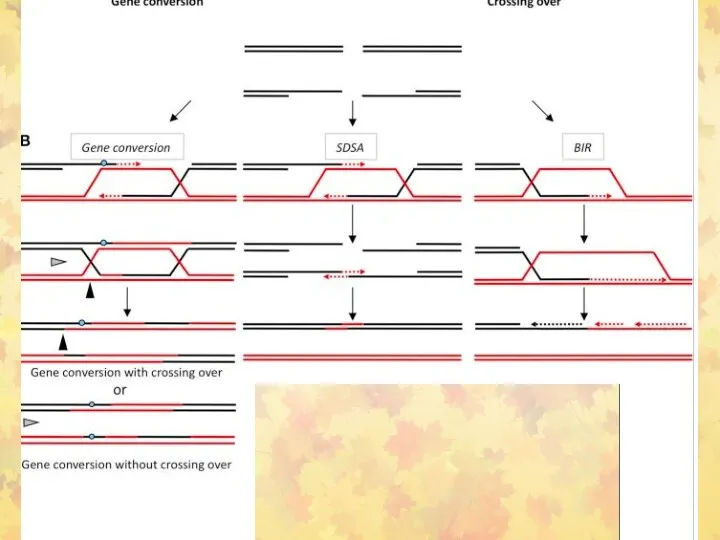
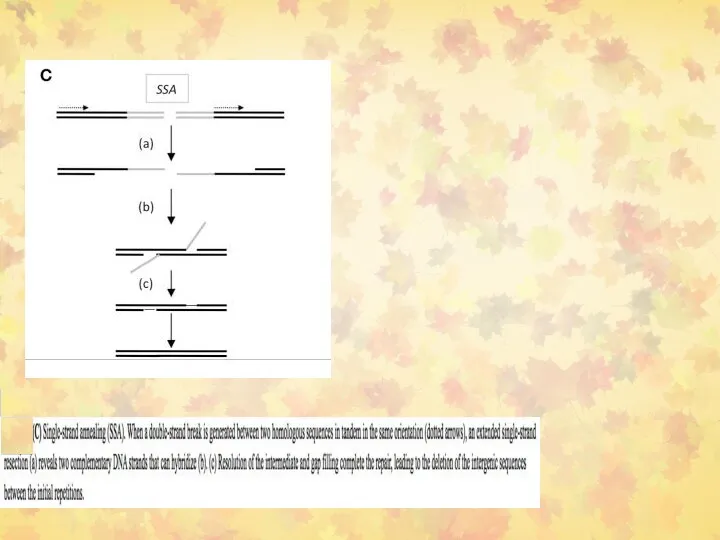
- 40. The double-strand break repair models through HR. Left panel: Gene conversion. After resection, the single-stranded 3′-tail
- 42. Белки ATM ATM (="ataxia telangiectasia mutated") получила название от болезни, пациенты, среди прочего, имеют высокий риск
- 43. Семейство АТМ белков
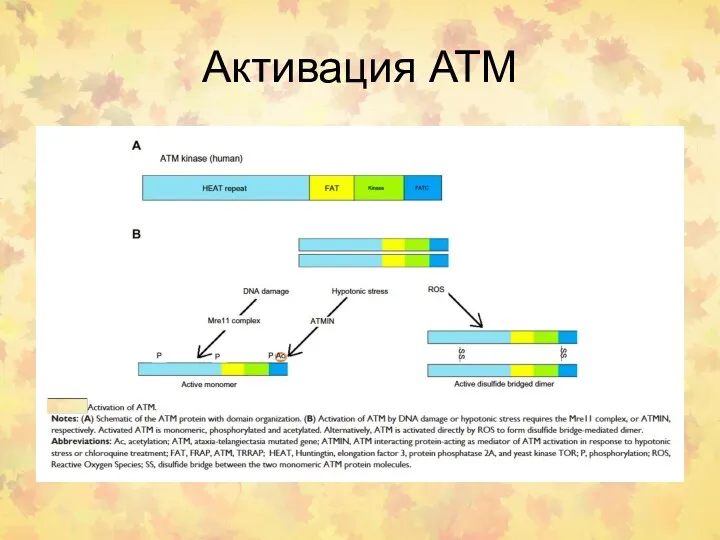
- 44. Активация АТМ
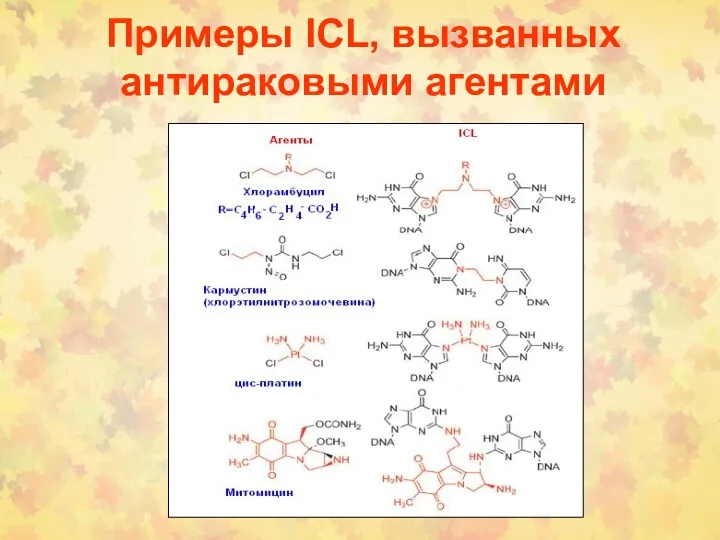
- 45. Примеры ICL, вызванных антираковыми агентами
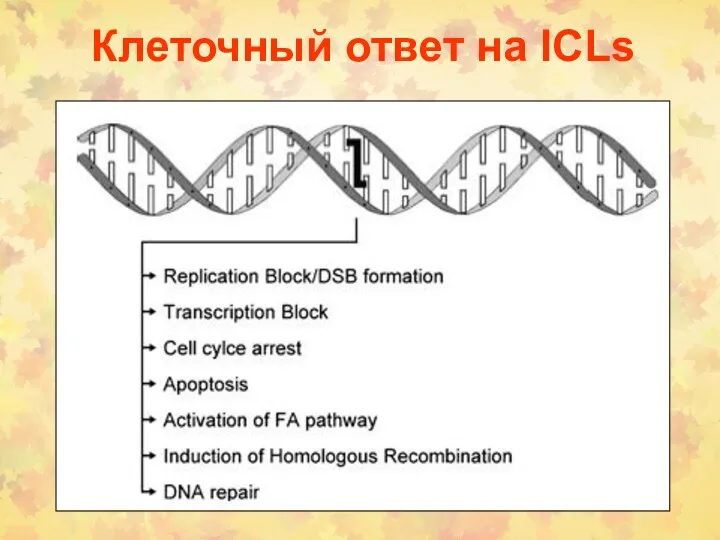
- 46. Клеточный ответ на ICLs
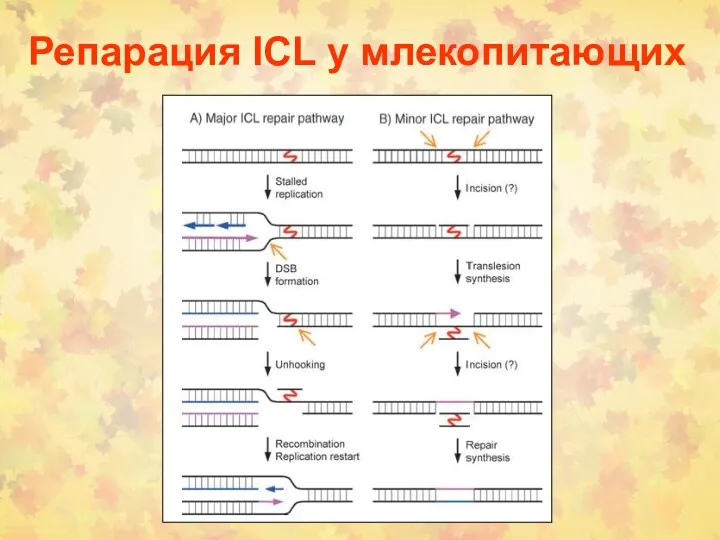
- 47. Репарация ICL у млекопитающих
- 48. Fanconi Anemia (FA)путь репарации У пациентов с FA повреждено, по крайней мере, 13 генов: FANCA, B,
- 49. Сравнение FA генов у человека, Drosophila, Dictyostelium and C. elegans
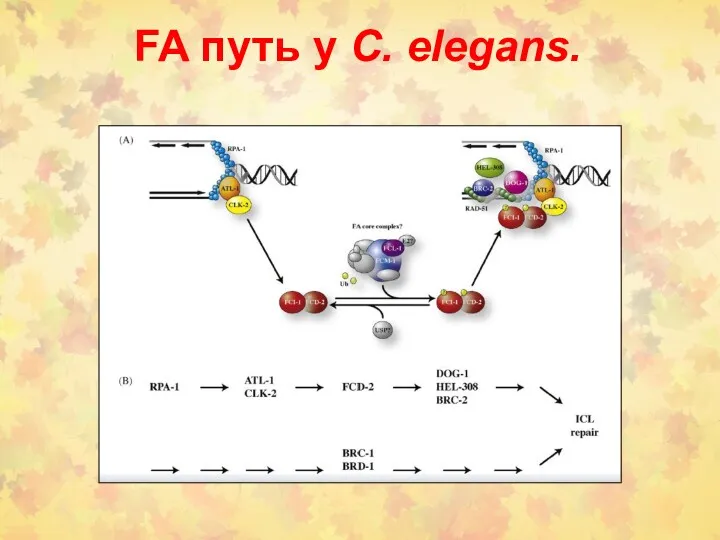
- 50. FA путь у C. elegans.
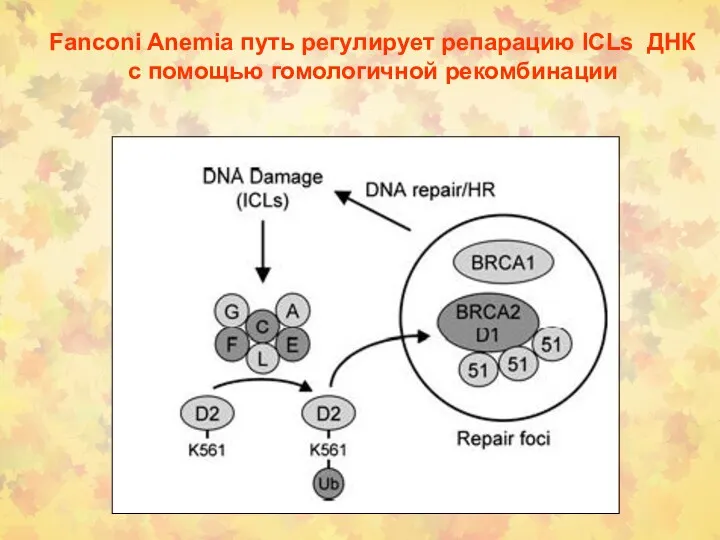
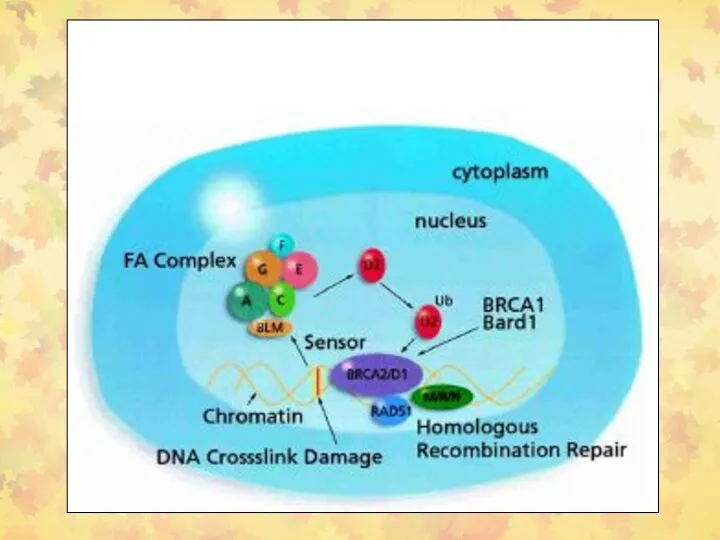
- 51. Fanconi Anemia путь регулирует репарацию ICLs ДНК с помощью гомологичной рекомбинации
- 53. BRCA1
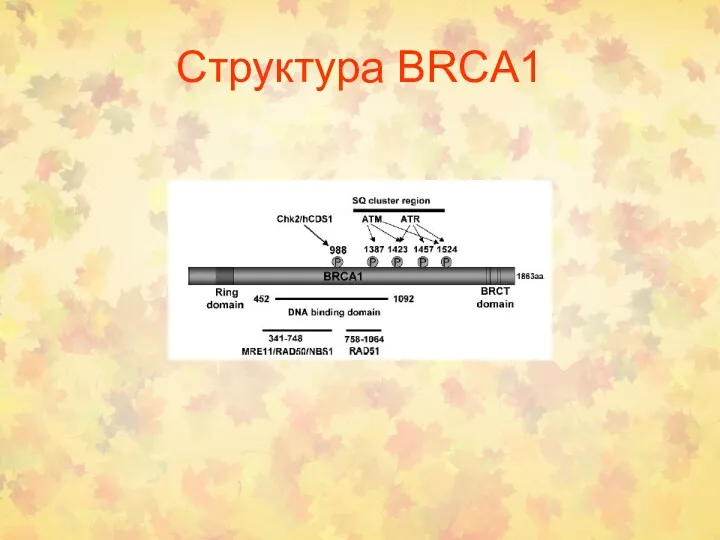
- 54. Структура BRCA1
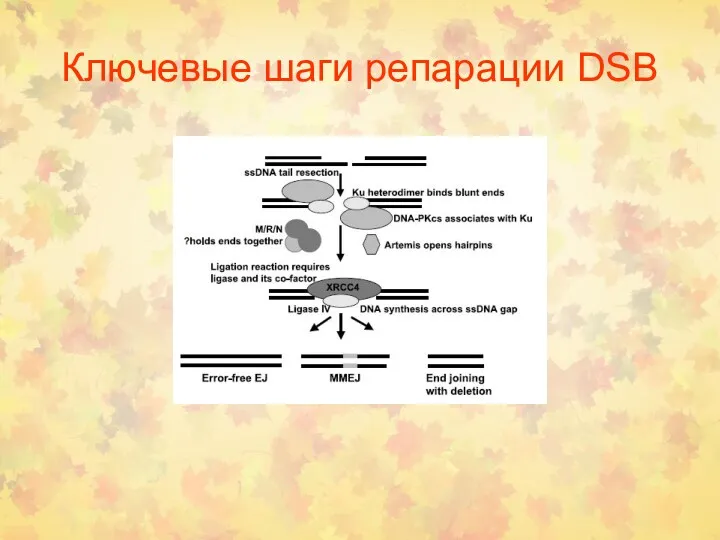
- 55. Ключевые шаги репарации DSB
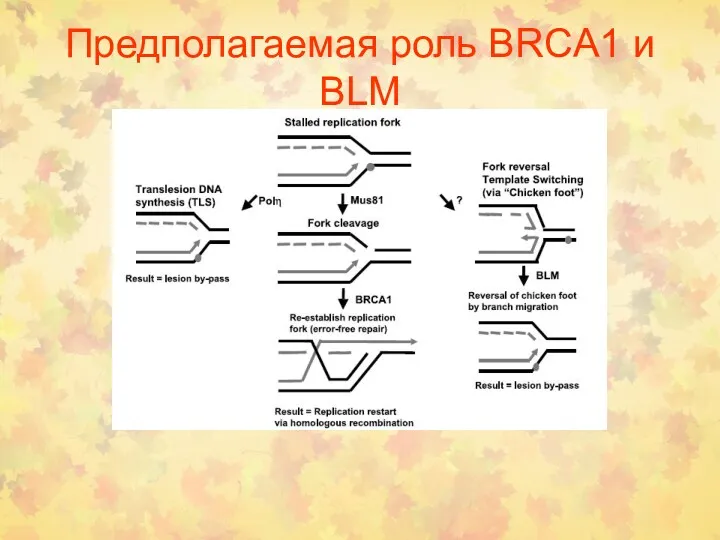
- 56. Предполагаемая роль BRCA1 и BLM
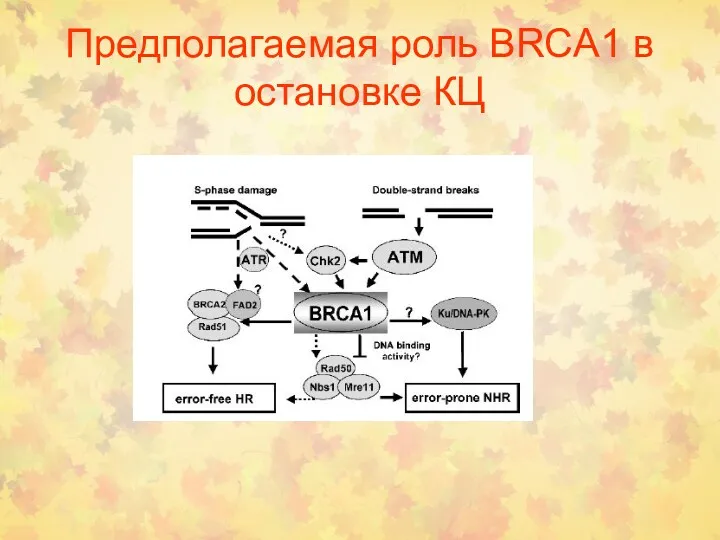
- 57. Предполагаемая роль BRCA1 в остановке КЦ
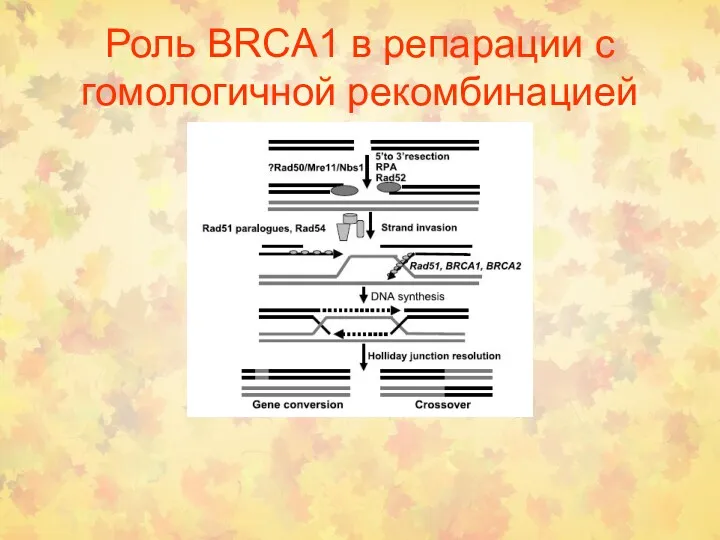
- 58. Роль BRCA1 в репарации с гомологичной рекомбинацией
- 59. SUMO (small ubiquitin-related modifier) конъюгация Несколько SUMO E3 лигаз выявлено: SP-RING (secretory protein with a RING
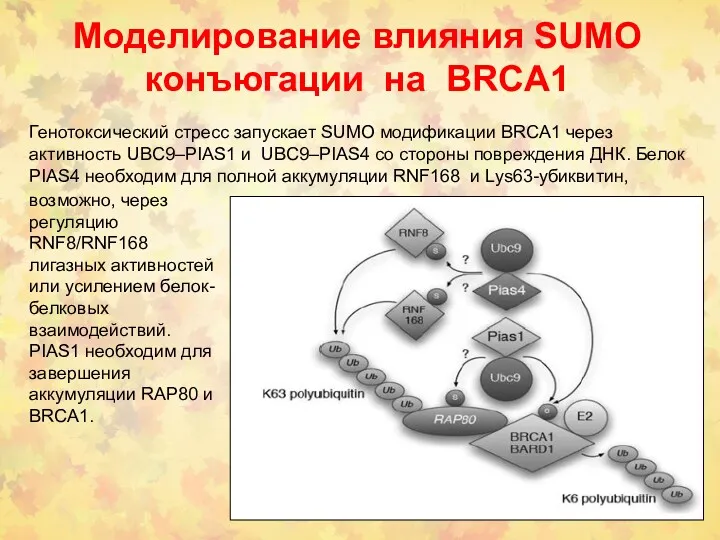
- 60. Моделирование влияния SUMO конъюгации на BRCA1 Генотоксический стресс запускает SUMO модификации BRCA1 через активность UBC9–PIAS1 и
- 61. Репарация и рак
- 62. IY. Эпигенетические модификации ДНК Модификации хроматина, Метилирование ДНК, Геномный импринтинг.
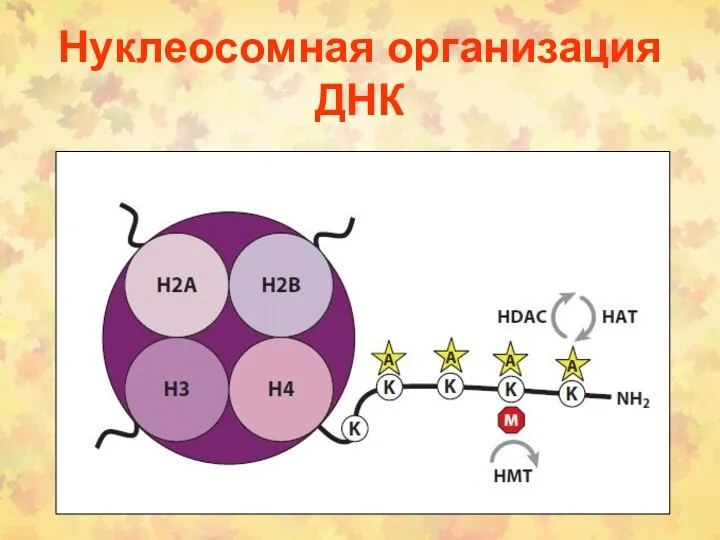
- 63. Нуклеосомная организация ДНК
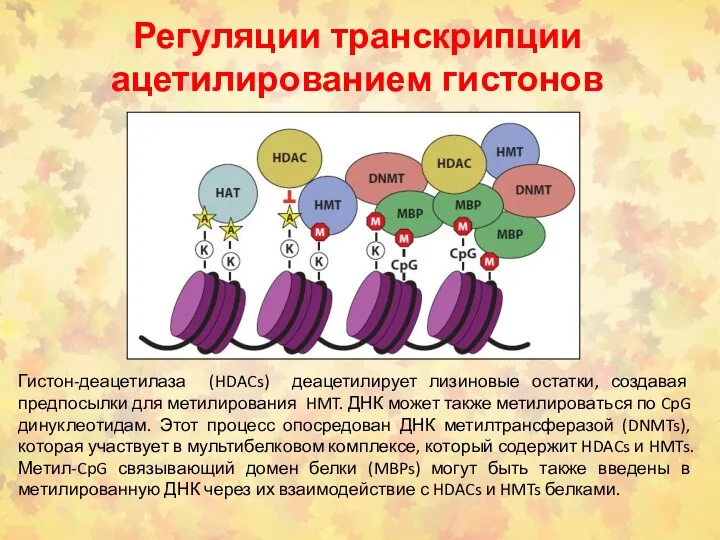
- 64. Регуляции транскрипции ацетилированием гистонов Гистон-деацетилаза (HDACs) деацетилирует лизиновые остатки, создавая предпосылки для метилирования HMT. ДНК может
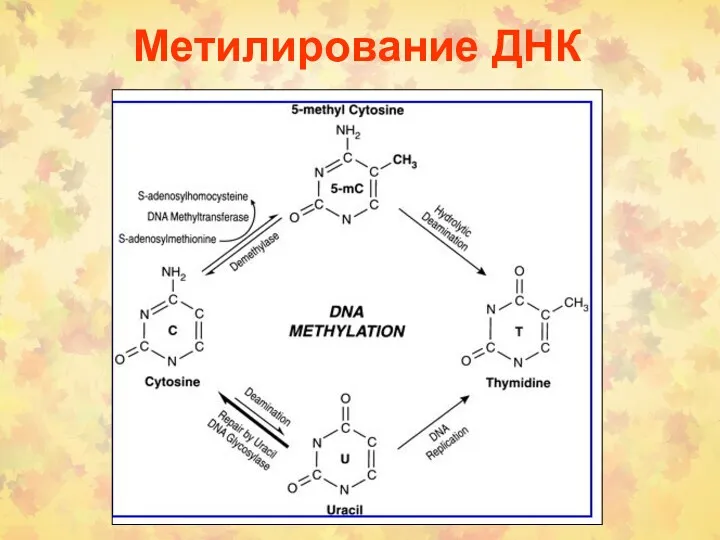
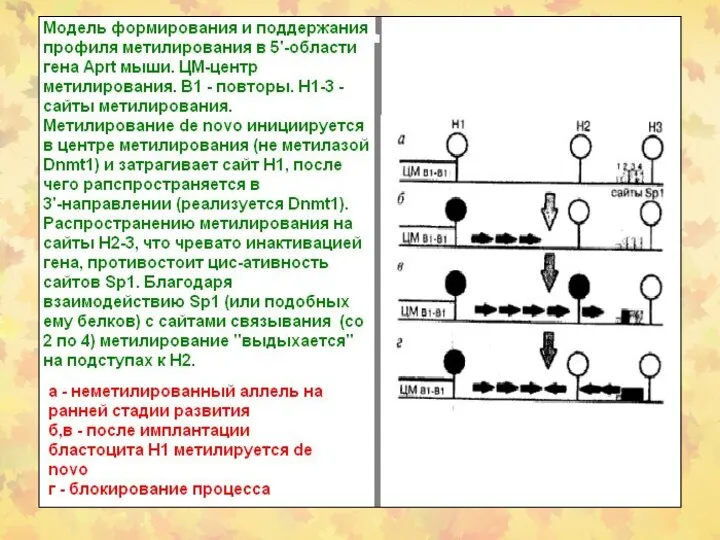
- 65. Метилирование ДНК
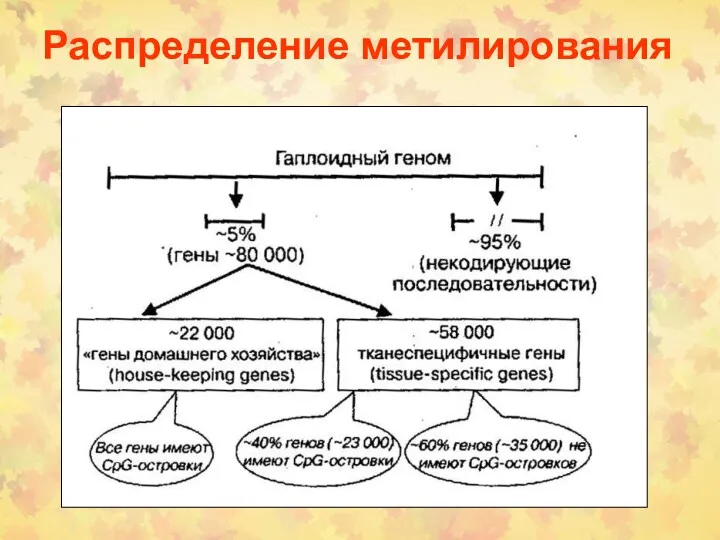
- 67. Распределение метилирования
- 68. Статус метилирования и функциональные особенности промоторов, содержащих CpG-островки
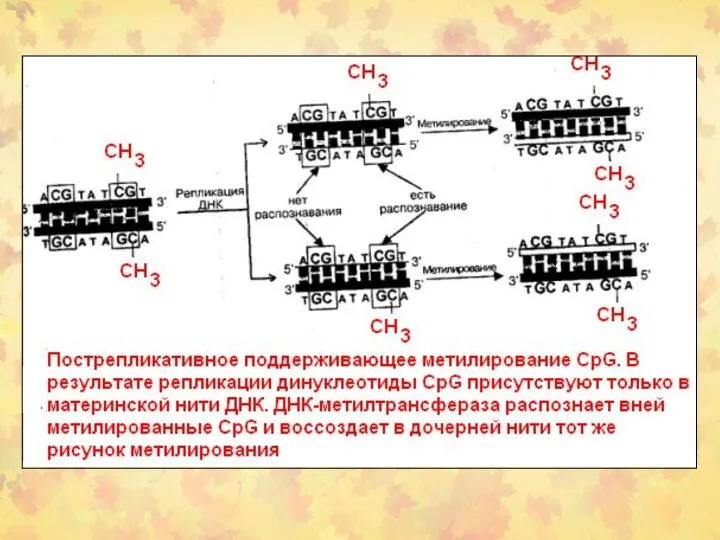
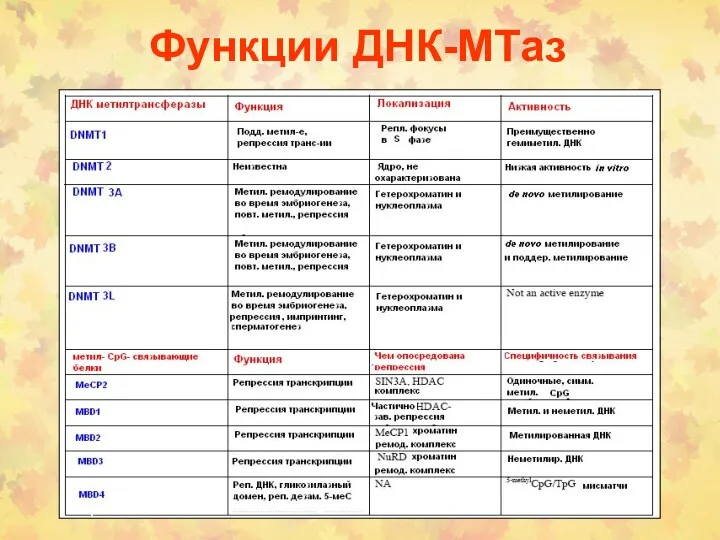
- 70. Функции ДНК-МТаз
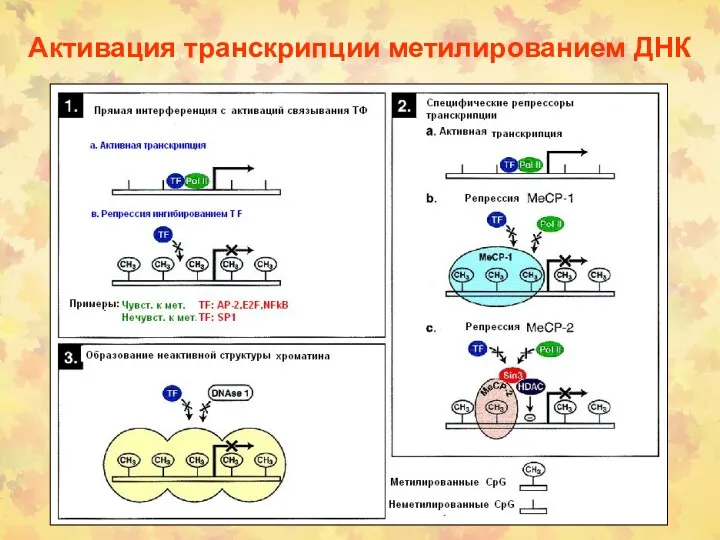
- 71. Активация транскрипции метилированием ДНК
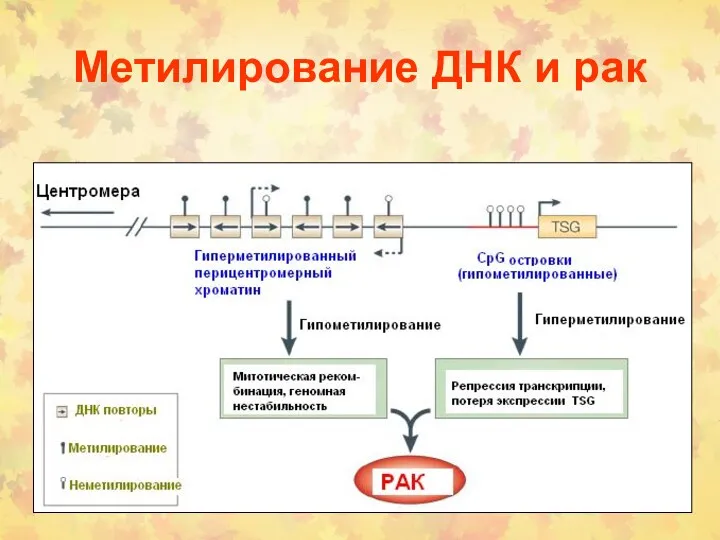
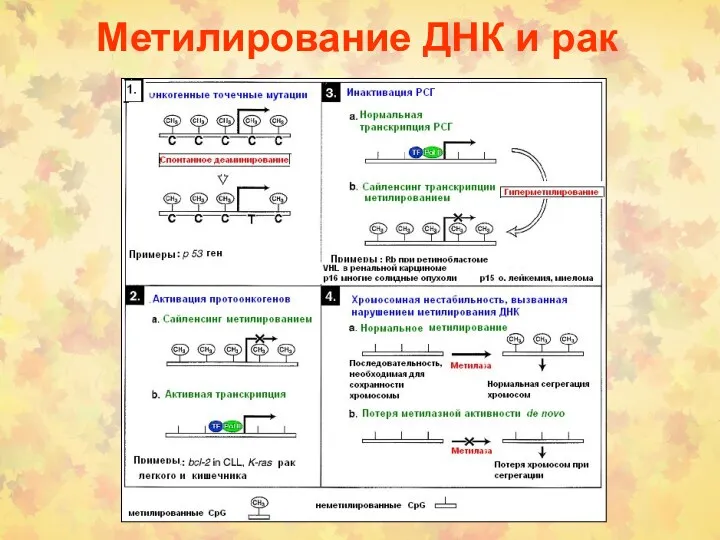
- 73. Метилирование ДНК и рак
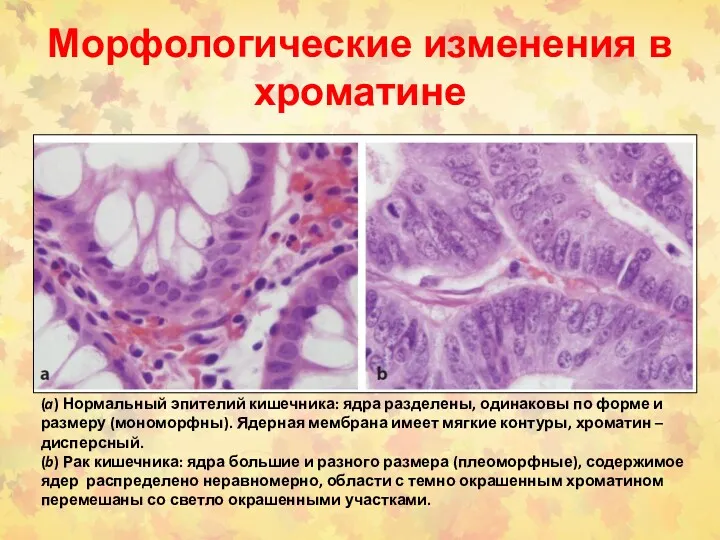
- 74. Морфологические изменения в хроматине (a) Нормальный эпителий кишечника: ядра разделены, одинаковы по форме и размеру (мономорфны).
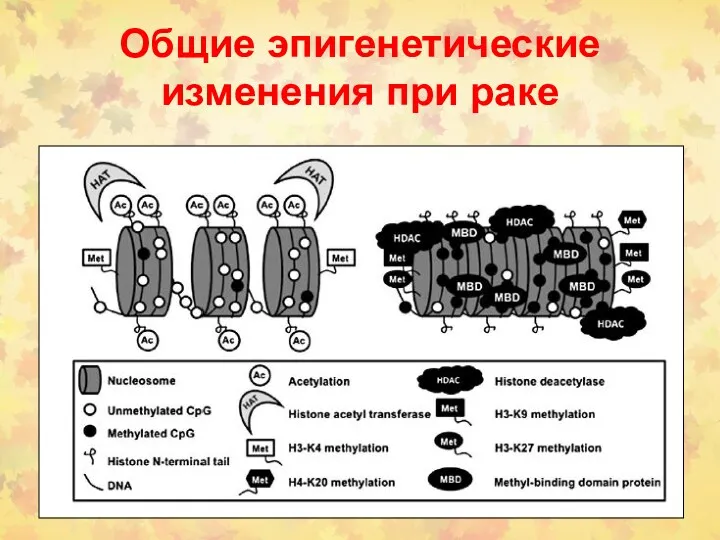
- 75. Общие эпигенетические изменения при раке
- 76. Метилирование ДНК и рак
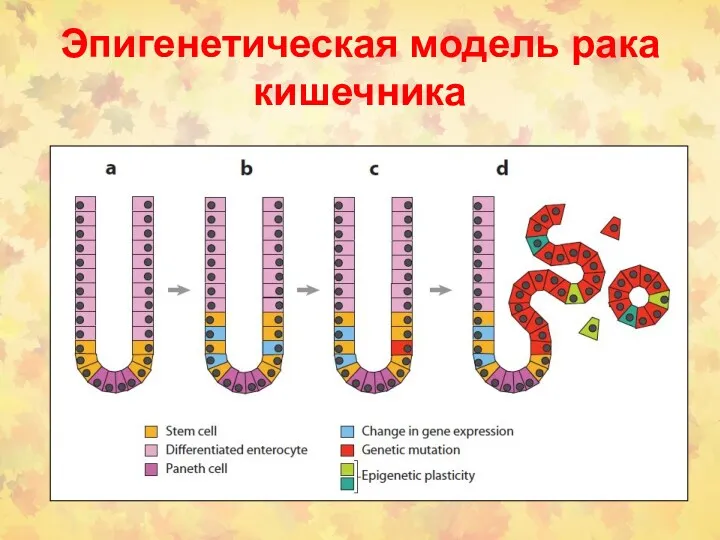
- 77. Эпигенетическая модель рака кишечника
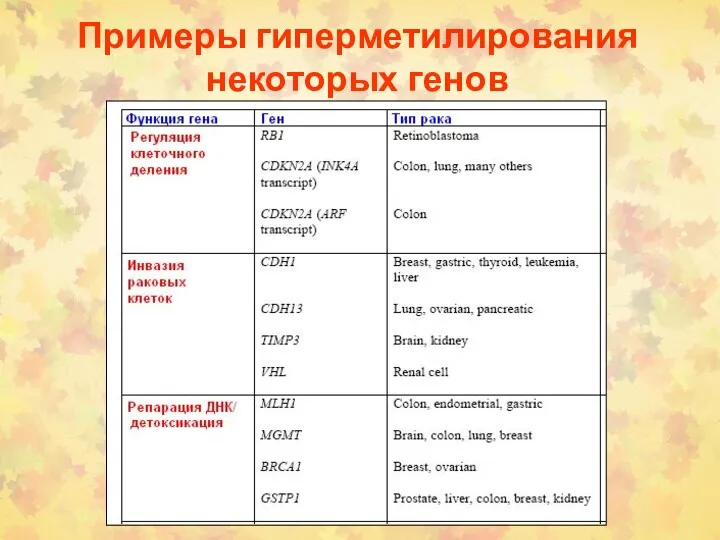
- 78. Примеры гиперметилирования некоторых генов
- 79. Примеры гиперметилирования некоторых генов
- 81. Скачать презентацию
























 Особенности межличностного общения с коллегами. Врачи Скорой помощи
Особенности межличностного общения с коллегами. Врачи Скорой помощи ЭКГ–диагностика нарушений сердечного ритма и проводимости
ЭКГ–диагностика нарушений сердечного ритма и проводимости Тұқым қуалайтын ауруларды емдеудің негізгі принциптері. Генотерапия
Тұқым қуалайтын ауруларды емдеудің негізгі принциптері. Генотерапия Артефакты КТ-изображений. (Лекция 2)
Артефакты КТ-изображений. (Лекция 2) Порядок оказания медицинской помощи беременным женщинам с врожденными пороками внутренних органов у плода
Порядок оказания медицинской помощи беременным женщинам с врожденными пороками внутренних органов у плода Болезнь, вызванная новым коронавирусом 2019-nCoV. Клиническая картина. Лечение. Профилактические мероприятия
Болезнь, вызванная новым коронавирусом 2019-nCoV. Клиническая картина. Лечение. Профилактические мероприятия Лабораторно-инструментальные методы исследования сердечно-сосудистой системы
Лабораторно-инструментальные методы исследования сердечно-сосудистой системы Гингивит, породонтит, ісік тәрізді және идиопатиялық породонт ауруларының клиникасы, диагностикасы және потологиялық анатомиясы
Гингивит, породонтит, ісік тәрізді және идиопатиялық породонт ауруларының клиникасы, диагностикасы және потологиялық анатомиясы Амбулатория жағдайында өкпелік гипертензия кезіндегі рационалды дифференциалды диагностика алгоритмі
Амбулатория жағдайында өкпелік гипертензия кезіндегі рационалды дифференциалды диагностика алгоритмі Неспецифическая хирургическая инфекция
Неспецифическая хирургическая инфекция Развитие в онтогенезе. Новорожденность
Развитие в онтогенезе. Новорожденность Предмет, история развития цель и задачи гигиены как науки и области практической деятельности
Предмет, история развития цель и задачи гигиены как науки и области практической деятельности Эндоцервикоз шейки матки
Эндоцервикоз шейки матки Принципы рациональной фармакотерапии артериальной гипертензии у беременных женщин, при гестационной гипертензии
Принципы рациональной фармакотерапии артериальной гипертензии у беременных женщин, при гестационной гипертензии АВС-реанимация. Принципы интенсивной терапии и реанимации новорожденных
АВС-реанимация. Принципы интенсивной терапии и реанимации новорожденных Методы исследования заболеваний органов дыхания (лекция 3)
Методы исследования заболеваний органов дыхания (лекция 3) 27 Физиология сосудистой системы
27 Физиология сосудистой системы Эпидемиологиялық көрсеткіштердін анализі
Эпидемиологиялық көрсеткіштердін анализі Метил спирті. Этил спирті
Метил спирті. Этил спирті Средства, действующие на периферическую нервную систему
Средства, действующие на периферическую нервную систему Профилактика туберкулеза
Профилактика туберкулеза Дизартрия. Определение понятия дизартрия и ее признаки
Дизартрия. Определение понятия дизартрия и ее признаки Общественное здоровье - современная парадигма улучшения жизни через профилактику хронических неинфекционных заболеваний
Общественное здоровье - современная парадигма улучшения жизни через профилактику хронических неинфекционных заболеваний Система гемостаза. Синдром диссеминированного внутрисосудистого свертывания
Система гемостаза. Синдром диссеминированного внутрисосудистого свертывания Роль медицинской сестры в уходе за пациентом после острого нарушения мозгового кровообращения
Роль медицинской сестры в уходе за пациентом после острого нарушения мозгового кровообращения Оказание первой помощи при укусах змей и насекомых
Оказание первой помощи при укусах змей и насекомых Психология детей с задержкой психического развития со слабовыраженными отклонениями в психическом развитии
Психология детей с задержкой психического развития со слабовыраженными отклонениями в психическом развитии Рентгенологическая диагностика патологии лёгких
Рентгенологическая диагностика патологии лёгких