Содержание
- 2. Руководство к действию По мотивам Principles of transcriptome analysis and gene expression quantification: an RNA-seq tutorial
- 3. «Мокрая» лаборатория Сбор образцов Контаминация Выделение РНК и оценка качества Истощение по рРНК или полиА-обогащение
- 4. «Мокрая» лаборатория Синтез кДНК Создание библиотеки ДНК Фрагментация ДНК/получение ампликонов Лигирование адапторов Амплификация библиотеки Нормализация библиотеки
- 5. «Мокрая» лаборатория Стратегия секвенирования Ion PGM/Ion Proton Пулирование образцов Покрытие Секвенирование Подготовка матрицы Секвенирование
- 6. Биоинформатика Вычислительные ресурсы Навыки программирования Форматы файлов Контроль качества
- 7. Биоинформатика Работа с транскриптомом Variant calling Количественная оценка экспрессии Картирование Определение генов
- 8. Статистика Нормализация Экспрессия генов Альтернативный сплайсинг Функции генов и их взаимодействие
- 9. Уровень экспрессии генов Транскриптом Таргетные исследования 2. Альтернативный сплайсинг 3. Малые РНК (miRNA, siRNA, snoRNA, snRNA,
- 10. RNA-seq Некорректное название изучения РНК с помощью высокопроизводительного секвенирования, т.к. секвенируется не РНК, а кДНК NGS
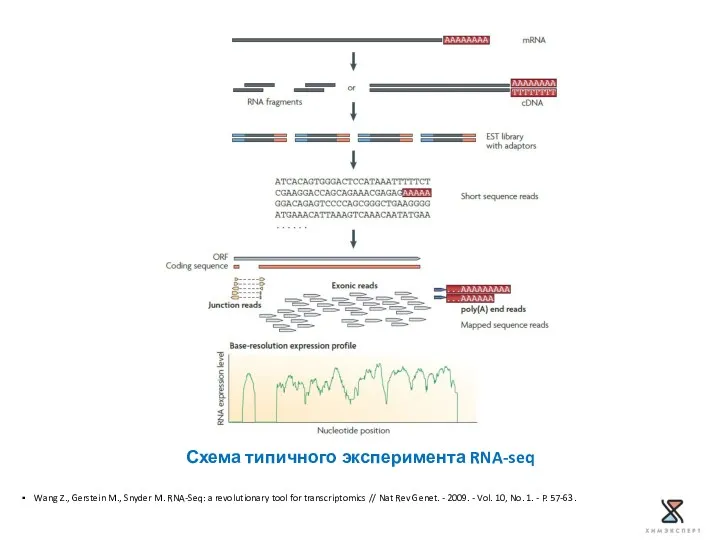
- 11. RNA-seq Принцип RNA-Seq заключается в глубоком секвенировании кДНК с добавленными адаптерами на обоих концах кДНК Wang
- 12. Wang Z., Gerstein M., Snyder M. RNA-Seq: a revolutionary tool for transcriptomics // Nat Rev Genet.
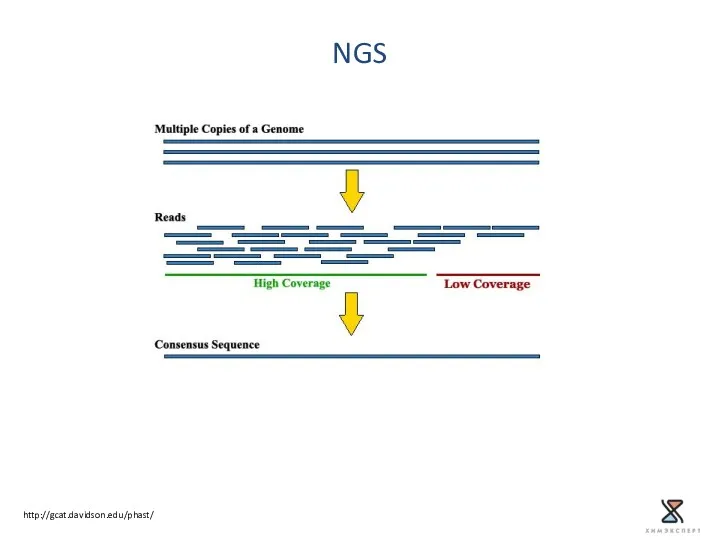
- 13. NGS http://gcat.davidson.edu/phast/
- 14. RNA-Seq для анализа РНК Шаг 1: Стабилизация РНК образце Шаг 2: Выделение РНК из образца Шаг
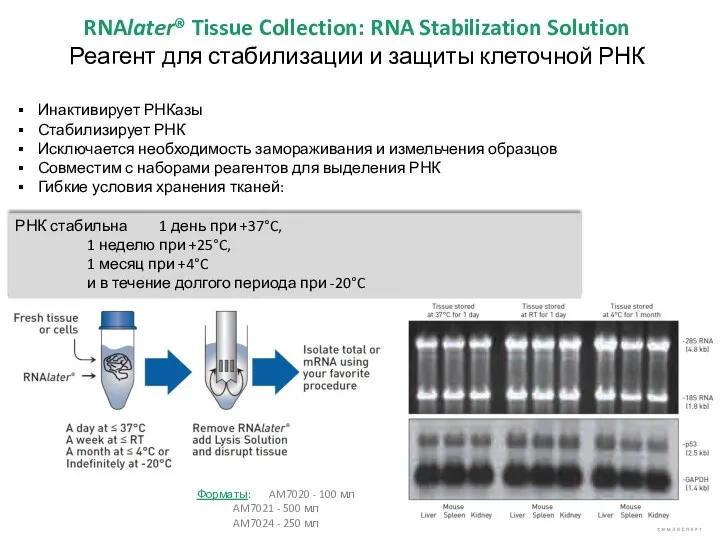
- 15. Шаг 1: Стабилизация РНК
- 16. Инактивирует РНКазы Стабилизирует РНК Исключается необходимость замораживания и измельчения образцов Совместим с наборами реагентов для выделения
- 17. Шаг 2: Выделение РНК
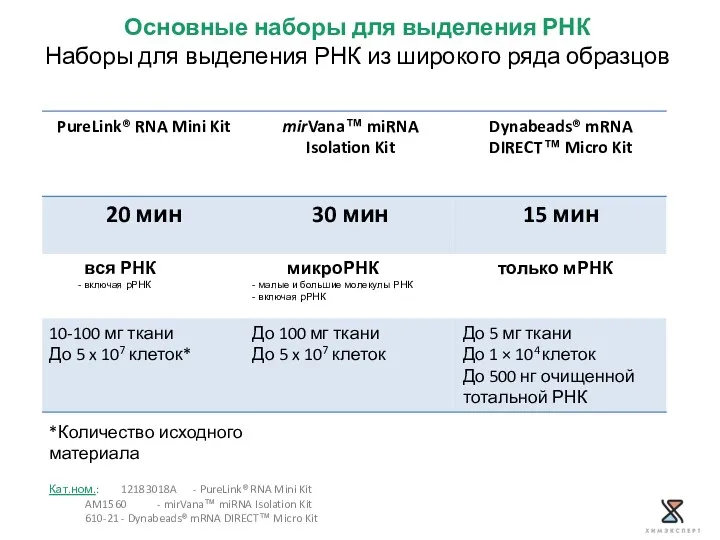
- 18. Основные наборы для выделения РНК Наборы для выделения РНК из широкого ряда образцов *Количество исходного материала
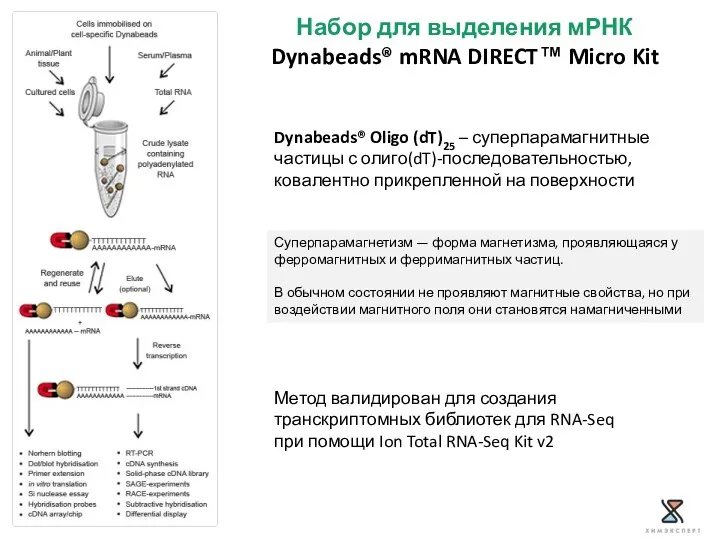
- 19. Набор для выделения мРНК Dynabeads® mRNA DIRECT™ Micro Kit Dynabeads® Oligo (dT)25 – суперпарамагнитные частицы с
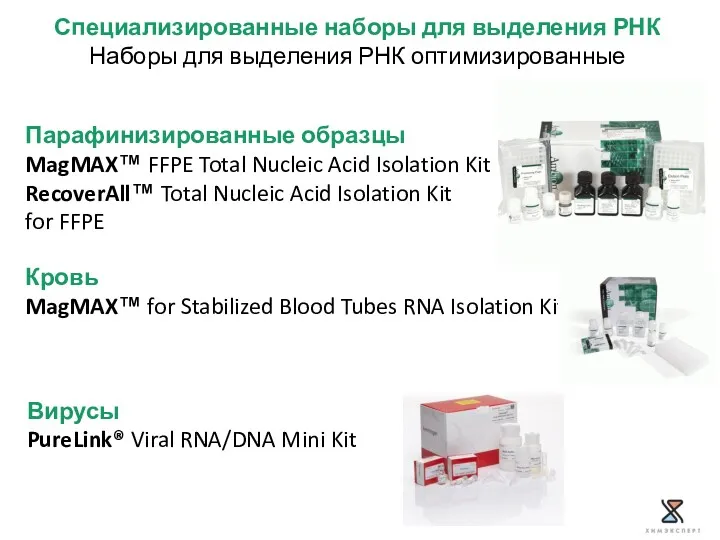
- 20. Специализированные наборы для выделения РНК Наборы для выделения РНК оптимизированные Парафинизированные образцы MagMAX™ FFPE Total Nucleic
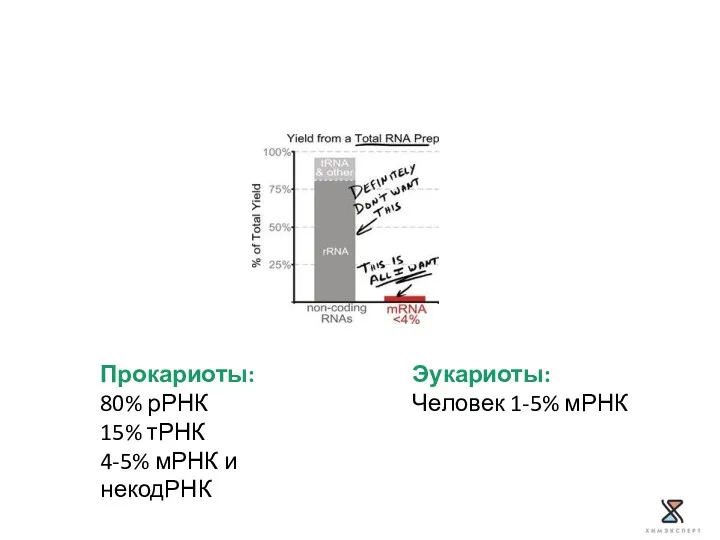
- 21. Прокариоты: 80% рРНК 15% тРНК 4-5% мРНК и некодРНК Эукариоты: Человек 1-5% мРНК
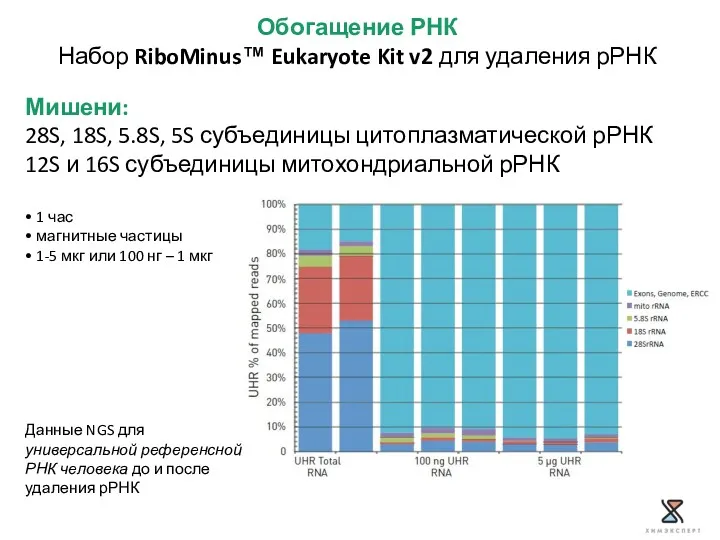
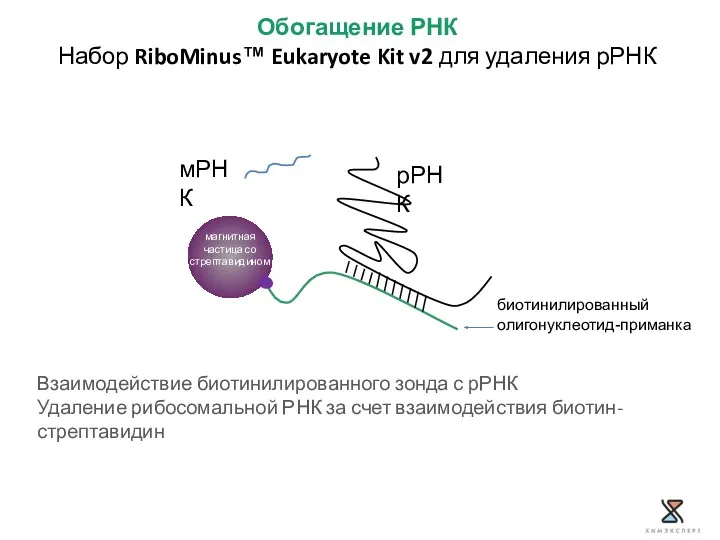
- 22. Обогащение РНК Набор RiboMinus™ Eukaryote Kit v2 для удаления рРНК Мишени: 28S, 18S, 5.8S, 5S субъединицы
- 23. Обогащение РНК Набор RiboMinus™ Eukaryote Kit v2 для удаления рРНК Взаимодействие биотинилированного зонда с рРНК Удаление
- 24. RiboMinus™ для гибридизации с высококонсервативными участками 5S, 5.8S, 18S и 28S рРНК, состоит из смеси олигонуклеотидов
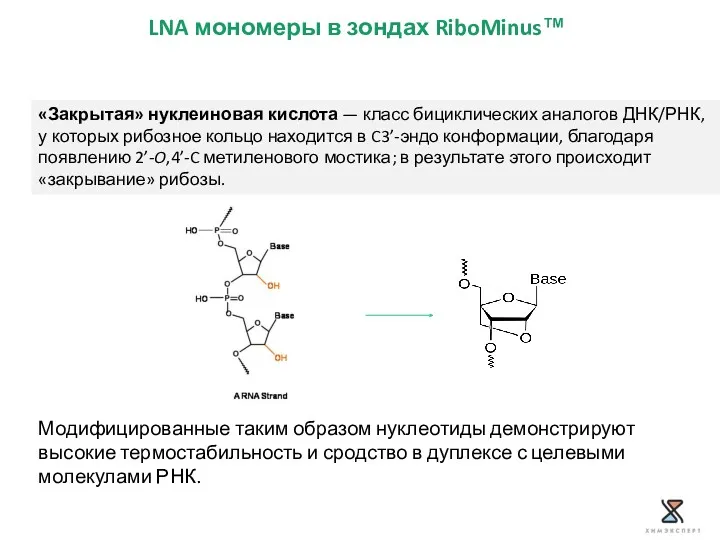
- 26. «Закрытая» нуклеиновая кислота — класс бициклических аналогов ДНК/РНК, у которых рибозное кольцо находится в C3’-эндо конформации,
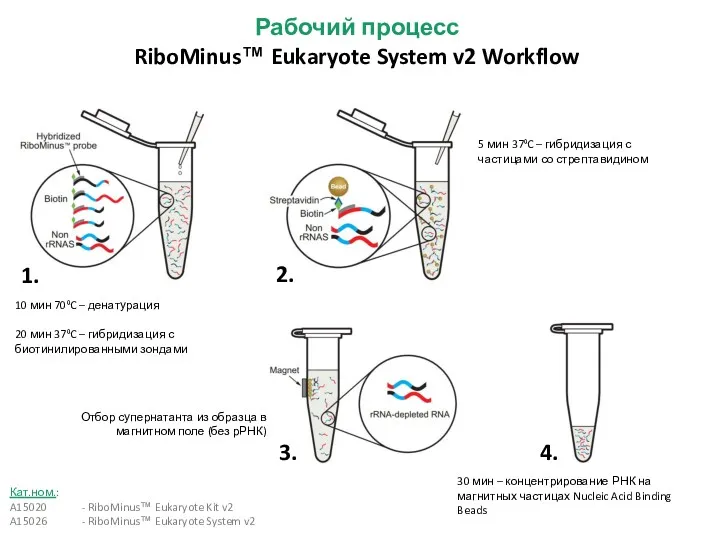
- 27. 1. 2. 3. 4. 10 мин 70⁰C – денатурация 20 мин 37⁰C – гибридизация с биотинилированными
- 28. Контроль The External RNA Controls Consortium (ERCC) Консорциум по контрольной внешней РНК (ERCC) - спецгруппа из
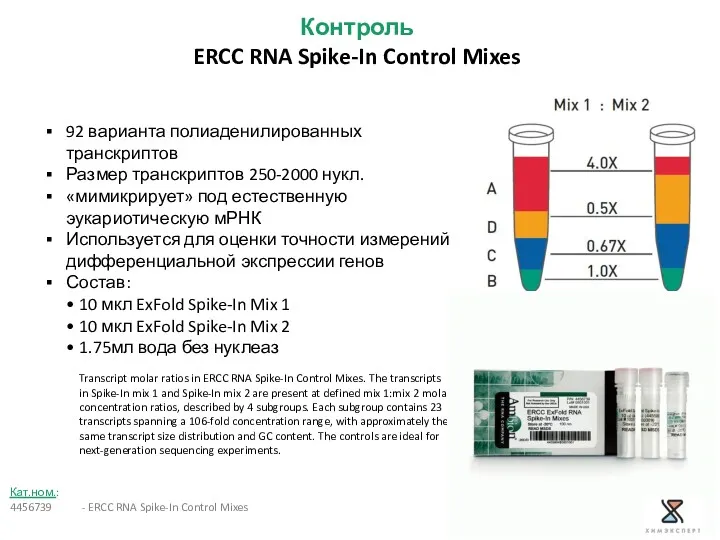
- 29. Контроль ERCC RNA Spike-In Control Mixes Кат.ном.: 4456739 - ERCC RNA Spike-In Control Mixes 92 варианта
- 30. Шаг 3: Приготовление библиотеки
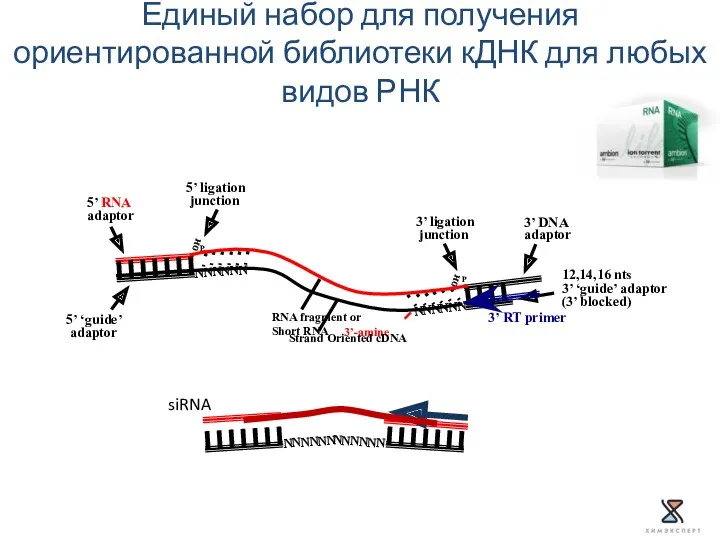
- 31. Набор Ion Total RNA-Seq Kit V2.0 Библиотека за 6 часов Единый набор для получения ориентированной библиотеки
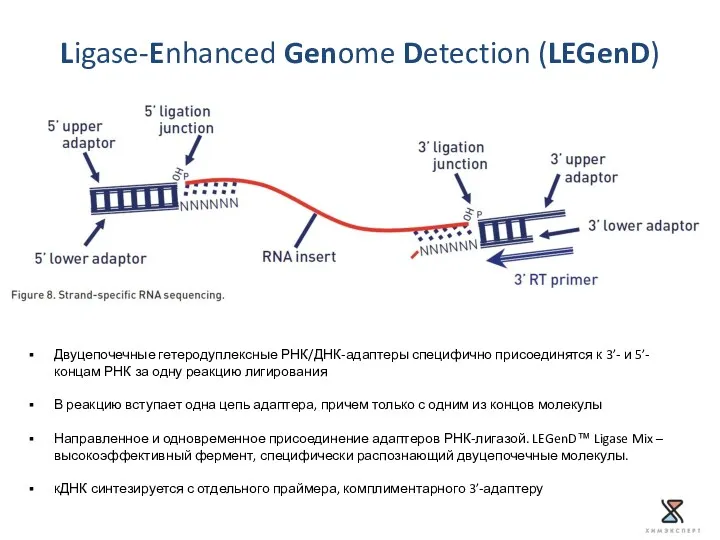
- 32. Ligase-Enhanced Genome Detection (LEGenD) Двуцепочечные гетеродуплексные РНК/ДНК-адаптеры специфично присоединятся к 3’- и 5’-концам РНК за одну
- 33. OH P P OH NNNNNN 3’ DNA adaptor 3’ ‘guide’ adaptor (3’ blocked) 12,14,16 nts Единый
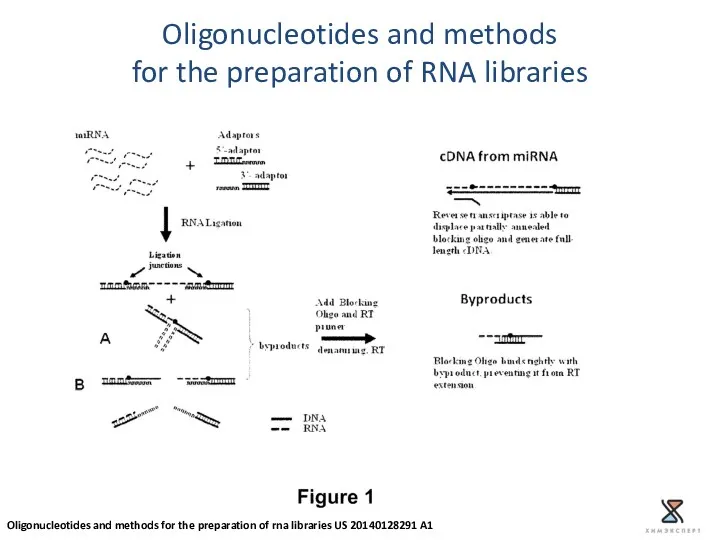
- 34. Oligonucleotides and methods for the preparation of RNA libraries Oligonucleotides and methods for the preparation of
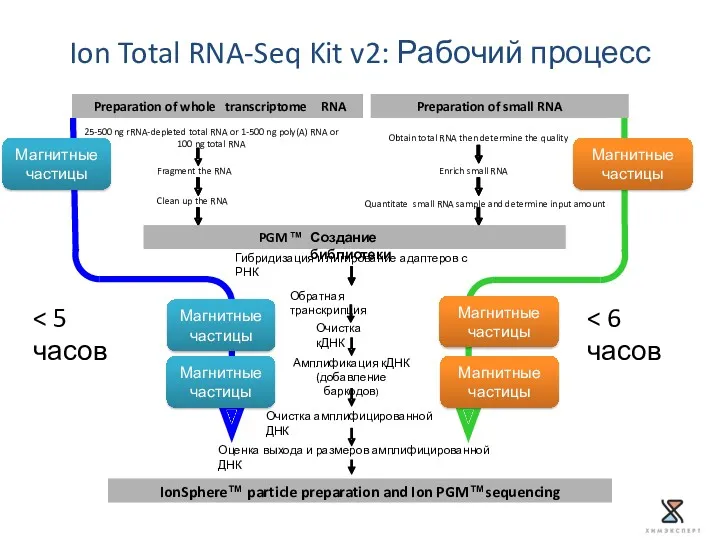
- 35. Ion Total RNA-Seq Kit v2: Рабочий процесс Магнитные частицы Магнитные частицы Магнитные частицы Магнитные частицы Магнитные
- 36. Секвенирование малых РНК Малые РНК – короткие некодирующие молекулы РНК tRNA - транспортировка аминокислот к месту
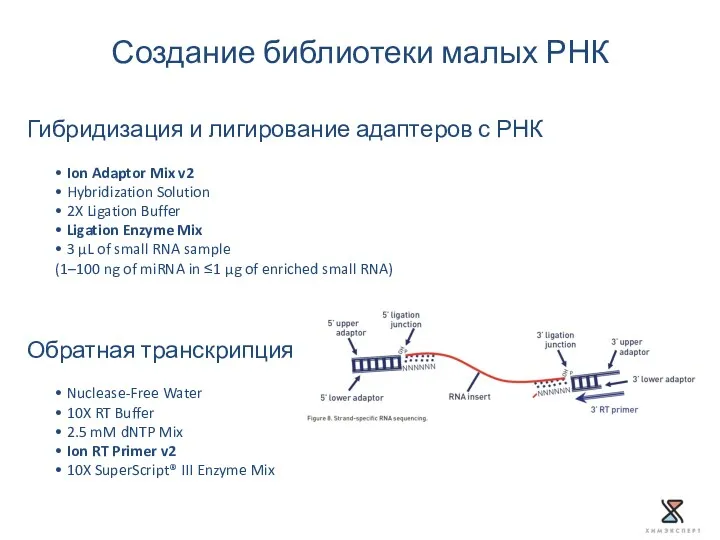
- 37. Создание библиотеки малых РНК Гибридизация и лигирование адаптеров с РНК • Ion Adaptor Mix v2 •
- 38. Фермент для обратной транскрипции SuperScript III (50°С оптимум температуры) Чем выше термостабильность ревертазы, тем выше эффективность
- 39. Создание библиотеки малых РНК Очистка и size-select кДНК на магнитных частицах 1) Первое связывание: на частицах
- 40. Создание библиотеки малых РНК Амплификация кДНК Компоненты Ion Total RNA-Seq Kit v2 для библиотек без баркода:
- 41. Создание библиотеки малых РНК Очистка и size-select амплифицированной ДНК на магнитных частицах 1) Первое связывание: на
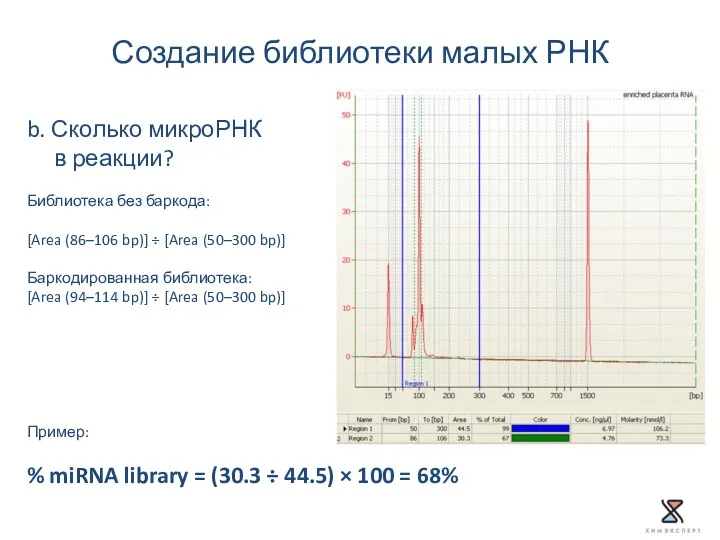
- 42. Создание библиотеки малых РНК Оценка выхода и размеров амплифицированной ДНК Agilent® 2100 Bioanalyzer® instrument with the
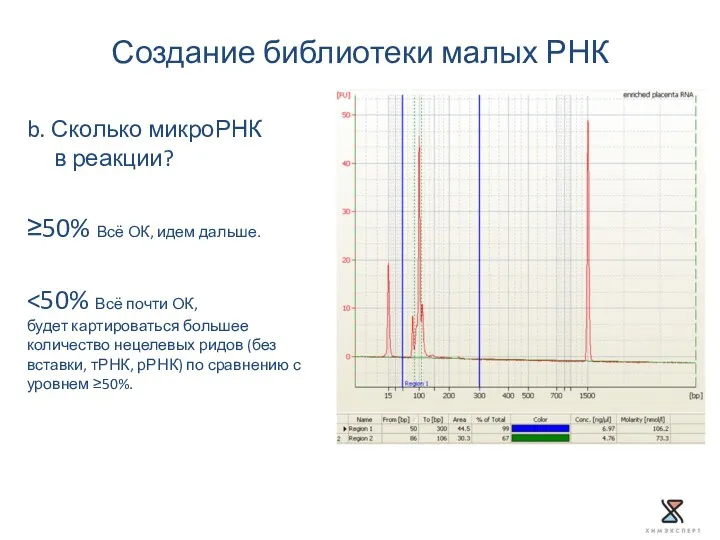
- 43. Создание библиотеки малых РНК b. Сколько микроРНК в реакции? Библиотека без баркода: [Area (86–106 bp)] ÷
- 44. Создание библиотеки малых РНК b. Сколько микроРНК в реакции? ≥50% Всё ОК, идем дальше. будет картироваться
- 45. Создание библиотеки малых РНК Разведение библиотек • Ion PI™ Template OT2 200 kit (обновления!) Конечная концентрация:
- 46. Шаг 4: Секвенирование
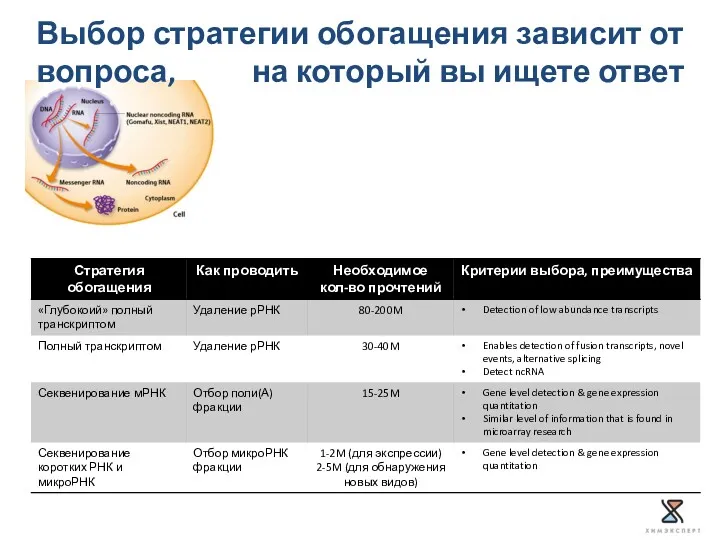
- 47. Выбор стратегии обогащения зависит от вопроса, на который вы ищете ответ
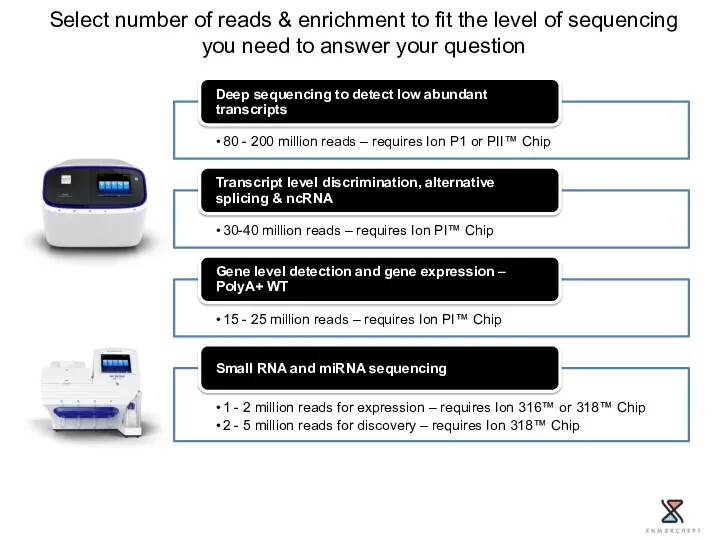
- 48. Select number of reads & enrichment to fit the level of sequencing you need to answer
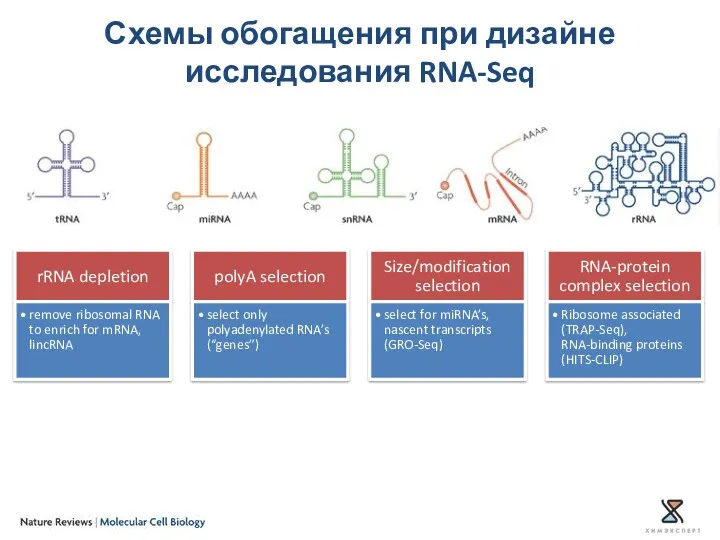
- 49. Схемы обогащения при дизайне исследования RNA-Seq
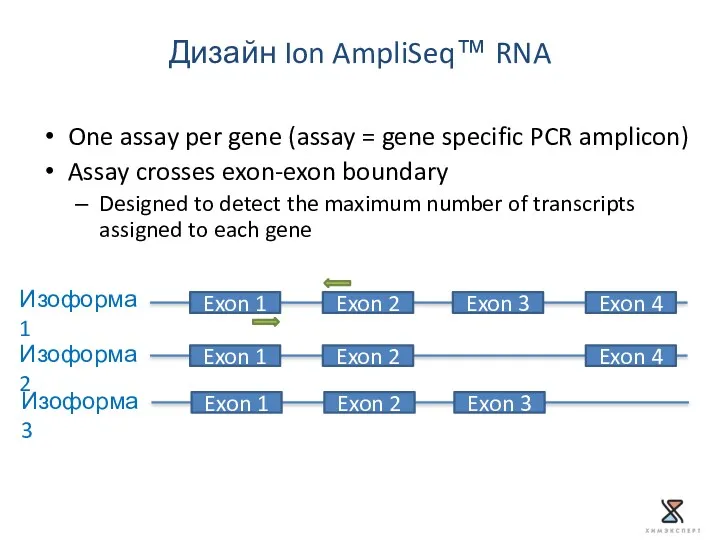
- 50. Технология Ion AmpliSeq™ RNA 1. Обратная транскрипция 2. Ген-специфическая ПЦР 3. Лигирование ION-адаптеров Ion AmpliSeq™ RNA
- 51. One assay per gene (assay = gene specific PCR amplicon) Assay crosses exon-exon boundary Designed to
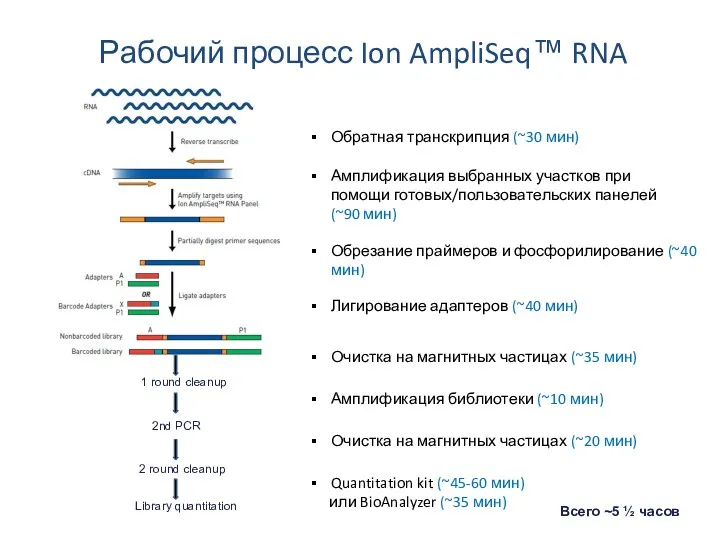
- 52. Обратная транскрипция (~30 мин) Амплификация выбранных участков при помощи готовых/пользовательских панелей (~90 мин) Лигирование адаптеров (~40
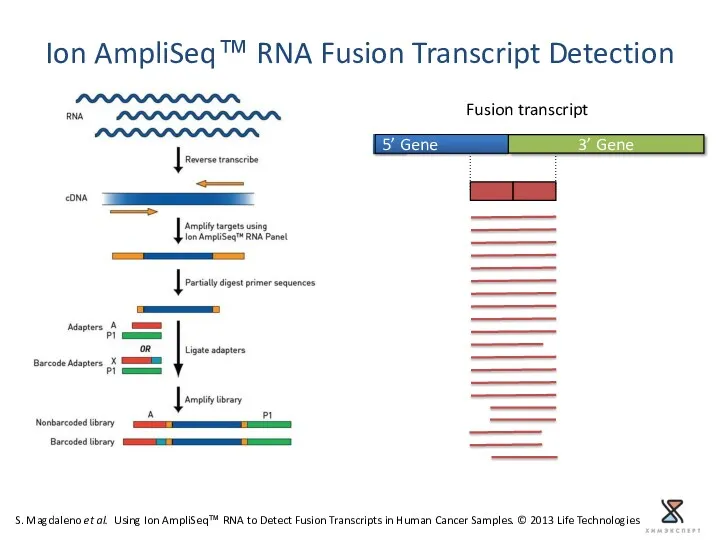
- 53. Fusion transcript Ion AmpliSeq™ RNA Fusion Transcript Detection S. Magdaleno et al. Using Ion AmpliSeq™ RNA
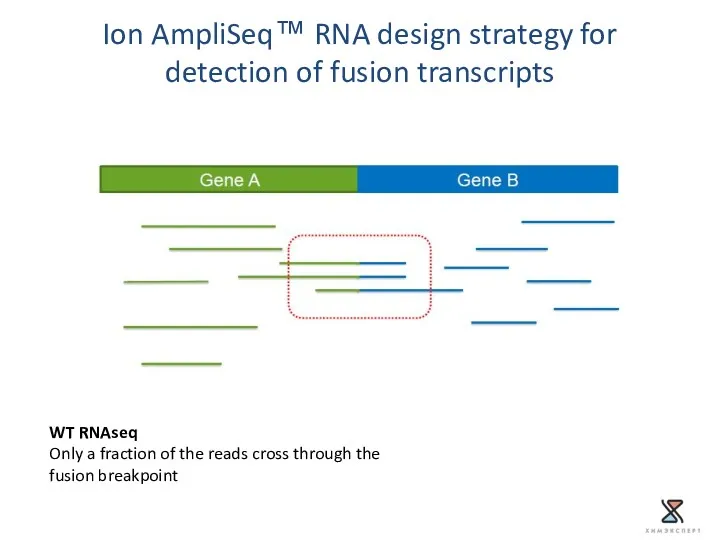
- 54. Ion AmpliSeq™ RNA design strategy for detection of fusion transcripts WT RNAseq Only a fraction of
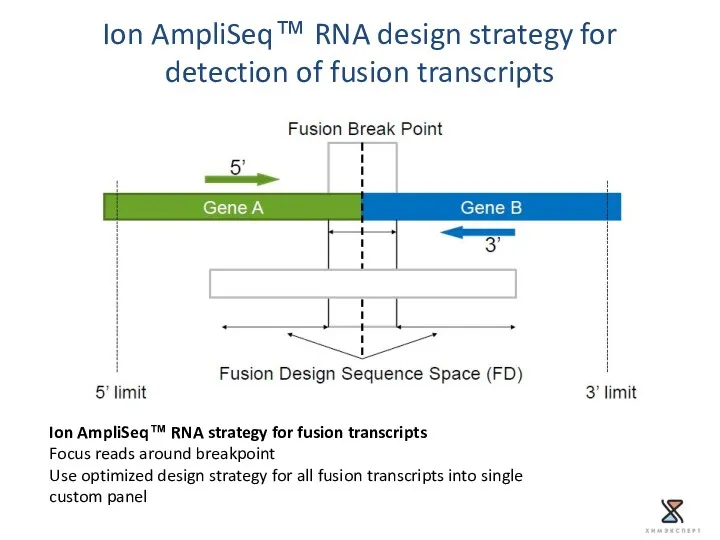
- 55. Ion AmpliSeq™ RNA design strategy for detection of fusion transcripts Ion AmpliSeq™ RNA strategy for fusion
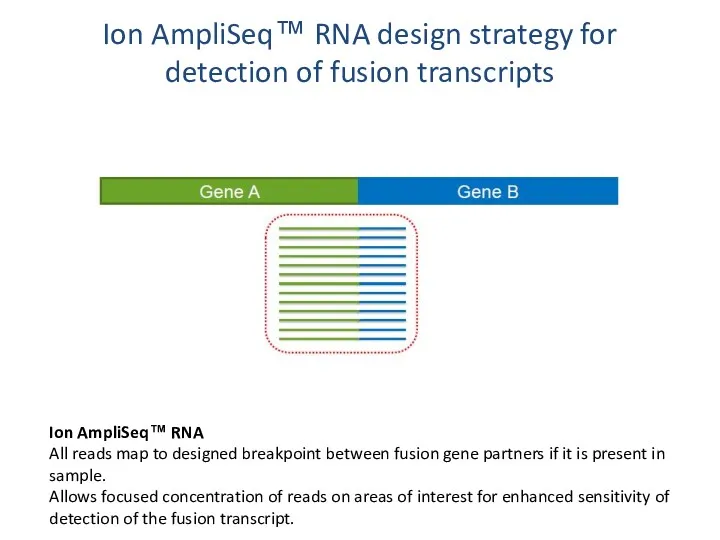
- 56. Ion AmpliSeq™ RNA design strategy for detection of fusion transcripts Ion AmpliSeq™ RNA All reads map
- 57. RNA Cancer Panel 50 генов Соответствует генам панели AmpliSeq Cancer Hotspot v2 RNA Stem Cell Panel
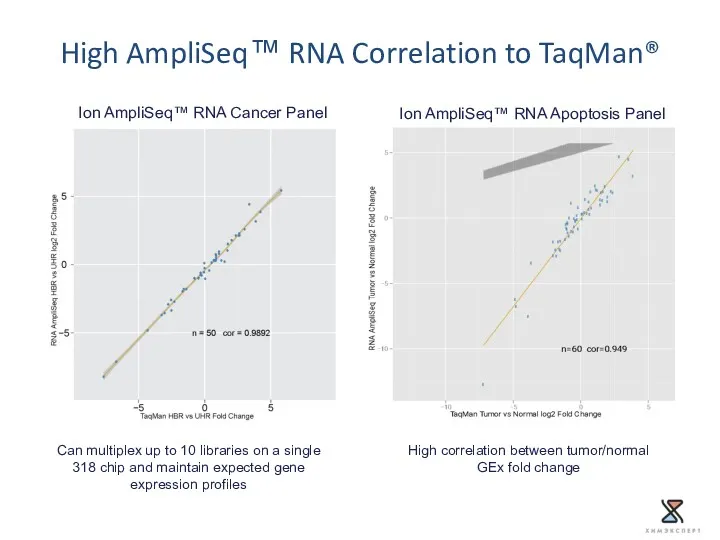
- 58. Can multiplex up to 10 libraries on a single 318 chip and maintain expected gene expression
- 59. Ion AmpliSeq™ Transcriptome Human Gene Expression Research Panel Панель рассчитана на выявление 18 574 кодирующих генов
- 60. Ion AmpliSeq™ Transcriptome Human Gene Expression Research Panel Рекомендация: секвенируйте 8 образцов на чипе Proton PI™
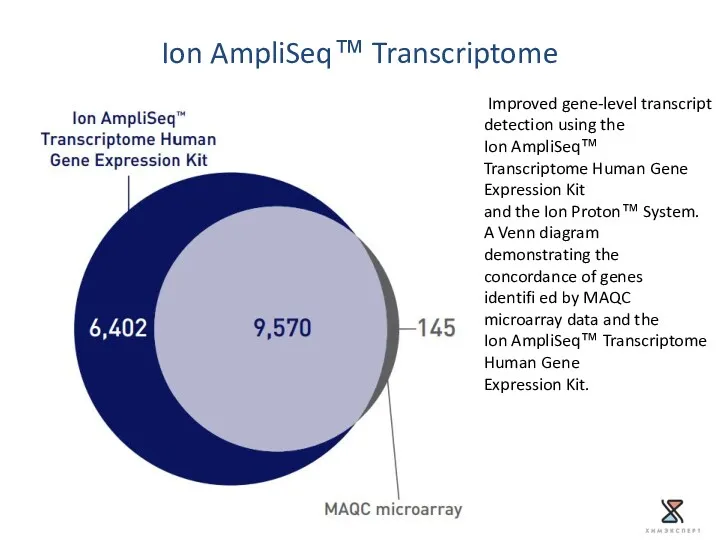
- 61. Ion AmpliSeq™ Transcriptome Improved gene-level transcript detection using the Ion AmpliSeq™ Transcriptome Human Gene Expression Kit
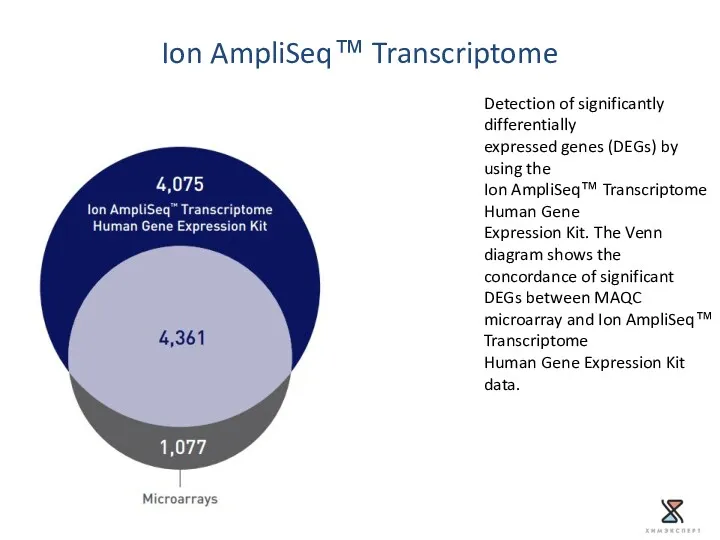
- 62. Ion AmpliSeq™ Transcriptome Detection of significantly differentially expressed genes (DEGs) by using the Ion AmpliSeq™ Transcriptome
- 63. Шаг 5: Анализ данных
- 65. 1. Малые РНК (miRNA, snoRNA, piRNA, snRNA, tRNA и др.)
- 66. 2. Уровень экспрессии генов
- 67. 3. Альтернативный сплайсинг
- 68. 4. Транскриптом одной клетки
- 69. 5. Антисмысловые и некодирующие РНК
- 70. 6. Транслируемые РНК
- 72. Скачать презентацию




































 Мастер- класс Применение модульной технологии в начальной школе
Мастер- класс Применение модульной технологии в начальной школе Птичий грипп
Птичий грипп Негроидная раса
Негроидная раса Проблемное обучение
Проблемное обучение Александр Васильевич Суворов (1729-1800)
Александр Васильевич Суворов (1729-1800) Понятие, виды, значение, условия назначения и выплаты пособий по социальному страхованию
Понятие, виды, значение, условия назначения и выплаты пособий по социальному страхованию Машинобудування
Машинобудування 10 правил презентации
10 правил презентации Символы России. Презентация.
Символы России. Презентация. Анатомо-физиологические особенности детского организма
Анатомо-физиологические особенности детского организма Общественное здоровье населения как экономическая категория. Здоровье населения. Медико-социальные аспекты здоровья
Общественное здоровье населения как экономическая категория. Здоровье населения. Медико-социальные аспекты здоровья Тест. Шинные интерфейсы. Устройства, подключенные к шине
Тест. Шинные интерфейсы. Устройства, подключенные к шине Отчет о проделанной работе волонтерского объединения Надежда
Отчет о проделанной работе волонтерского объединения Надежда Интерактивная игра по безопасности
Интерактивная игра по безопасности Писатели-фронтовики
Писатели-фронтовики Prezentatsia_bez_nazvania 1
Prezentatsia_bez_nazvania 1 Истинное сокровище для людей - умение трудиться
Истинное сокровище для людей - умение трудиться Хранимые процедуры
Хранимые процедуры Лекция 10 проектирование АТК. Тиристорные электроприводы постоянного тока
Лекция 10 проектирование АТК. Тиристорные электроприводы постоянного тока Перечень нормативно-правовых документов для ФГОС дошкольного образования
Перечень нормативно-правовых документов для ФГОС дошкольного образования Разработка проекта внеурочного воспитательного мероприятия
Разработка проекта внеурочного воспитательного мероприятия Электрические трансформаторы. Характеристики трансформаторов
Электрические трансформаторы. Характеристики трансформаторов История развития языков программирования. Языки программирования
История развития языков программирования. Языки программирования Я фантазирую
Я фантазирую Кровотечения в последовом и раннем послеродовом периоде
Кровотечения в последовом и раннем послеродовом периоде Площадь треугольника. Отношение площадей треугольников, имеющих по равному углу
Площадь треугольника. Отношение площадей треугольников, имеющих по равному углу Основные итоги работы в области метрологического обеспечения
Основные итоги работы в области метрологического обеспечения Projekt Soziales Engagement Präsentation
Projekt Soziales Engagement Präsentation