Содержание
- 2. Плазмиды - внехромосомные элементы наследственности, способные к автономной репликации. Термин введен Ледербергом в 1952 году. Эписомы
- 3. F - плазмида – прототип “fertility factor” – ответственный за конъюгационный перенос в штаммах E. coli.
- 4. Плазмиды Структура - кольцевые или линейные молекулы ДНК размером от 2 до 600 т.п.н. Число копий
- 5. Плазмиды Контролируемый фенотип: Устойчивость к антибиотикам, тяжелым металлам. R – плазмиды (resistance). Деградация органических соединений. D
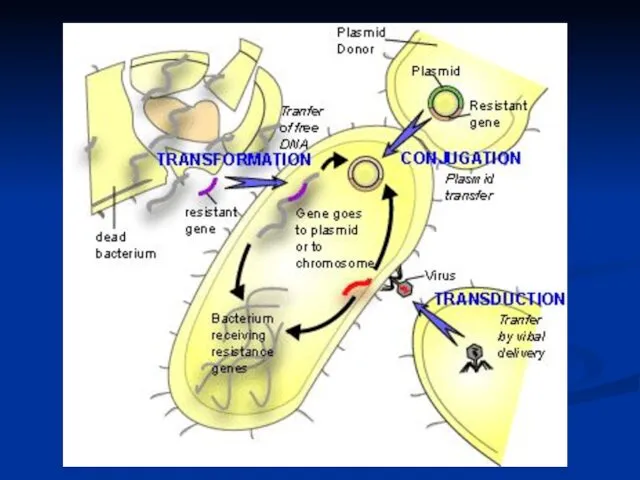
- 6. Конъюгация – процесс обмена генетической информацией между бактериальными клетками, обеспечиваемый плазмидами, путем переноса генетического материала из
- 7. Обозначение плазмид Префикс “p” «Исторические» обозначения: RSF1010, R100, NAH7. R100 фенотип – Tc Cm Sm Su
- 8. Молекулярная организация плазмид Базовый репликон (basic replicon): ori (origin) inc/cop - ген (ы) rep – ген
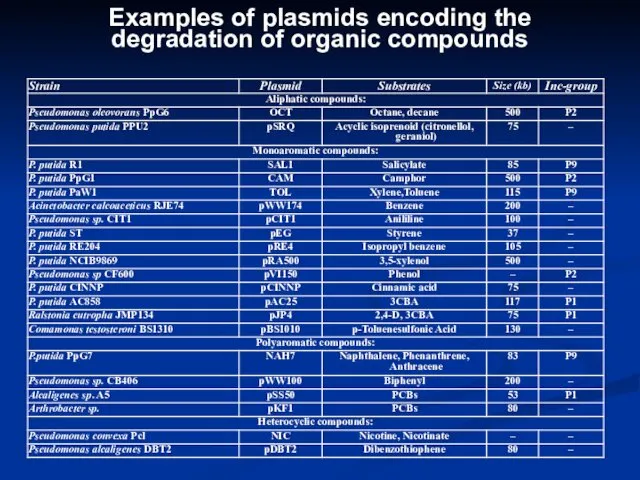
- 9. Examples of plasmids encoding the degradation of organic compounds
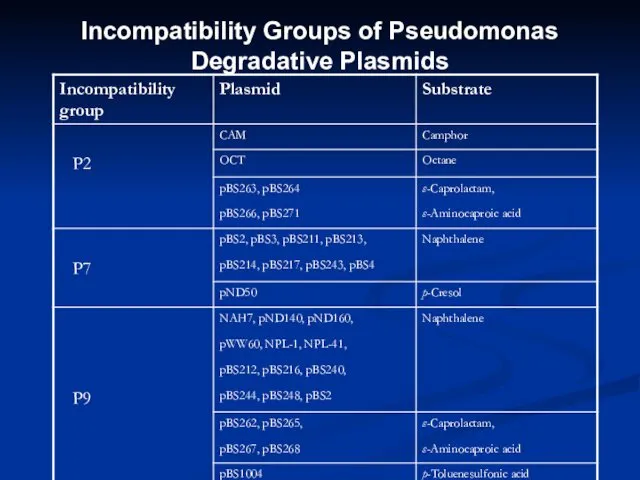
- 10. Incompatibility Groups of Pseudomonas Degradative Plasmids
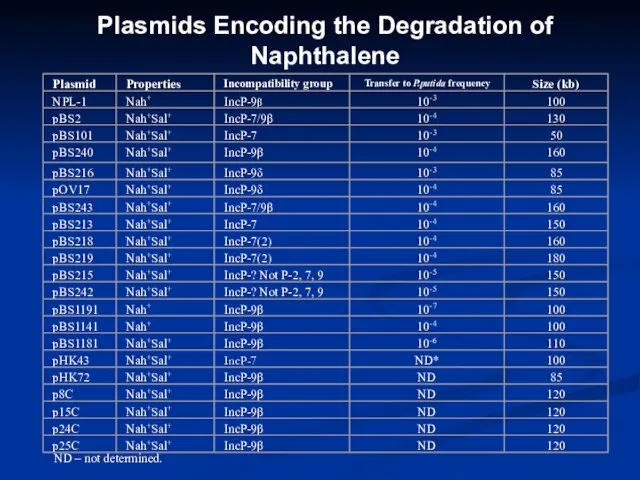
- 11. Plasmids Encoding the Degradation of Naphthalene ND – not determined.
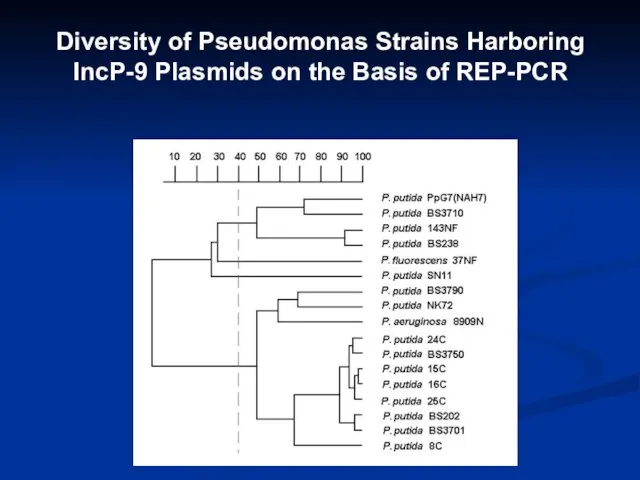
- 12. Diversity of Pseudomonas Strains Harboring IncP-9 Plasmids on the Basis of REP-PCR
- 13. Diversity of the Naphthalene Catabolic Plasmids Belonging to IncP-9 Group on the Basis of RFLP Analysis
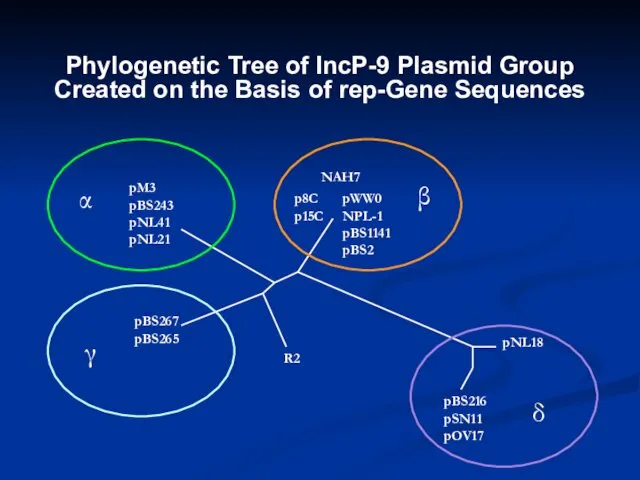
- 14. Phylogenetic Tree of IncP-9 Plasmid Group Created on the Basis of rep-Gene Sequences
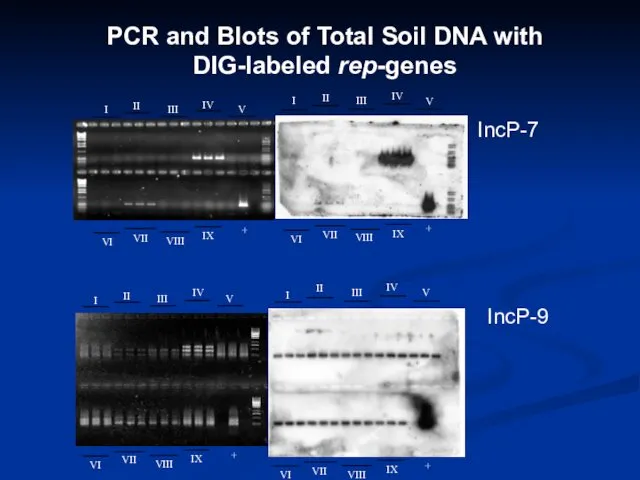
- 15. PCR and Blots of Total Soil DNA with DIG-labeled rep-genes IncP-7 IncP-9
- 16. Phylogenetic Tree of IncP-7 Naphthalene Degrading Plasmids Based on RFLP
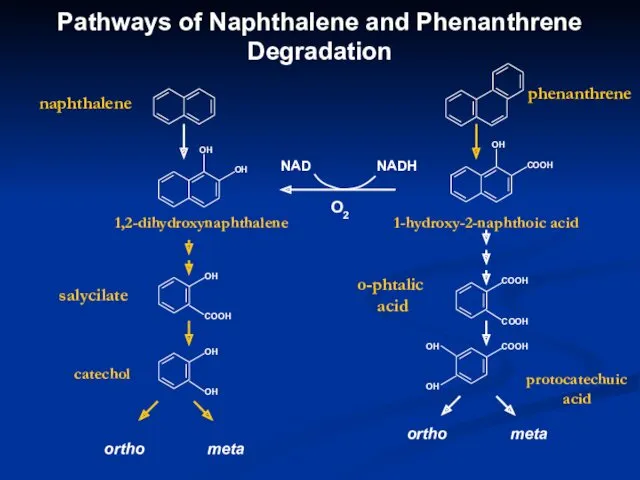
- 17. Pathways of Naphthalene and Phenanthrene Degradation naphthalene phenanthrene 1,2-dihydroxynaphthalene 1-hydroxy-2-naphthoic acid NADН salycilate catechol protocatechuic acid
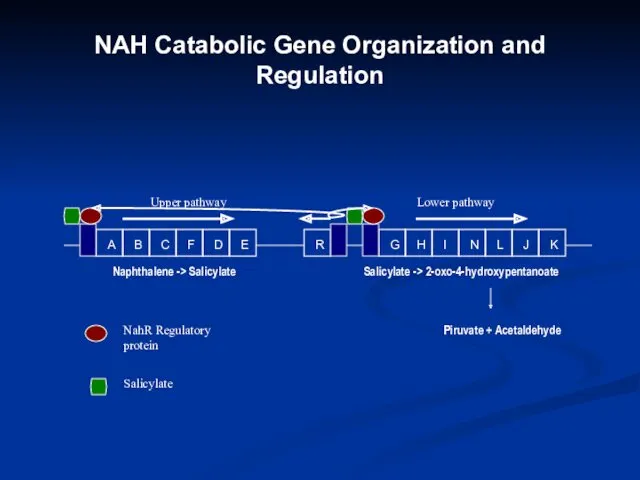
- 18. NAH Catabolic Gene Organization and Regulation A B C F D E R G H I
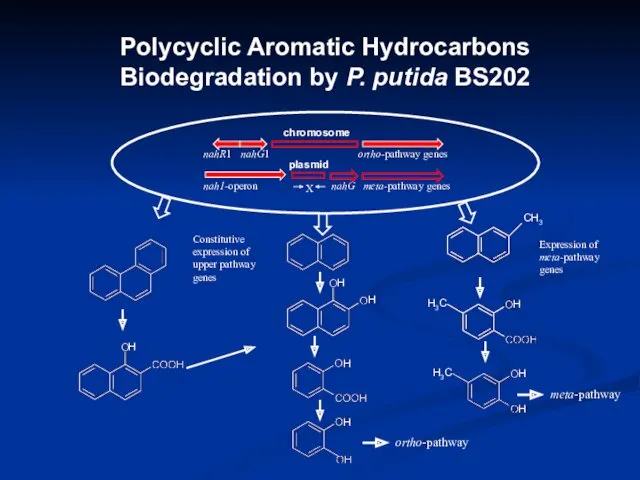
- 19. Polycyclic Aromatic Hydrocarbons Biodegradation by P. putida BS202 meta-pathway
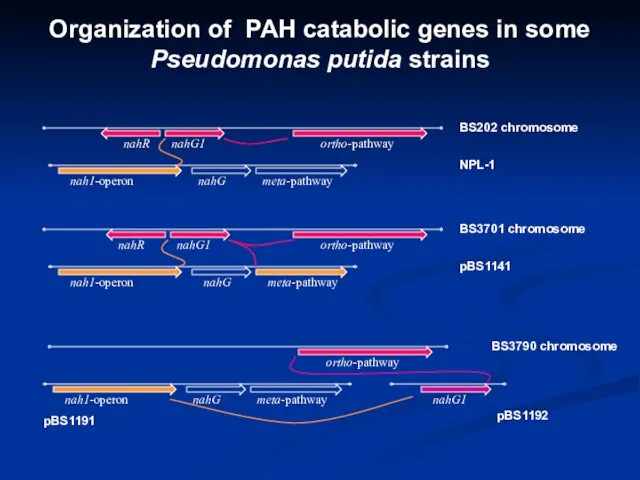
- 20. Organization of PAH catabolic genes in some Pseudomonas putida strains nahG1 nahG meta-pathway nah1-operon BS3790 chromosome
- 21. Diversity of Pseudomonas nahAc genes (RFLP analysis)
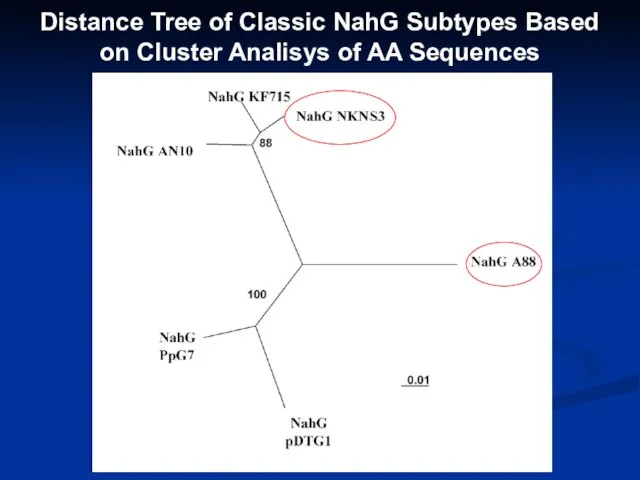
- 22. Distance Tree of Classic NahG Subtypes Based on Cluster Analisys of AA Sequences
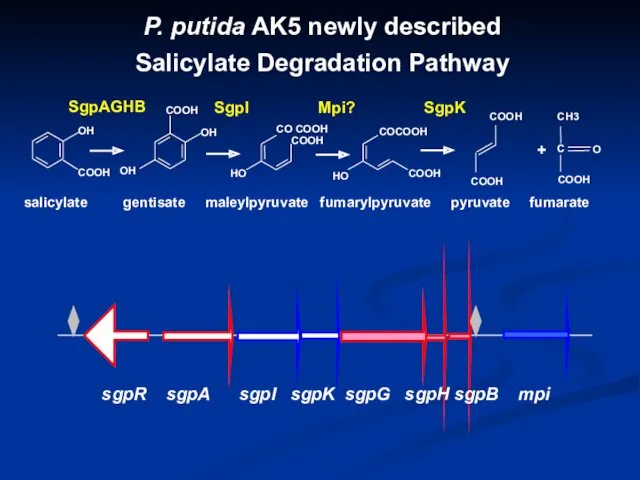
- 23. SgpI Mpi? SgpK + OH COOH salicylate gentisate maleylpyruvate fumarylpyruvate pyruvate fumarate СОOH OH OH SgpAGHB
- 24. Is there a degradative (D) “superplasmid” capable of determining the most efficient degradation of a particular
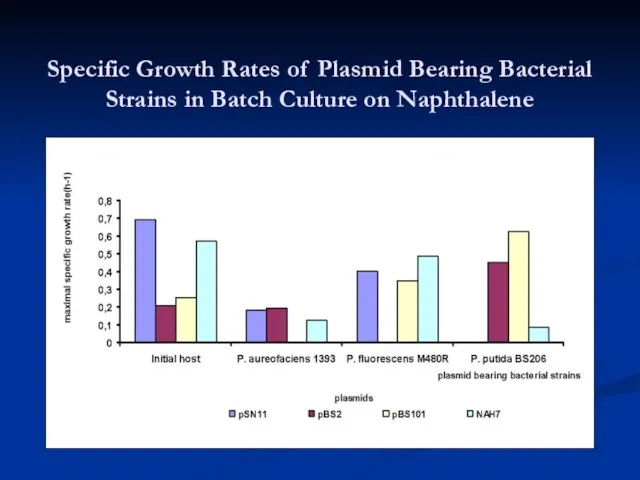
- 25. Specific Growth Rates of Plasmid Bearing Bacterial Strains in Batch Culture on Naphthalene
- 26. Diversity of Microorganisms from Oil Slimes
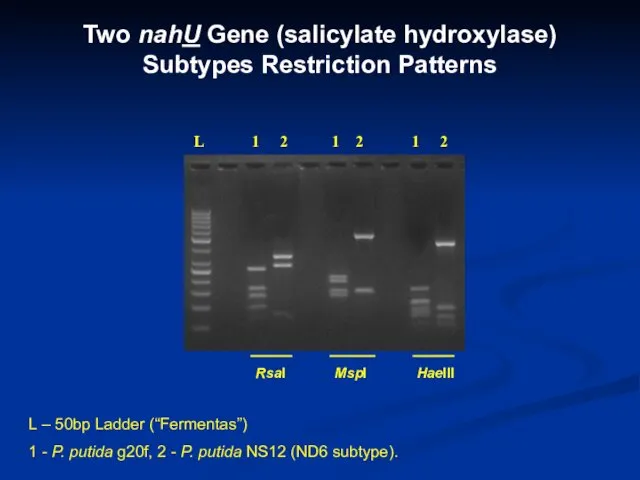
- 27. Two nahU Gene (salicylate hydroxylase) Subtypes Restriction Patterns L – 50bp Ladder (“Fermentas”) 1 - P.
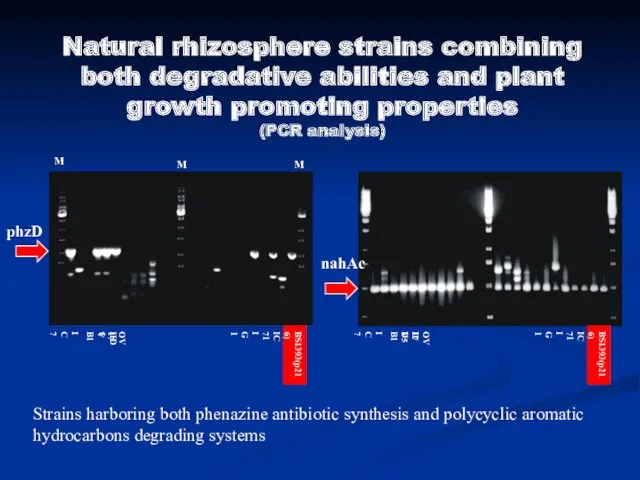
- 28. Natural rhizosphere strains combining both degradative abilities and plant growth promoting properties (PCR analysis) Strains harboring
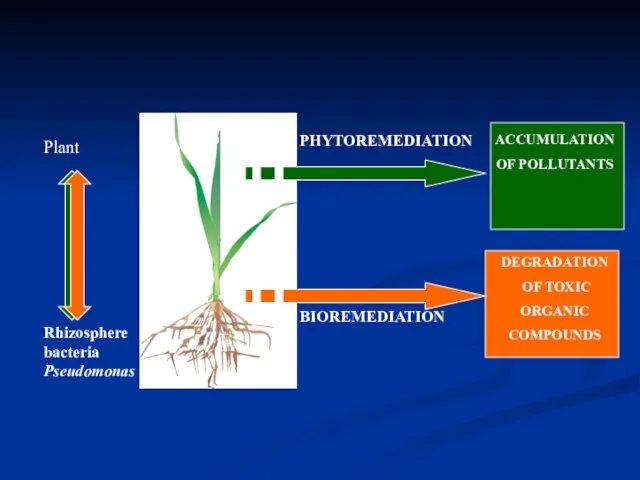
- 29. Plant PHYTOREMEDIATION BIOREMEDIATION Rhizosphere bacteria Pseudomonas DEGRADATION OF TOXIC ORGANIC COMPOUNDS ACCUMULATION OF POLLUTANTS
- 30. Effect of naphthalene degradative plasmids on biosynthesis of phenazine antibiotics by PGPR Pseudomonas 1 – P.
- 31. Mechanisms of arsenic resistance in microorganisms
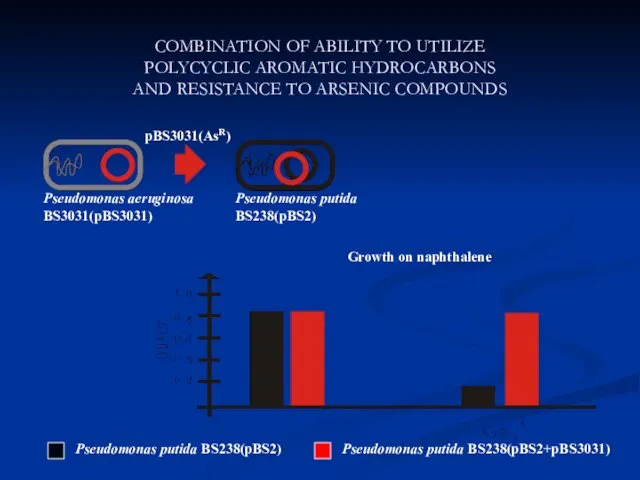
- 32. COMBINATION OF ABILITY TO UTILIZE POLYCYCLIC AROMATIC HYDROCARBONS AND RESISTANCE TO ARSENIC COMPOUNDS pBS3031(AsR) Pseudomonas aeruginosa
- 33. Oil Contamination in Western Siberia The view of oil-contaminated site. July, 1999.
- 34. Extreme Environmental Factors Toxic chemical agents Heavy metals Radionucleides UV-light Low or high pH values High
- 35. Dendrogram of Oil-degrading Strains Based on Their Catabolic and Physiological Properties
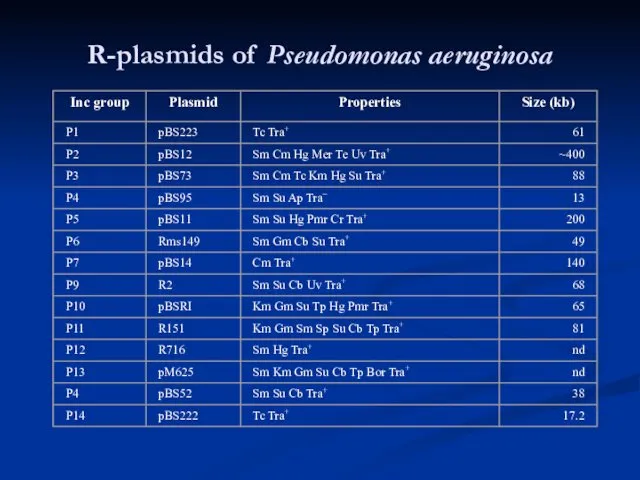
- 38. R-plasmids of Pseudomonas aeruginosa
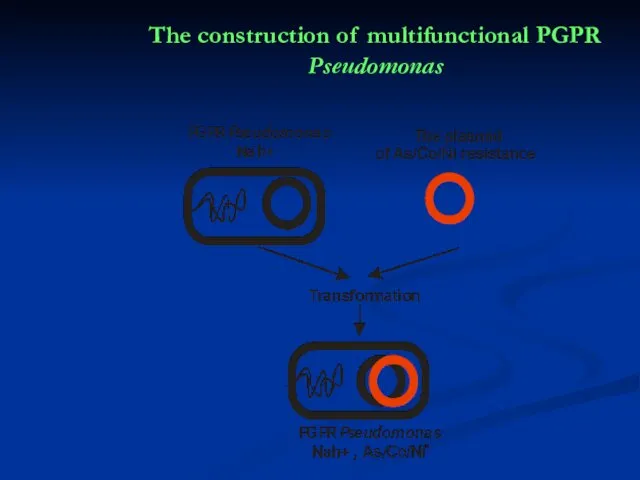
- 39. The construction of multifunctional PGPR Pseudomonas
- 40. The effect of crude oil hydrocarbons on microbial processes in soils, providing CO2 emission into atmosphere,
- 41. The History of Bacterial Genetics (S.E. Luria, 1968) The Stone Age or the Luria-Delbrück Age (1943-1946)
- 43. Скачать презентацию




















 Диаграммы деятельности
Диаграммы деятельности Интегрированный урок обобщающего повторения Путешествуем по миру (география + математика)
Интегрированный урок обобщающего повторения Путешествуем по миру (география + математика) Концепция общества и человека в драматических произведениях М. Горького
Концепция общества и человека в драматических произведениях М. Горького Проектирование многоэтажных жилых зданий массовой застройки для Крайнего Севера
Проектирование многоэтажных жилых зданий массовой застройки для Крайнего Севера Лекция №3. Функции промышленных СУБД
Лекция №3. Функции промышленных СУБД Обыкновенные дроби. Игра Счастливый случай
Обыкновенные дроби. Игра Счастливый случай Разработка алгоритма повышения помехоустойчивости канала связи с использованием технологии MIMO
Разработка алгоритма повышения помехоустойчивости канала связи с использованием технологии MIMO Педагогический опыт
Педагогический опыт Презентация системы работы по профилактике оптической дисграфии
Презентация системы работы по профилактике оптической дисграфии Внутрилабораторный контроль качества лабораторных исследований в УЗ
Внутрилабораторный контроль качества лабораторных исследований в УЗ Мой педагогический опыт
Мой педагогический опыт Технологическая оснастка обсадных колонн. Лекция 8
Технологическая оснастка обсадных колонн. Лекция 8 Сбор макулатуры
Сбор макулатуры Христианский антисемитизм
Христианский антисемитизм Кроссворд Насекомые; 1-2 класс
Кроссворд Насекомые; 1-2 класс Игра Вокгуг света
Игра Вокгуг света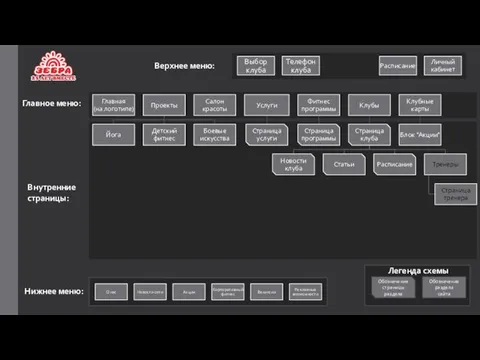 Структура сайта фитнес-клуба
Структура сайта фитнес-клуба презентация по теме: Физические явления в химии
презентация по теме: Физические явления в химии Новые приоритеты государственной образовательной политики в ХХI веке, в сфере исторического и обществоведческого образования
Новые приоритеты государственной образовательной политики в ХХI веке, в сфере исторического и обществоведческого образования Статистические методы обработки информации
Статистические методы обработки информации The Cry of a Wounded Heart
The Cry of a Wounded Heart Острый аппендицит
Острый аппендицит Терминальные состояния. Сердечно-легочная реанимация. Шок
Терминальные состояния. Сердечно-легочная реанимация. Шок Дифференциация обучения с использованием ИКТ для повышения качества обучения.
Дифференциация обучения с использованием ИКТ для повышения качества обучения. Оригами из бумаги. Аппликация Аквариум 1 класс
Оригами из бумаги. Аппликация Аквариум 1 класс Путешествие финикийских мореплавателей
Путешествие финикийских мореплавателей Бальные танцы. Румба.
Бальные танцы. Румба. Тест по химии (8 класс) по теме Генетическая связь классов неорганических соединений для системы тестирования PROClass
Тест по химии (8 класс) по теме Генетическая связь классов неорганических соединений для системы тестирования PROClass