Содержание
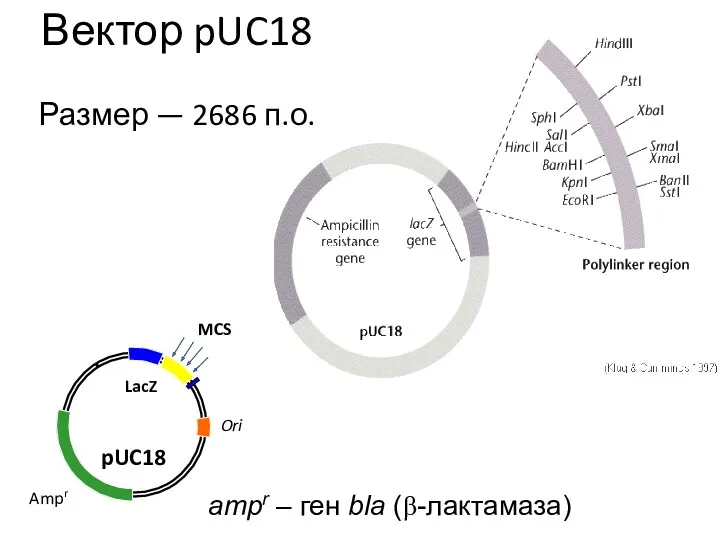
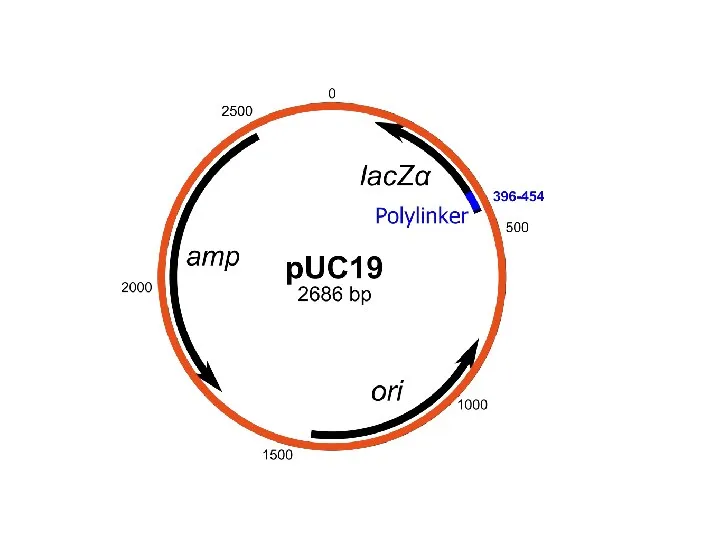
- 2. Вектор pUC18 Размер — 2686 п.о. ampr – ген bla (β-лактамаза)
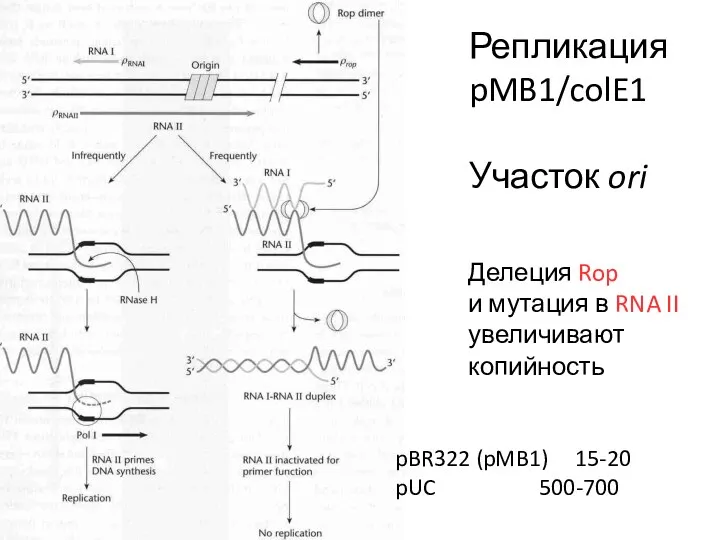
- 3. Делеция Rop и мутация в RNA II увеличивают копийность pBR322 (pMB1) 15-20 pUC 500-700 Репликация pMB1/colE1
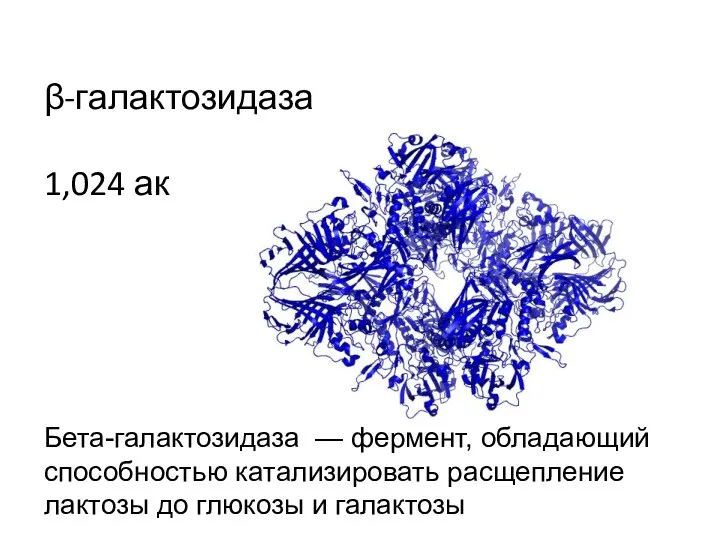
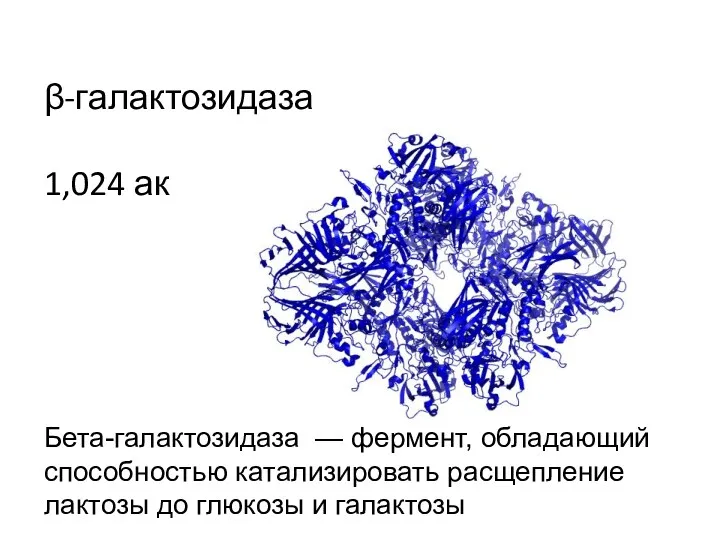
- 4. β-галактозидаза 1,024 ак Бета-галактозидаза — фермент, обладающий способностью катализировать расщепление лактозы до глюкозы и галактозы
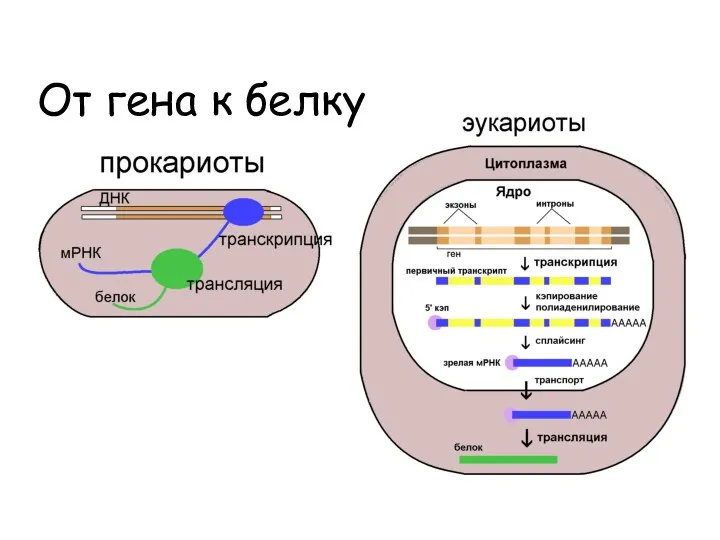
- 5. От гена к белку
- 6. Считываемая цепь ДНК называется матричной или некодирующей. Противоположная цепь называется кодирующей РНК считывается с одной цепи
- 7. Синтез РНК РНК-полимеразой 5’-3’ направление синтеза затравка не нужна РНК-полимераза сама расплетает ДНК Синтез РНК происходит
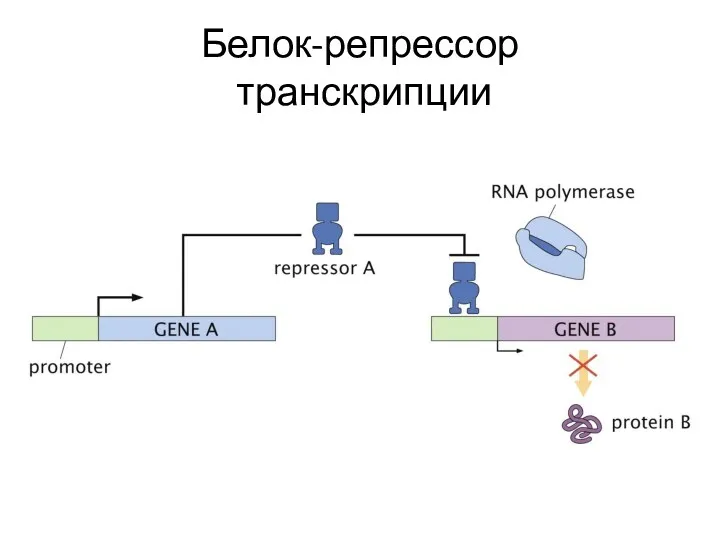
- 8. Белок-репрессор транскрипции
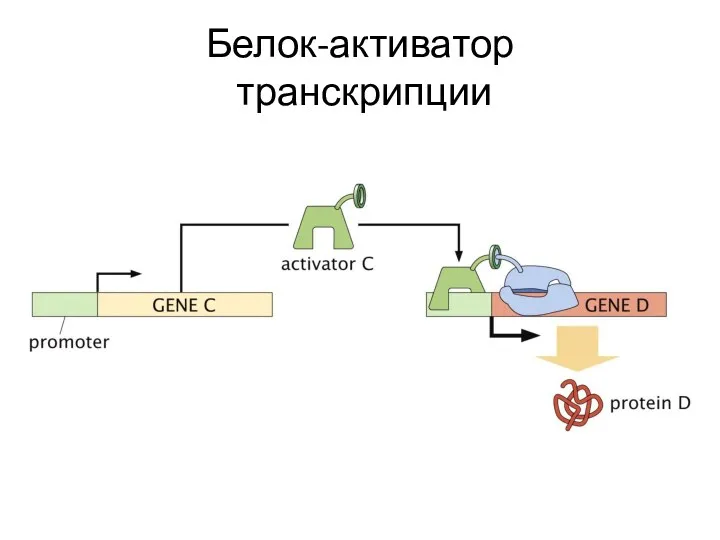
- 9. Белок-активатор транскрипции
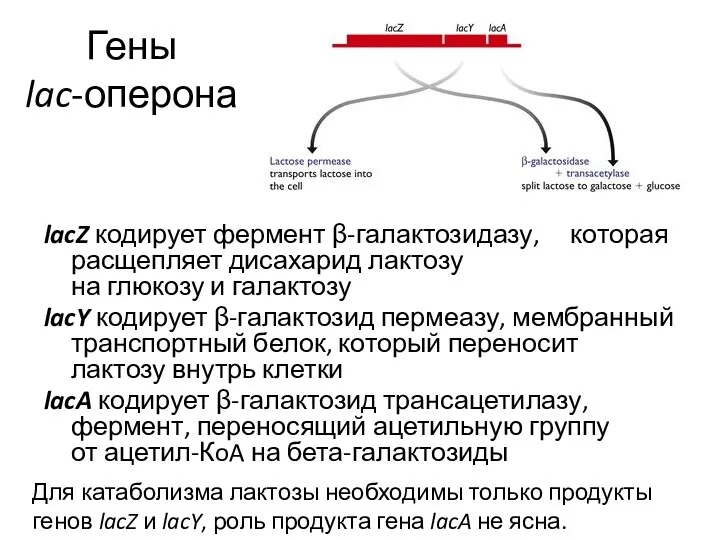
- 10. Гены lac-оперона lacZ кодирует фермент β-галактозидазу, которая расщепляет дисахарид лактозу на глюкозу и галактозу lacY кодирует
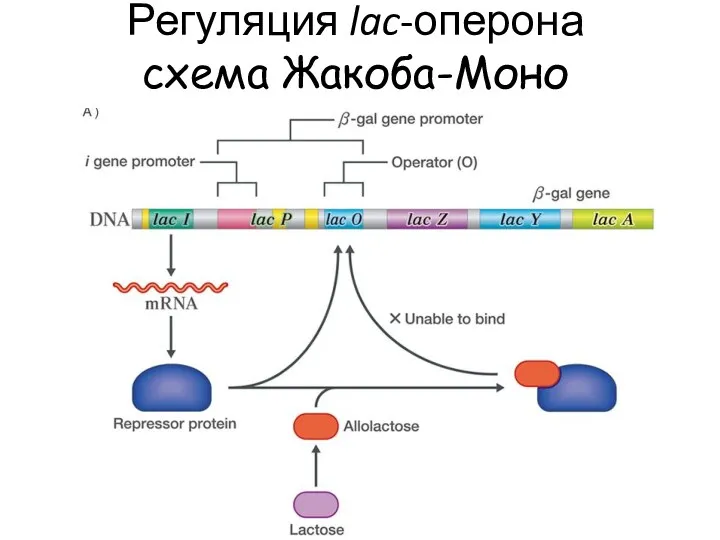
- 11. Регуляция lac-оперона схема Жакоба-Моно
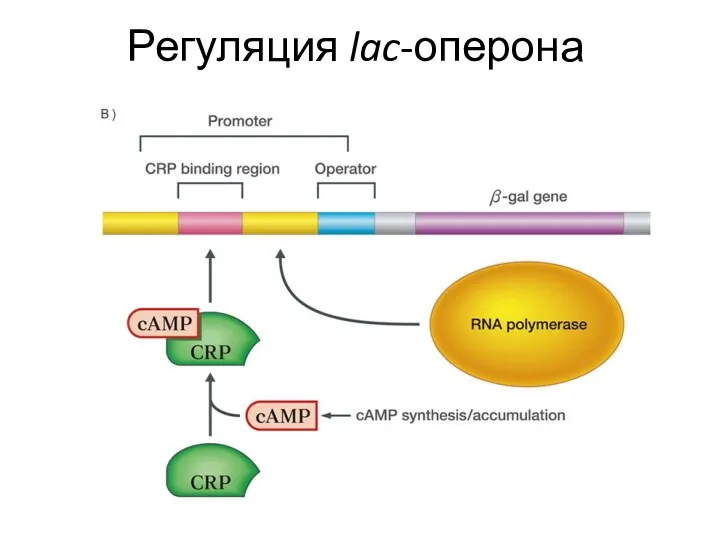
- 12. Регуляция lac-оперона
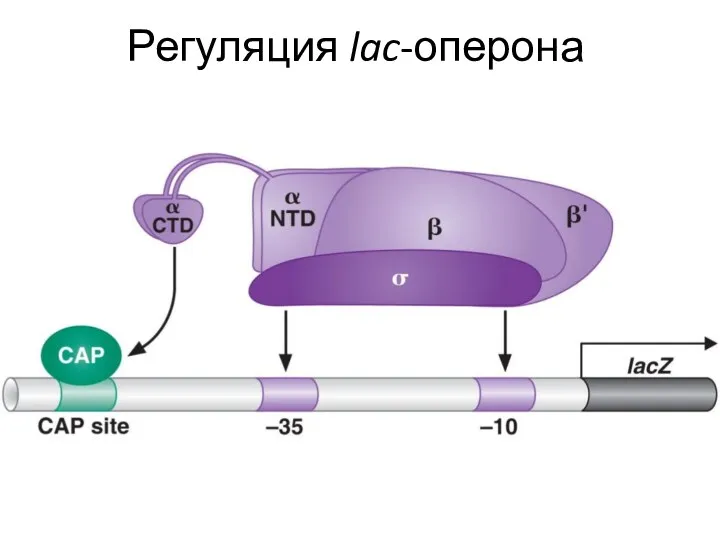
- 13. Регуляция lac-оперона
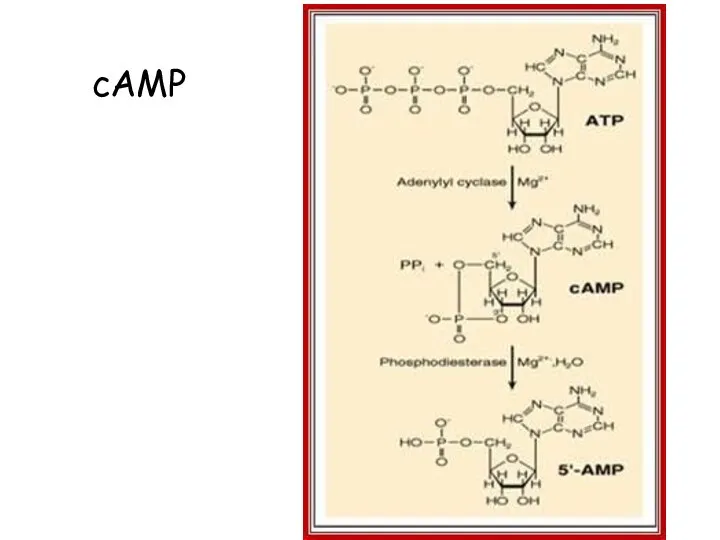
- 14. cAMP
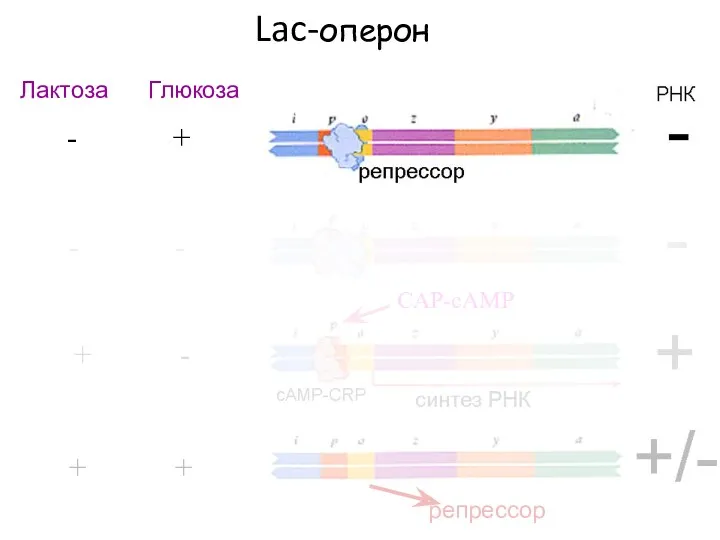
- 15. Lac-оперон фф
- 16. β-галактозидаза 1,024 ак Бета-галактозидаза — фермент, обладающий способностью катализировать расщепление лактозы до глюкозы и галактозы
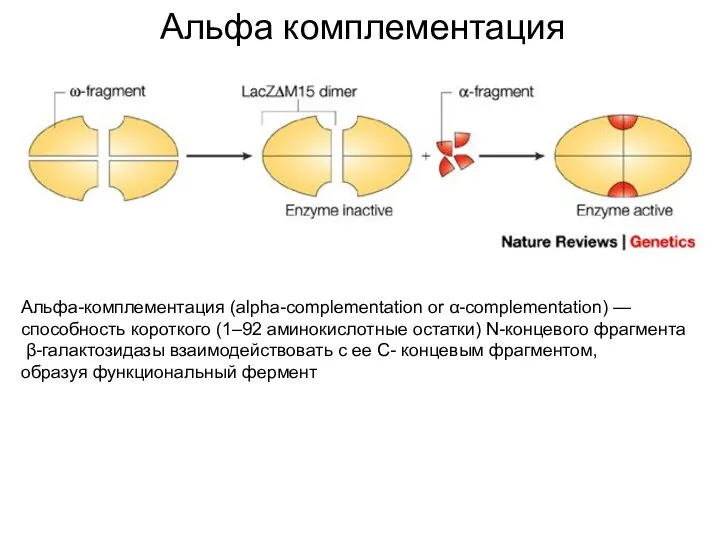
- 17. Альфа комплементация Альфа-комплементация (аlpha-complementation or α-complementation) — способность короткого (1–92 аминокислотные остатки) N-концевого фрагмента β-галактозидазы взаимодействовать
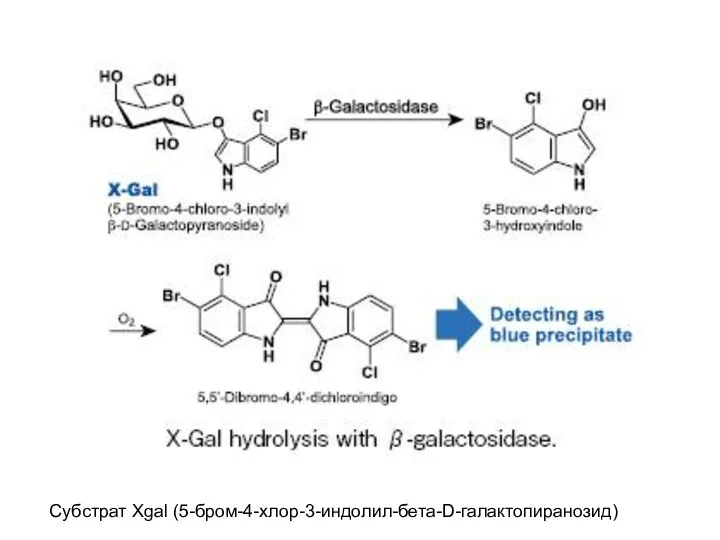
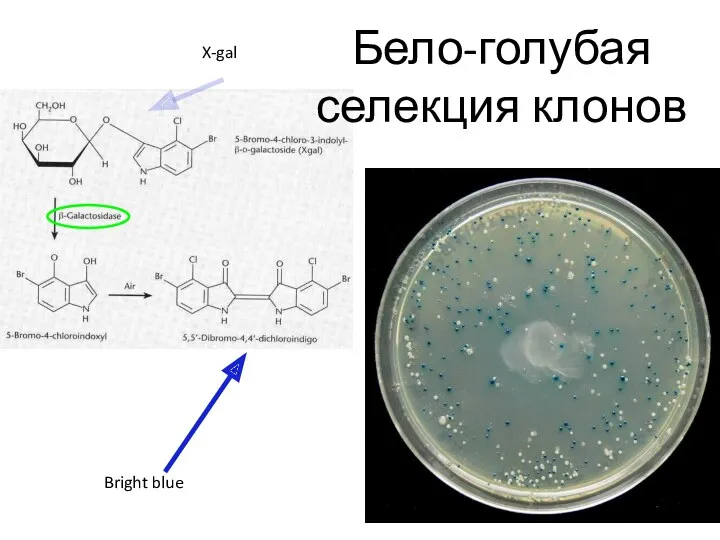
- 18. Субстрат Xgal (5-бром-4-хлор-3-индолил-бета-D-галактопиранозид)
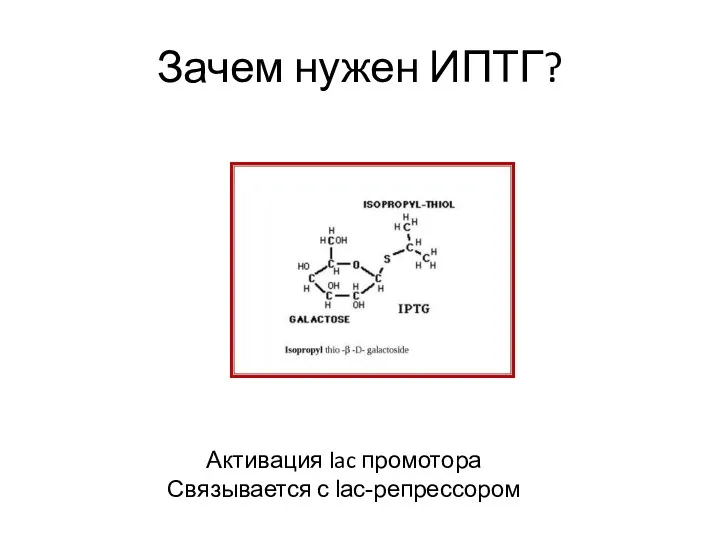
- 19. Зачем нужен ИПТГ? Активация lac промотора Связывается с lac-репрессором
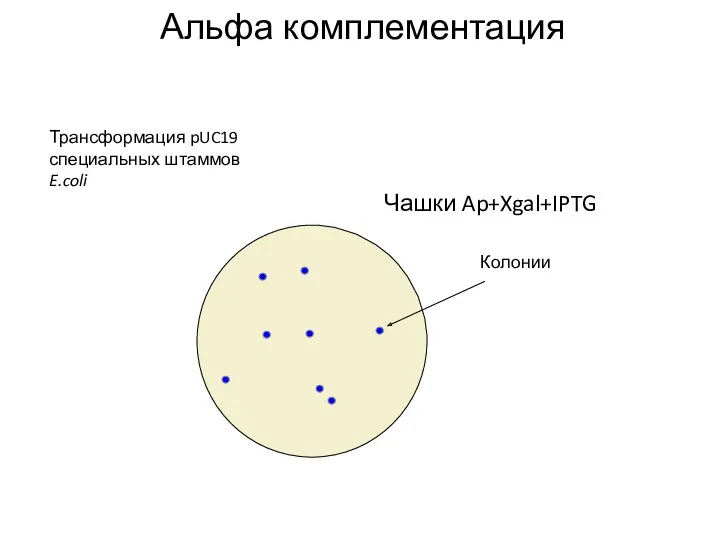
- 20. Трансформация pUC19 специальных штаммов E.coli Колонии Альфа комплементация Чашки Ap+Xgal+IPTG
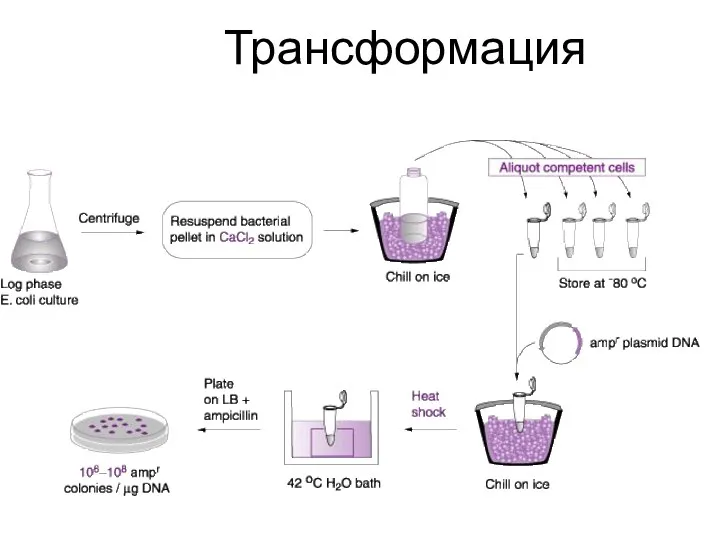
- 22. Трансформация
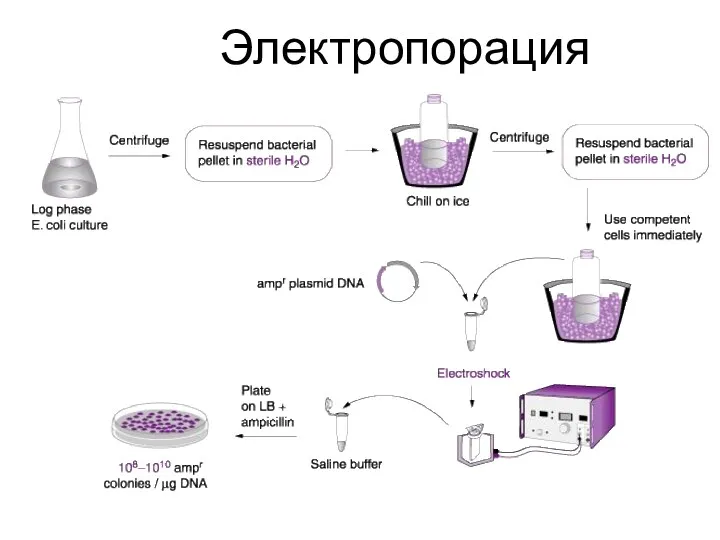
- 23. Электропорация
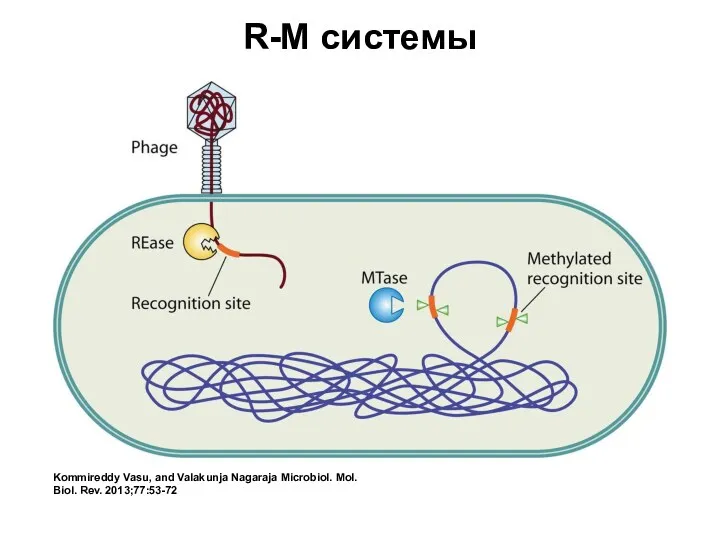
- 24. R-M системы Kommireddy Vasu, and Valakunja Nagaraja Microbiol. Mol. Biol. Rev. 2013;77:53-72
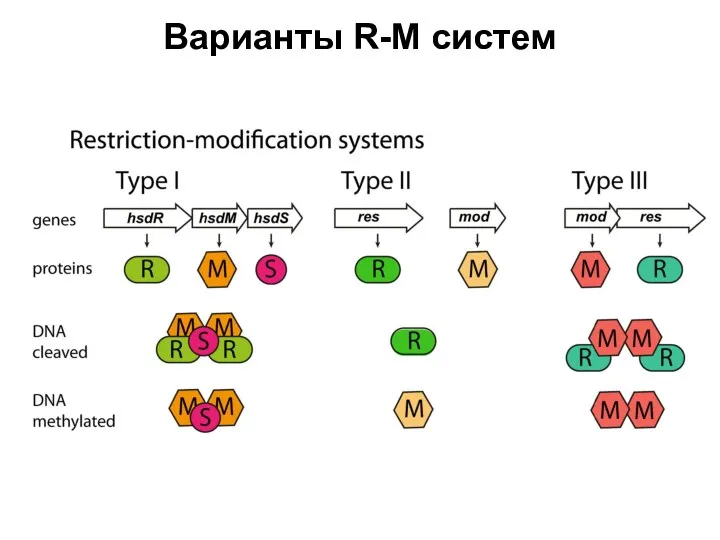
- 25. Варианты R-M систем
- 26. Рестриктазы
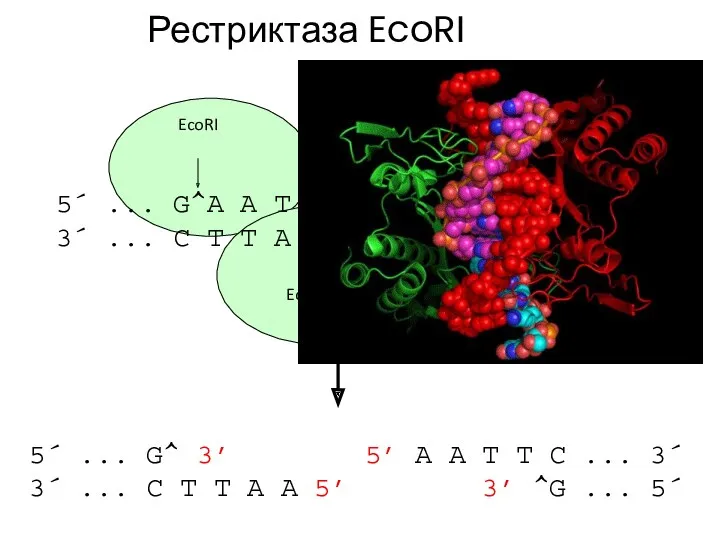
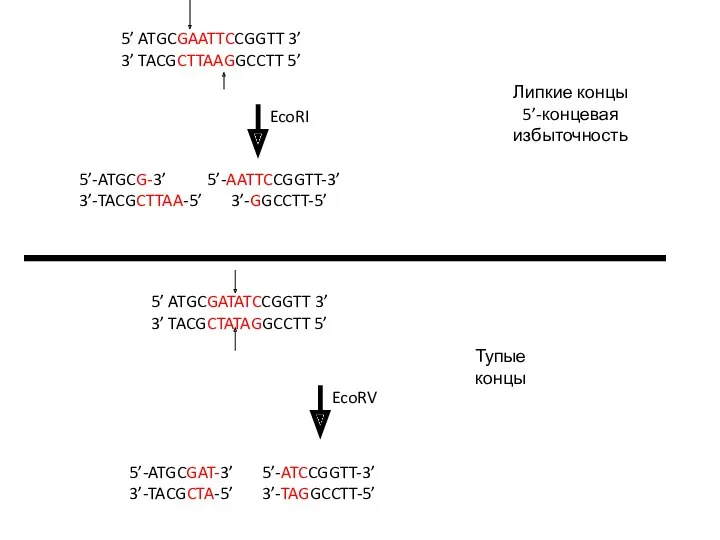
- 27. Рестриктаза EcoRI 5´ ... G^A A T T C ... 3´ 3´ ... C T T
- 28. X. Смит и Д. Натане в 1973 г. предложили номенклатуру рестриктаз Genus+SPecies (strain) EcoRI – Escherichia
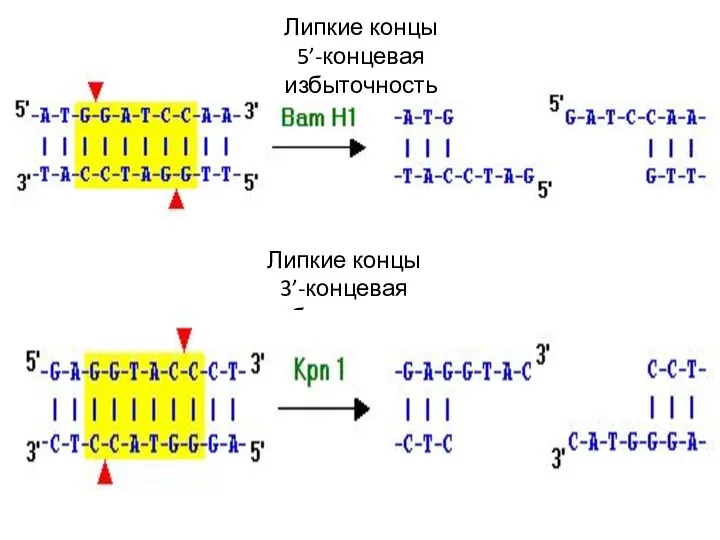
- 29. Липкие концы 5’-концевая избыточность Липкие концы 3’-концевая избыточность
- 30. Липкие концы 5’-концевая избыточность Тупые концы
- 31. 4: AluI 5´ ... AG^CT ... 3´ тупые MspI 5´ ... C^CGG ... 3´ 5’ концевая
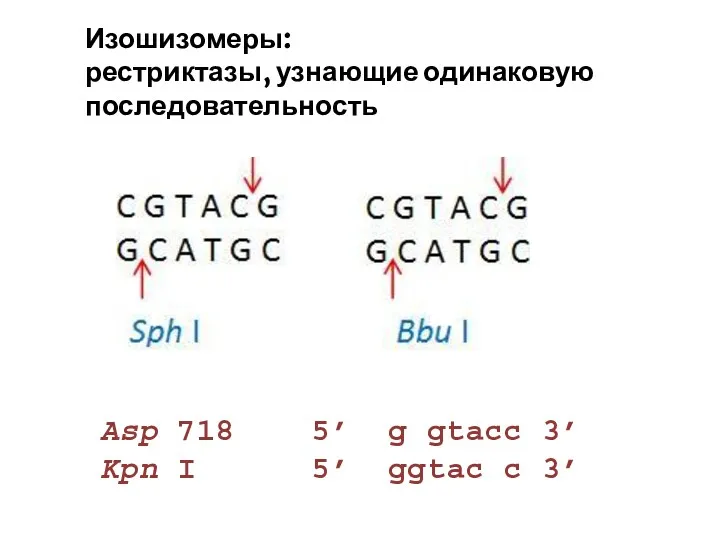
- 32. Изошизомеры: рестриктазы, узнающие одинаковую последовательность Asp 718 5’ g gtacc 3’ Kpn I 5’ ggtac c
- 33. Картирование 1
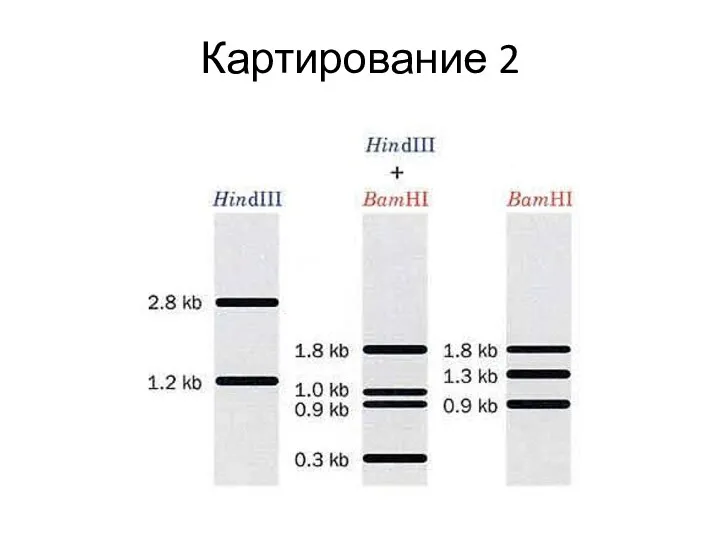
- 34. Картирование 2
- 35. Картирование 3
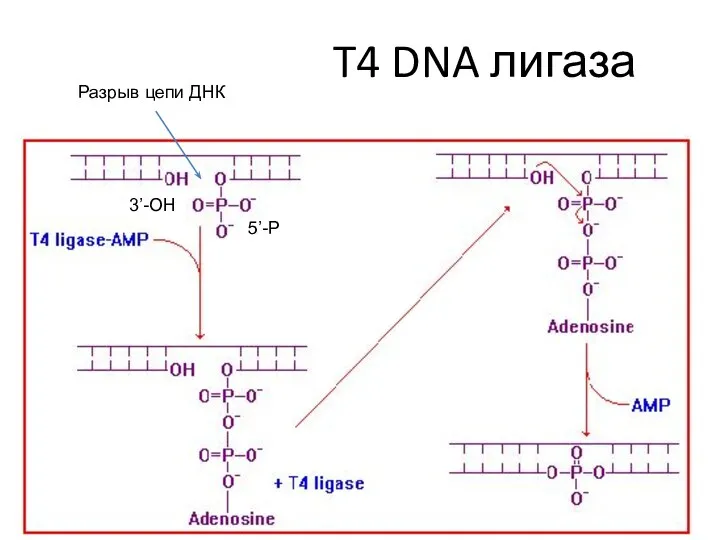
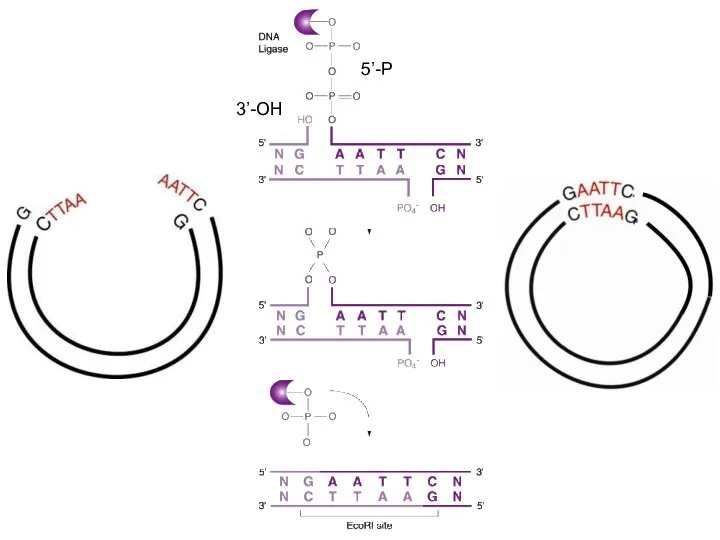
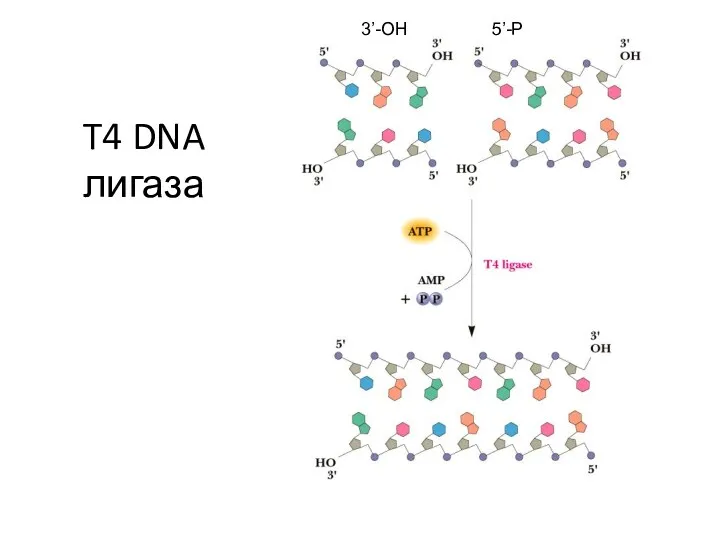
- 37. T4 DNA лигаза 3’-OH 5’-P Разрыв цепи ДНК
- 38. 3’-OH 5’-P
- 39. T4 DNA лигаза 3’-OH 5’-P
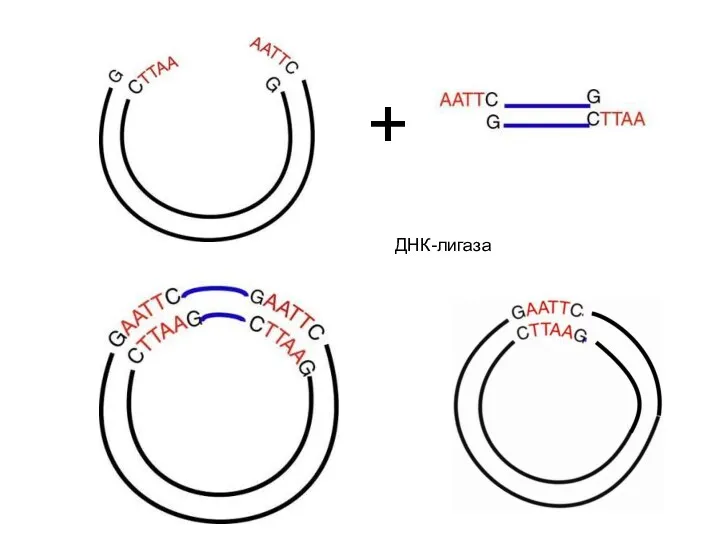
- 40. + ДНК-лигаза
- 41. Бело-голубая селекция клонов
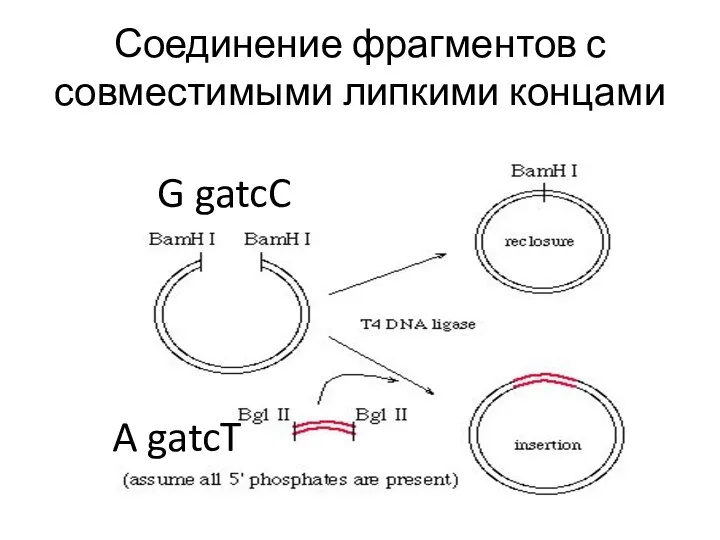
- 42. Соединение фрагментов с совместимыми липкими концами G gatcC A gatcT
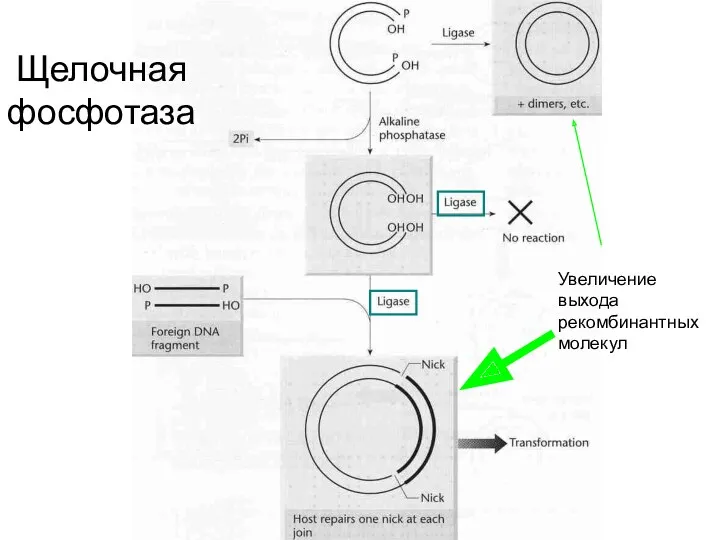
- 43. Щелочная фосфотаза Увеличение выхода рекомбинантных молекул
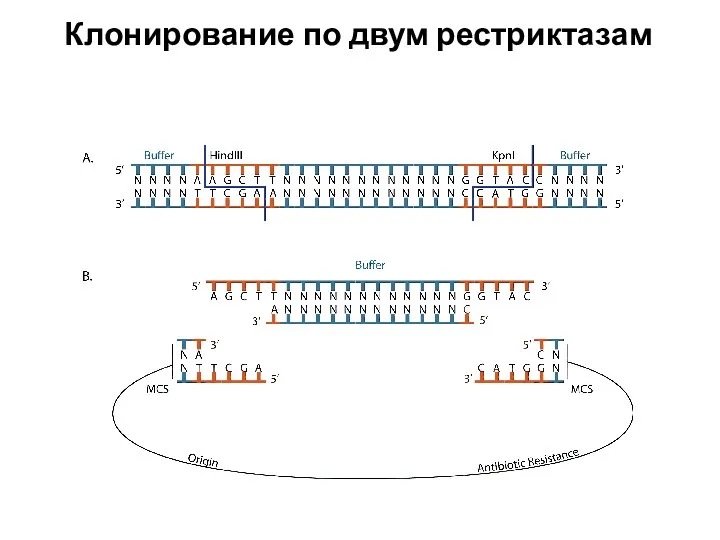
- 44. Клонирование по двум рестриктазам
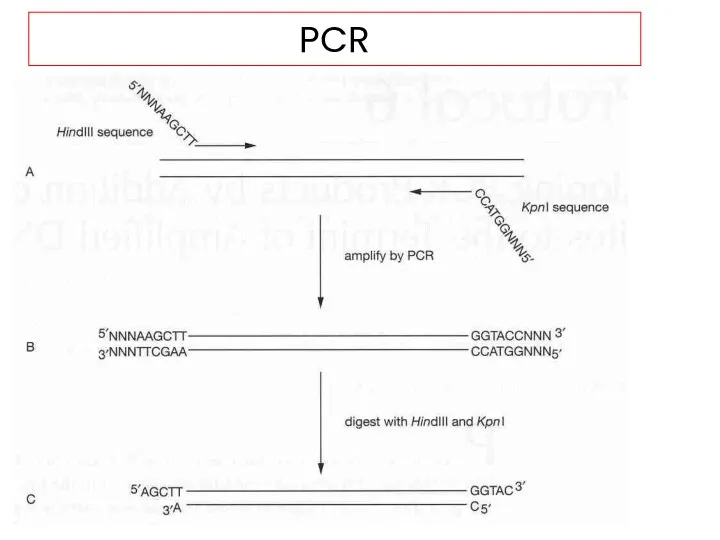
- 45. PCR
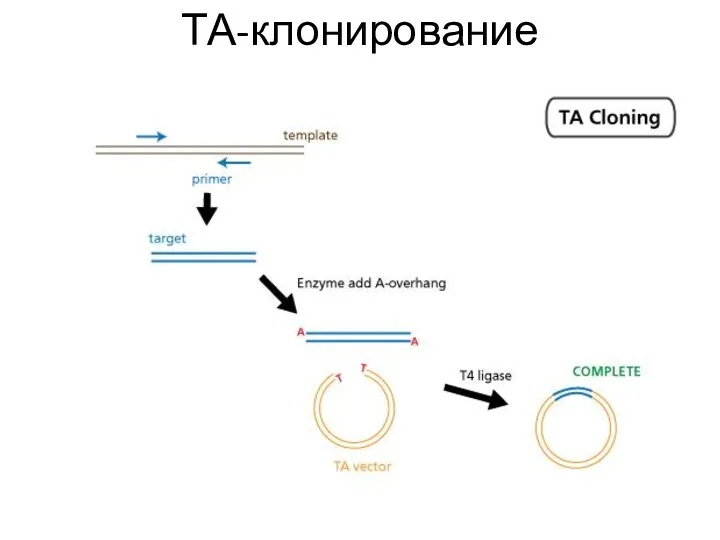
- 46. ТА-клонирование
- 47. Нематричный синтез ДНК ДНК-полимеразами
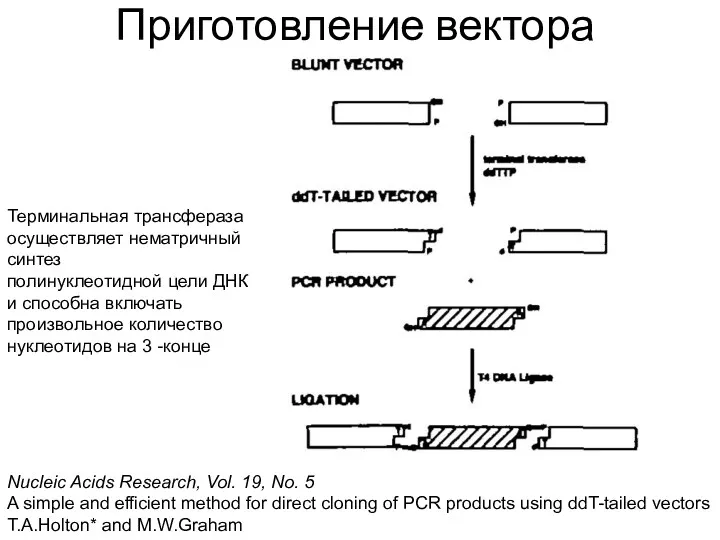
- 48. Приготовление вектора Nucleic Acids Research, Vol. 19, No. 5 A simple and efficient method for direct
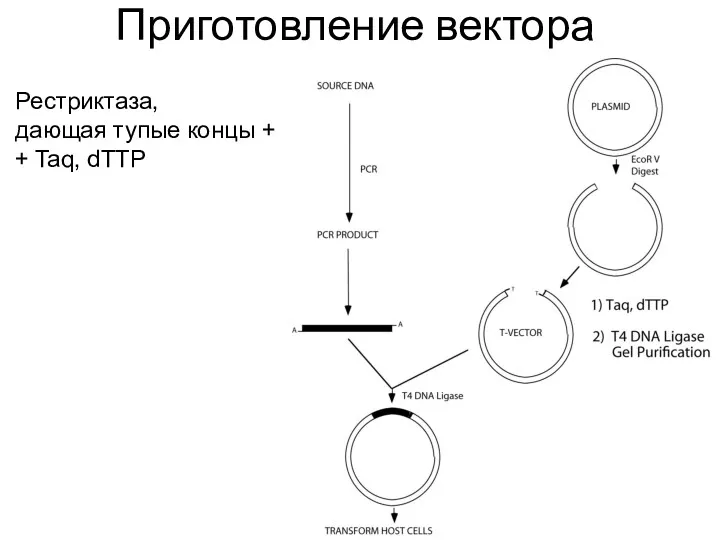
- 49. Приготовление вектора Рестриктаза, дающая тупые концы + + Taq, dTTP
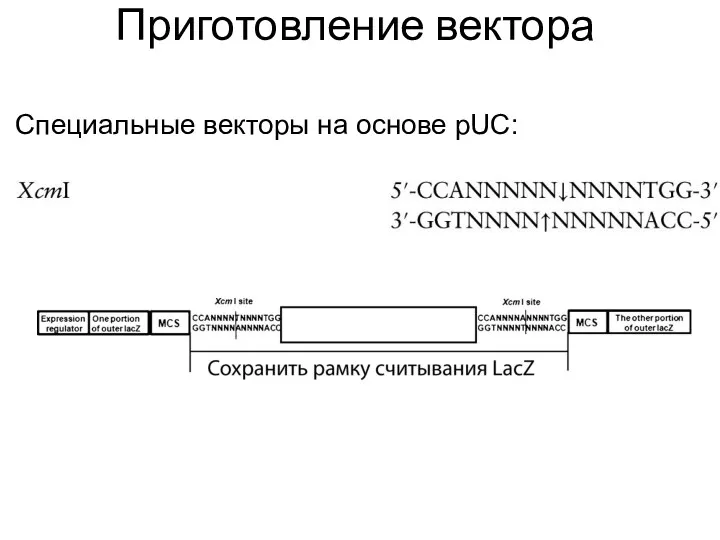
- 50. Приготовление вектора Специальные векторы на основе pUC:
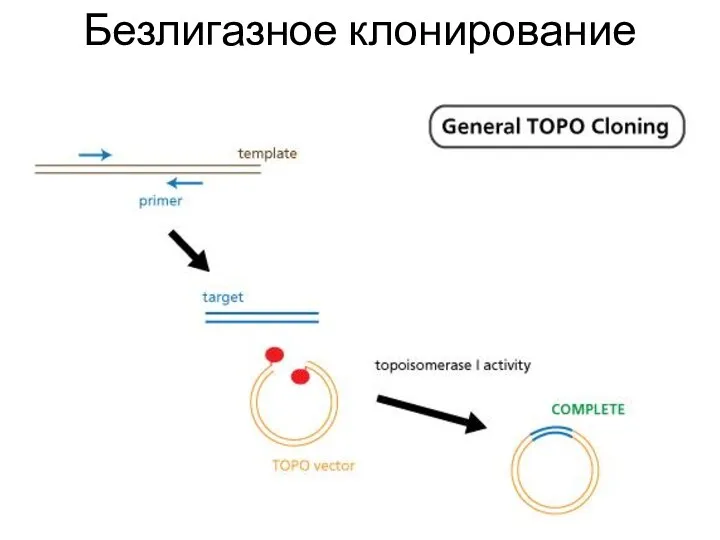
- 51. Безлигазное клонирование
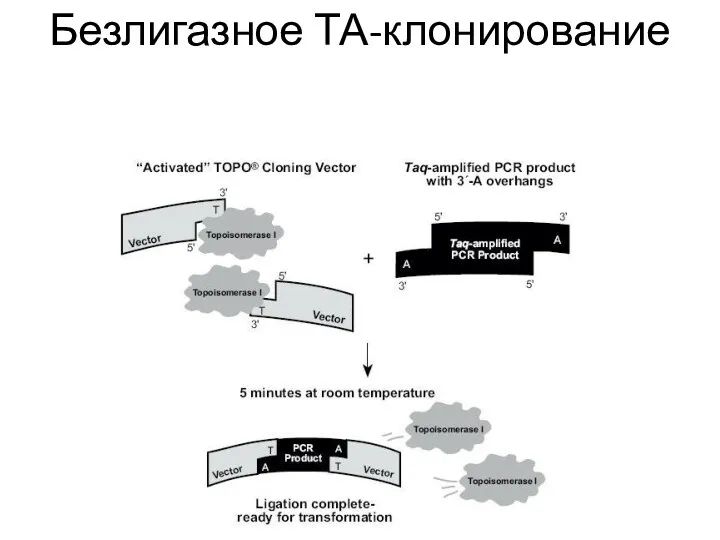
- 52. Безлигазное ТА-клонирование
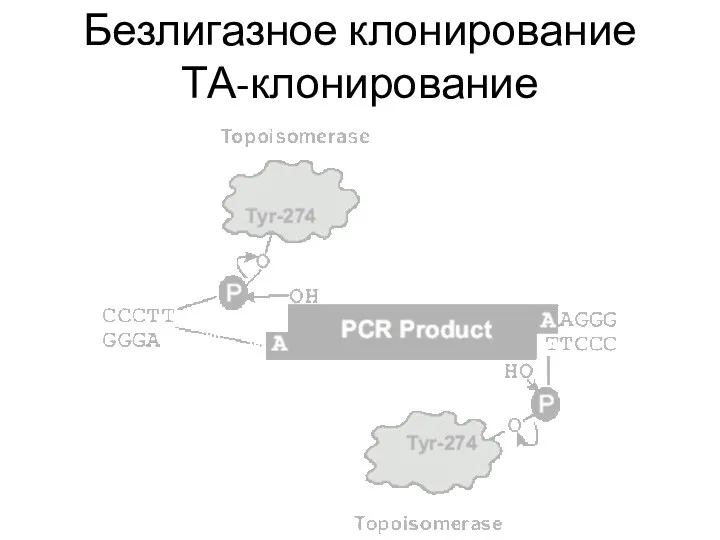
- 53. Безлигазное клонирование ТА-клонирование
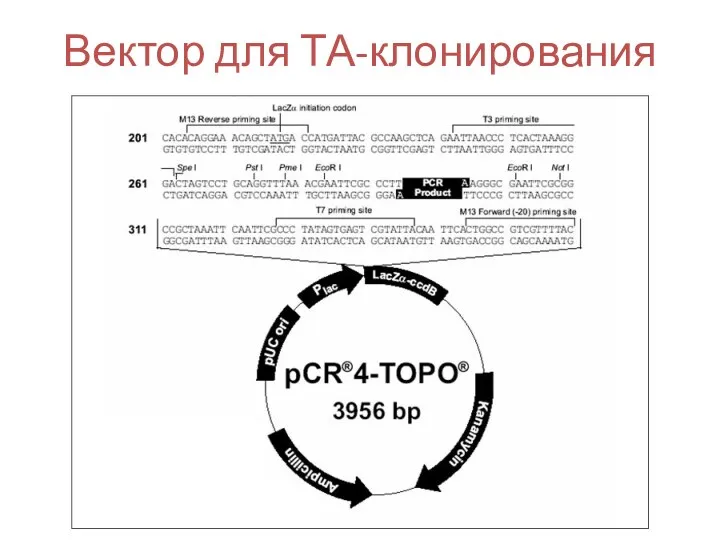
- 54. Вектор для ТА-клонирования
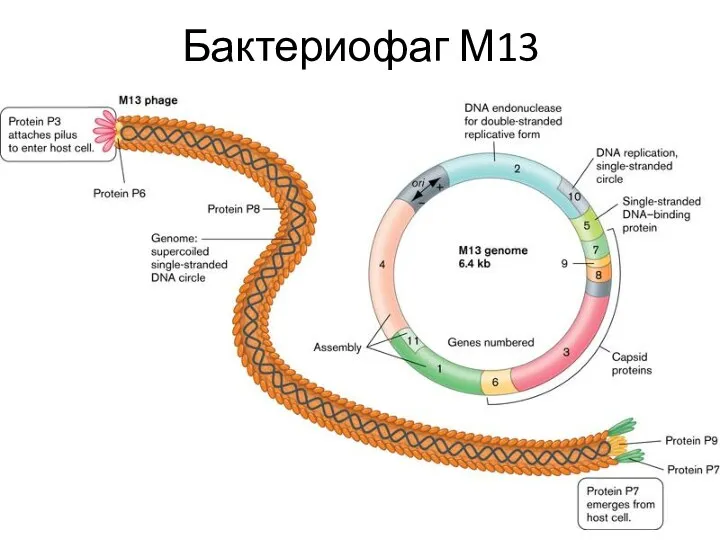
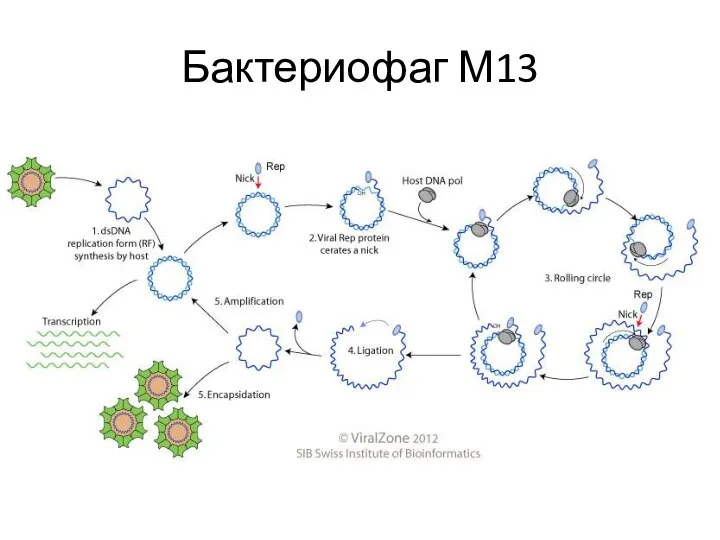
- 55. Бактериофаг М13
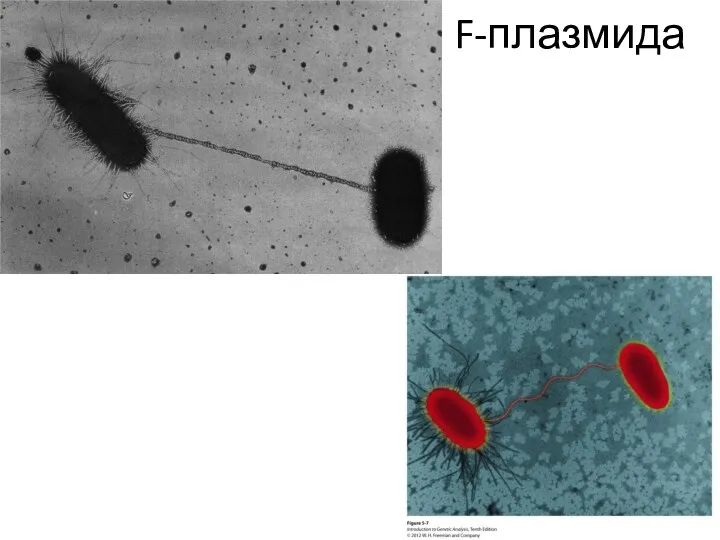
- 57. F-плазмида
- 58. Бактериофаг М13
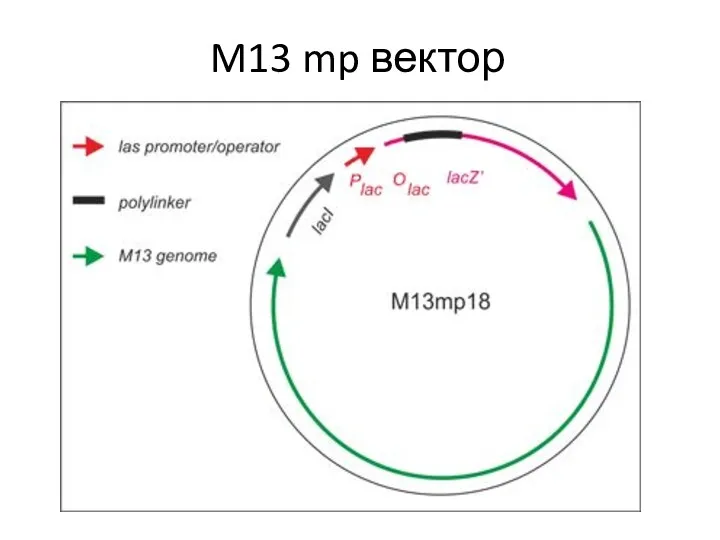
- 59. M13 mp вектор
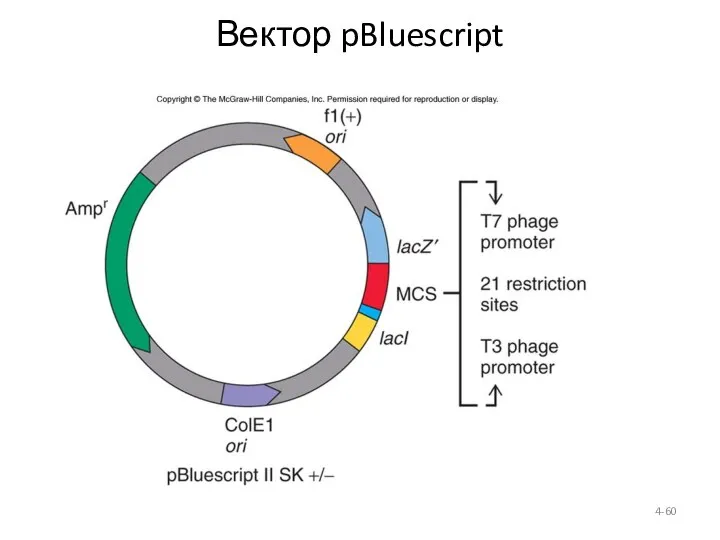
- 60. 4- Вектор pBluescript
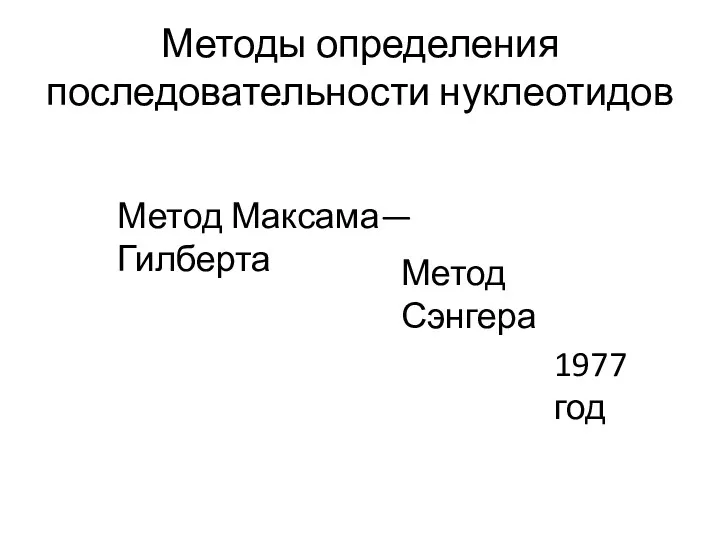
- 61. Методы определения последовательности нуклеотидов Метод Максама—Гилберта Метод Сэнгера 1977 год
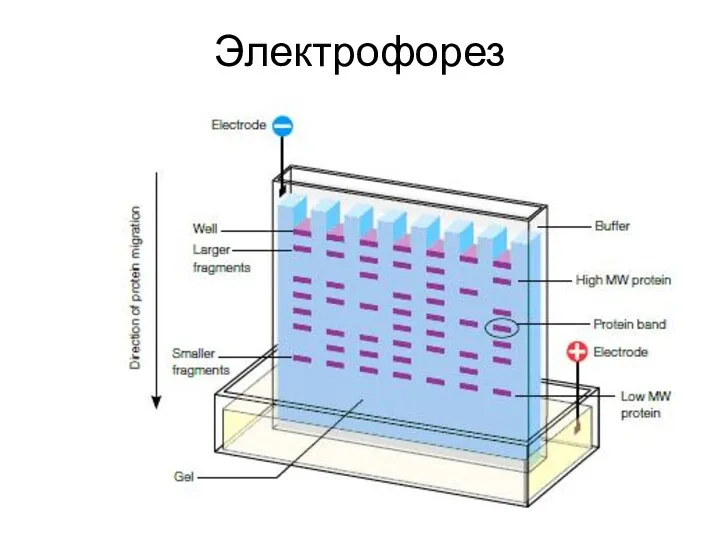
- 62. Электрофорез
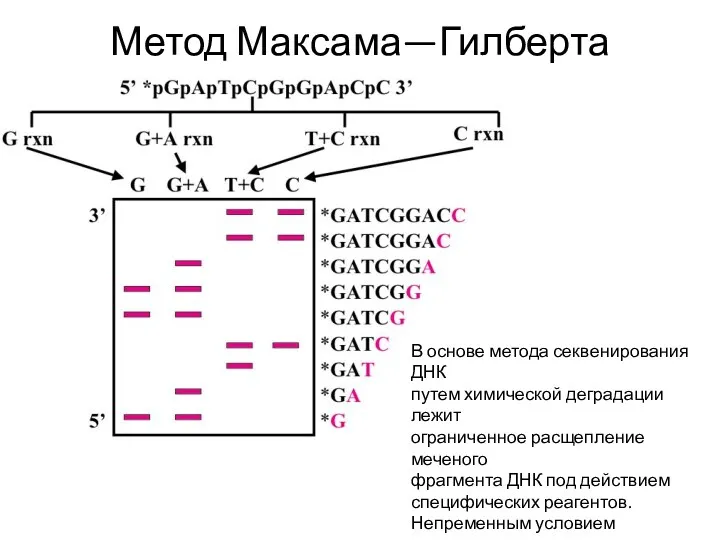
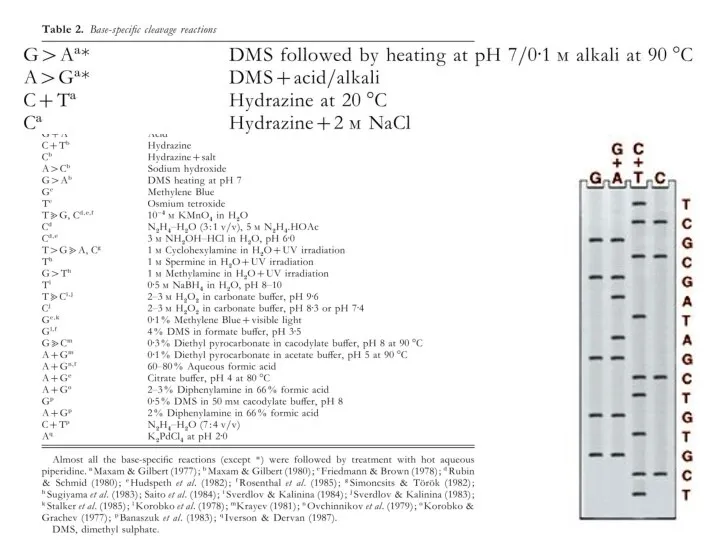
- 63. Метод Максама—Гилберта В основе метода секвенирования ДНК путем химической деградации лежит ограниченное расщепление меченого фрагмента ДНК
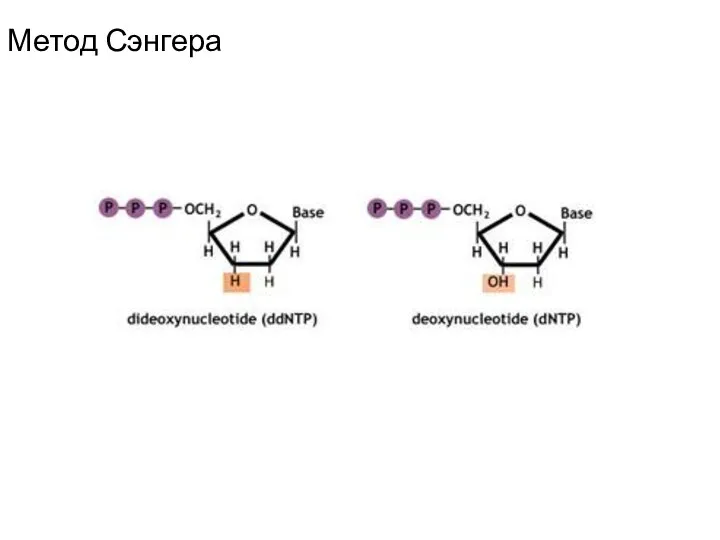
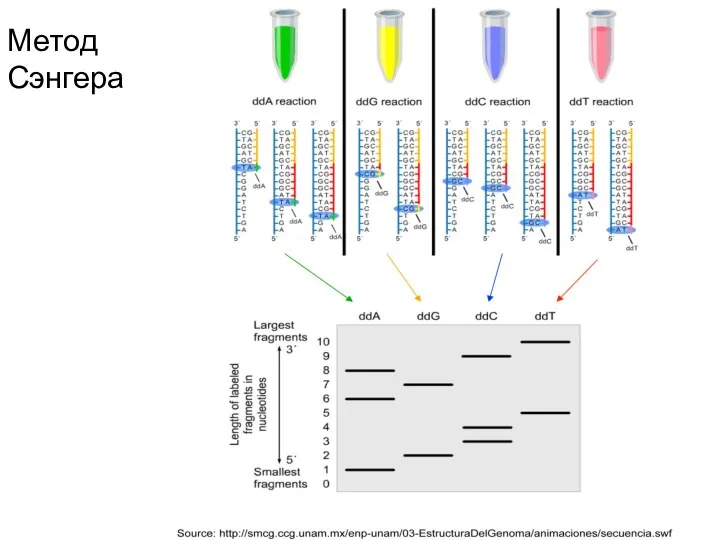
- 65. Метод Сэнгера
- 66. Метод Сэнгера
- 67. Метод Сэнгера AGCT
- 68. Для разделения фрагментов ДНК с разрешением в 1 нуклеотид используют: 8–10% полиакриламидный гель 7М мочевина Температура
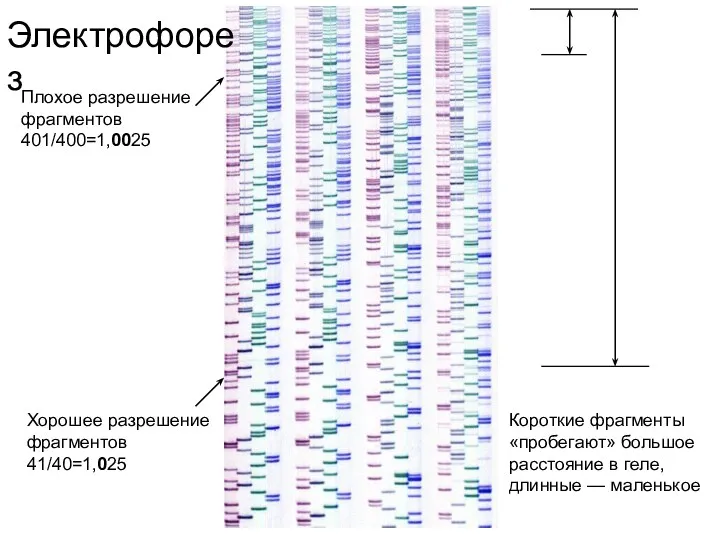
- 69. Электрофорез Хорошее разрешение фрагментов 41/40=1,025 Плохое разрешение фрагментов 401/400=1,0025 Короткие фрагменты «пробегают» большое расстояние в геле,
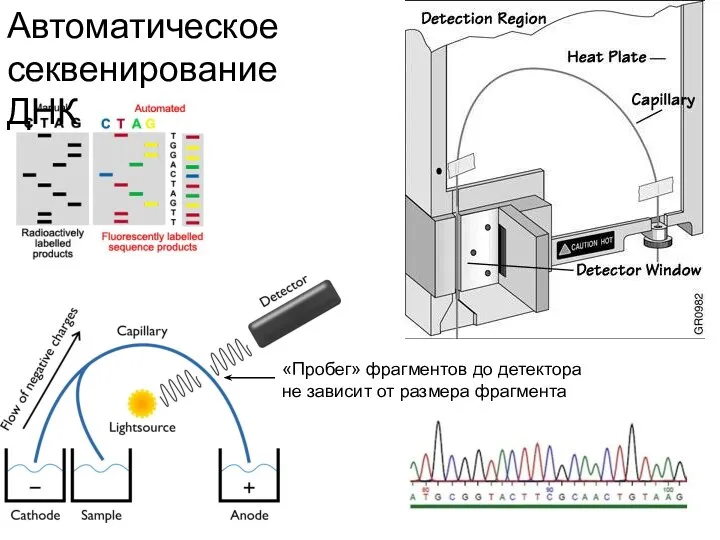
- 71. Автоматическое секвенирование ДНК
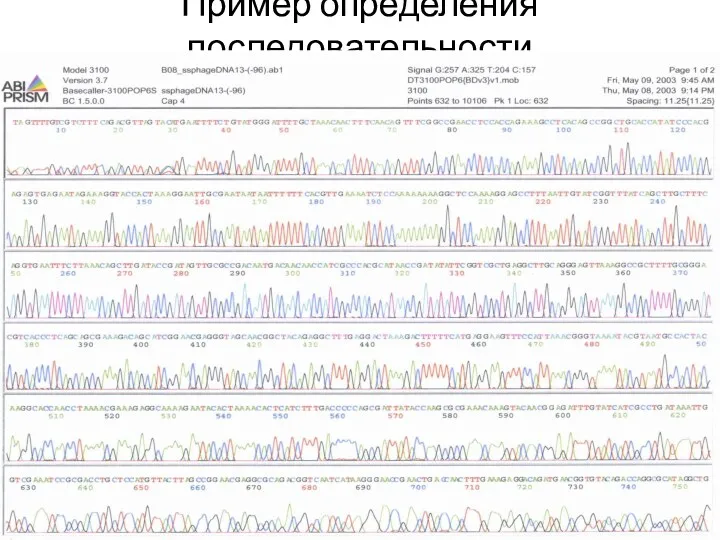
- 72. Пример определения последовательности
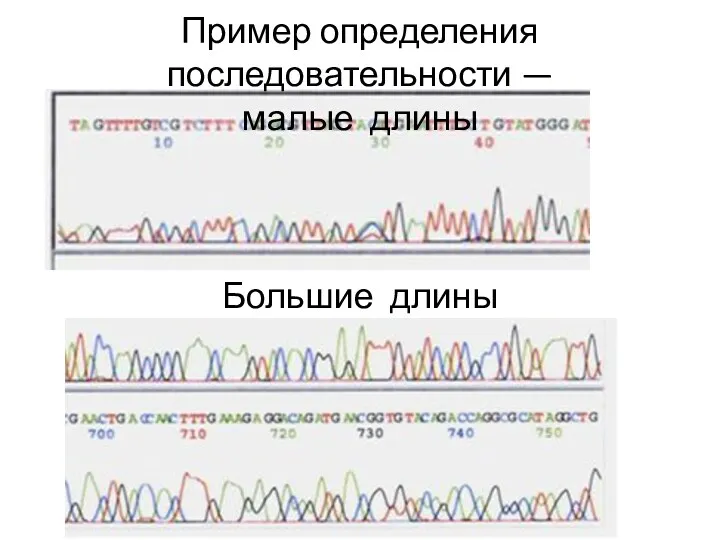
- 73. Пример определения последовательности — малые длины
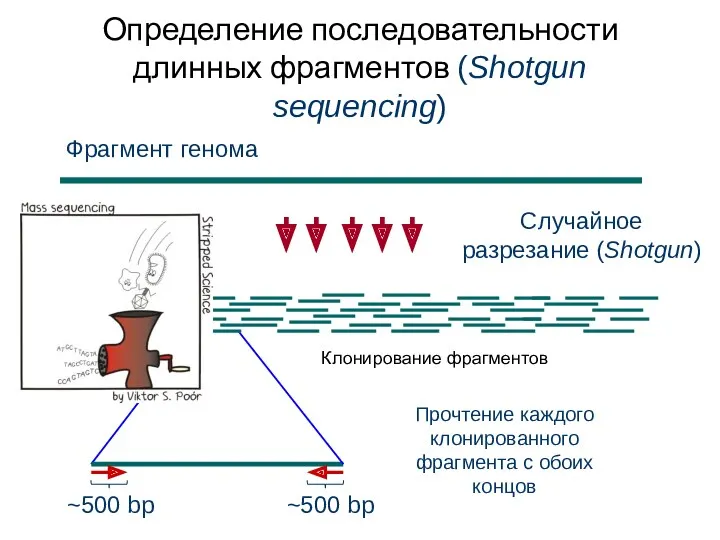
- 74. Определение последовательности длинных фрагментов (Shotgun sequencing) Случайное разрезание (Shotgun) Фрагмент генома Прочтение каждого клонированного фрагмента с
- 75. Реконструирование последовательности (Fragment Assembly) Необходимо 7–10 кратное прочтение каждого участка reads
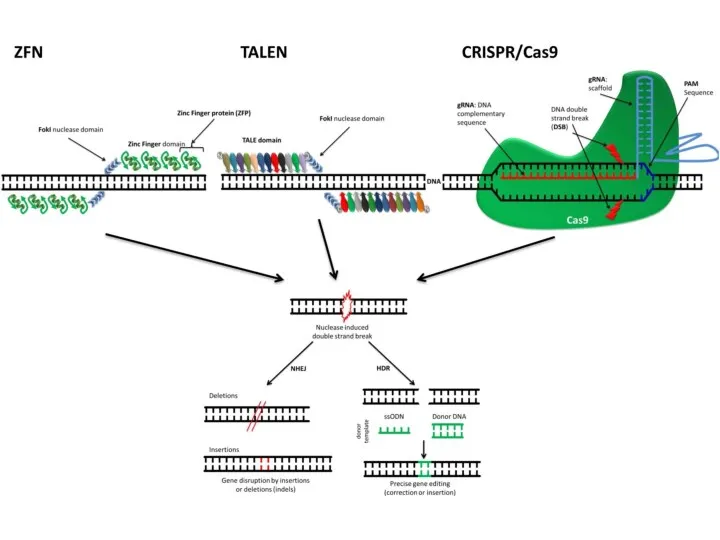
- 76. Редактирование генома
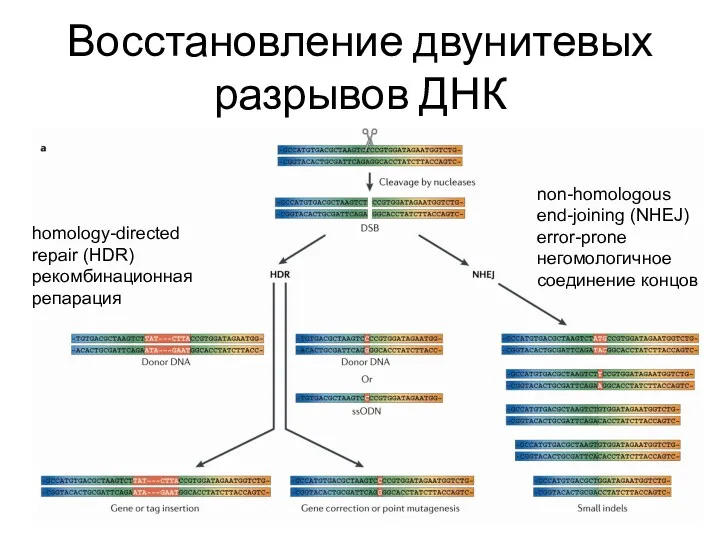
- 77. Восстановление двунитевых разрывов ДНК homology-directed repair (HDR) рекомбинационная репарация non-homologous end-joining (NHEJ) error-prone негомологичное соединение концов
- 78. 4L = 109 (размер генома человека) log4L = log109 2L log2 = 9 L = 15
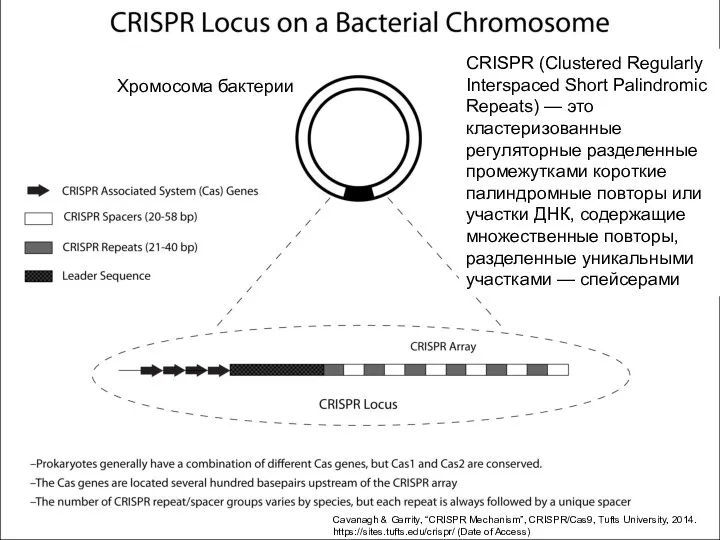
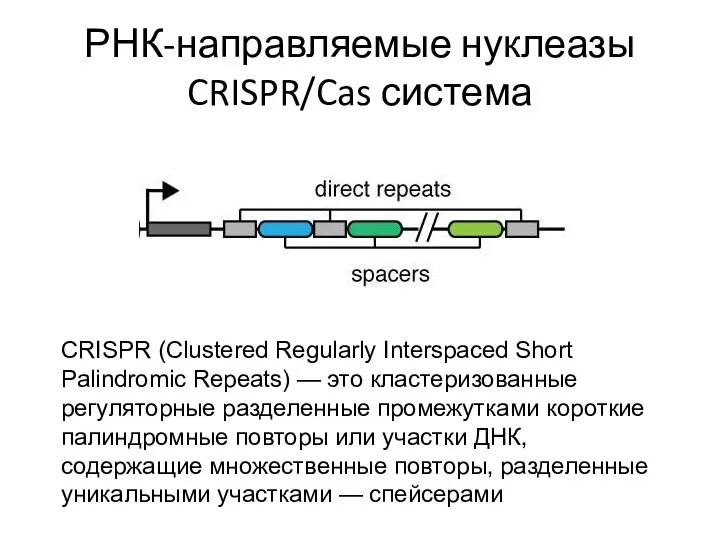
- 80. CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats) — это кластеризованные регуляторные разделенные промежутками короткие палиндромные повторы
- 81. РНК-направляемые нуклеазы CRISPR/Cas система CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats) — это кластеризованные регуляторные разделенные
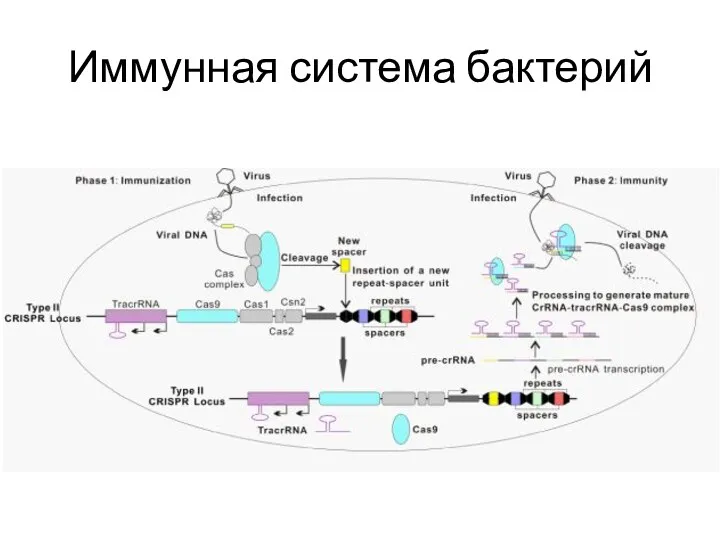
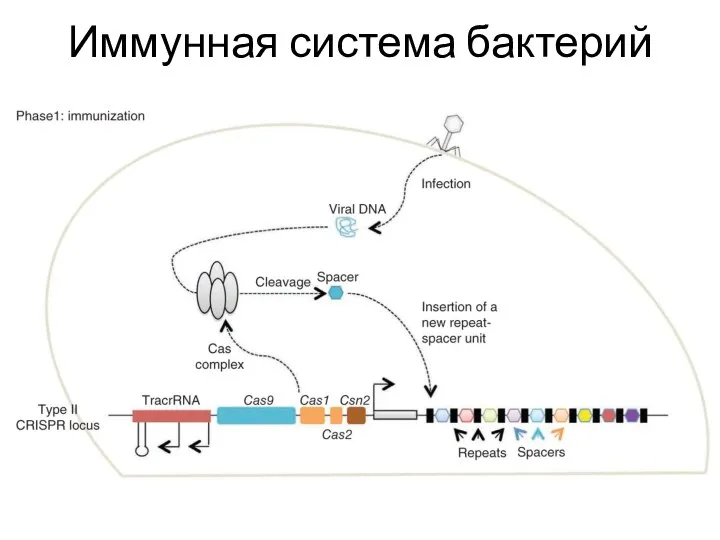
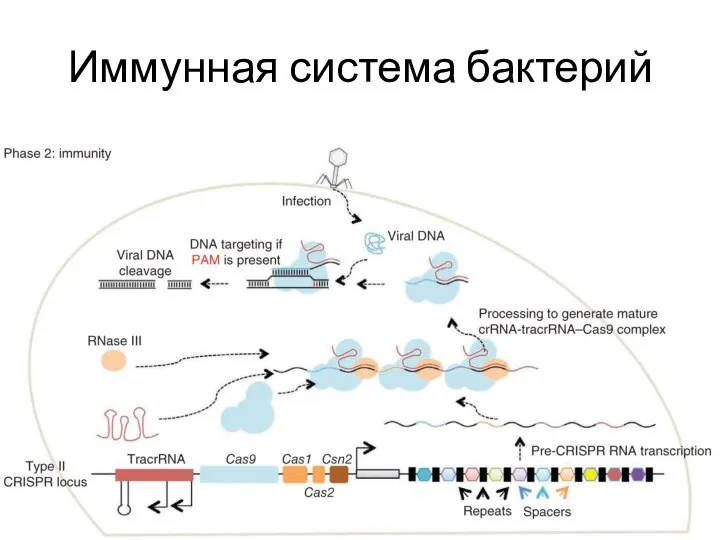
- 82. Иммунная система бактерий
- 83. Иммунная система бактерий
- 84. Иммунная система бактерий
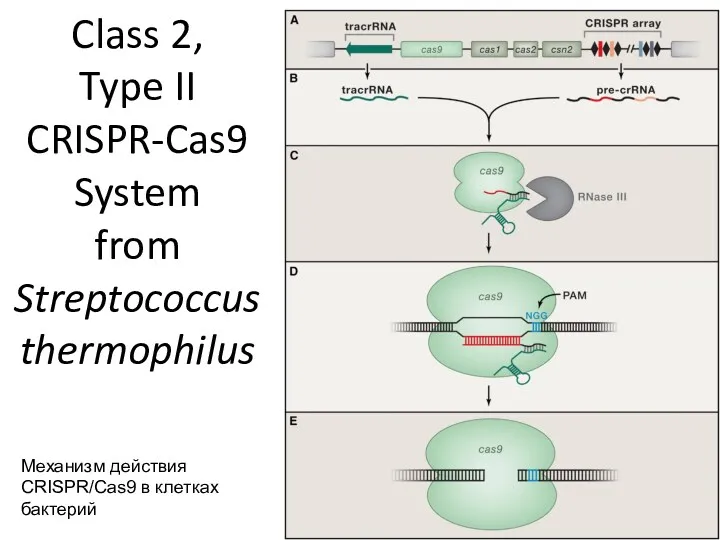
- 85. Class 2, Type II CRISPR-Cas9 System from Streptococcus thermophilus Механизм действия CRISPR/Cas9 в клетках бактерий
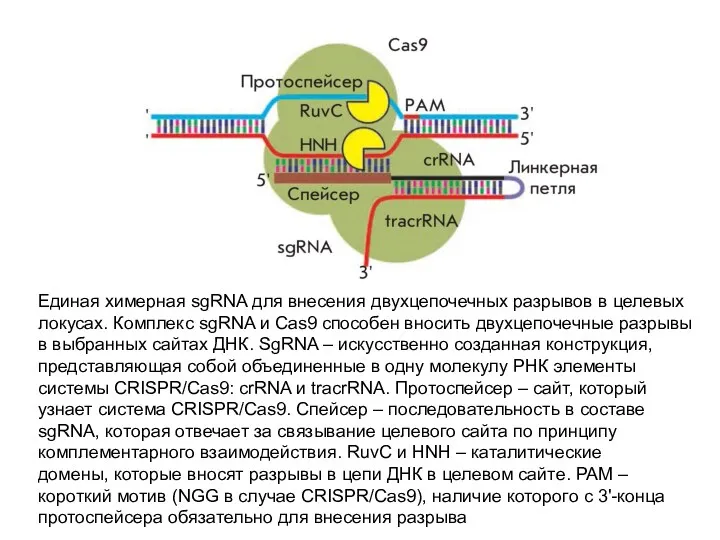
- 88. Единая химерная sgRNA для внесения двухцепочечных разрывов в целевых локусах. Комплекс sgRNA и Cas9 способен вносить
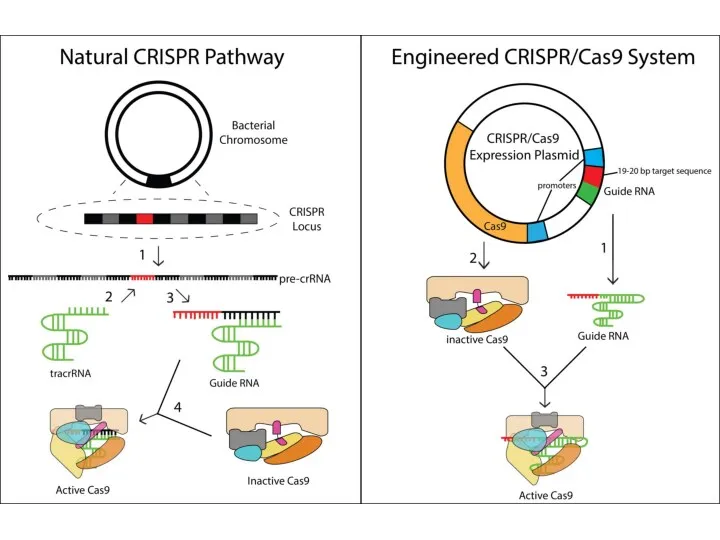
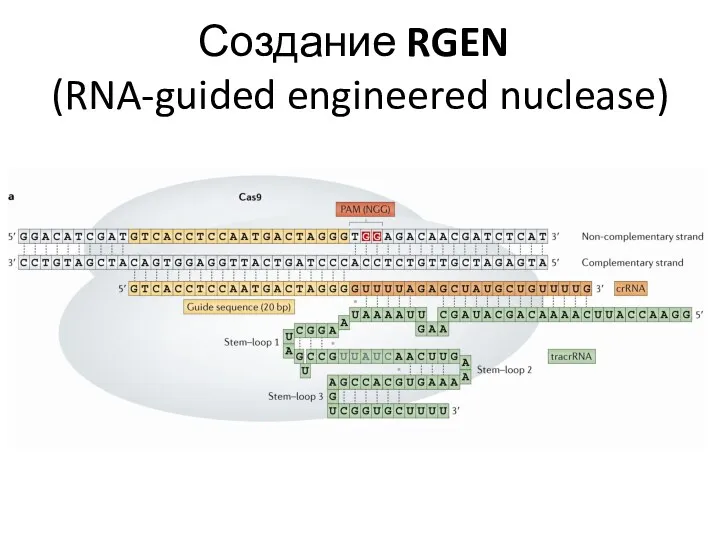
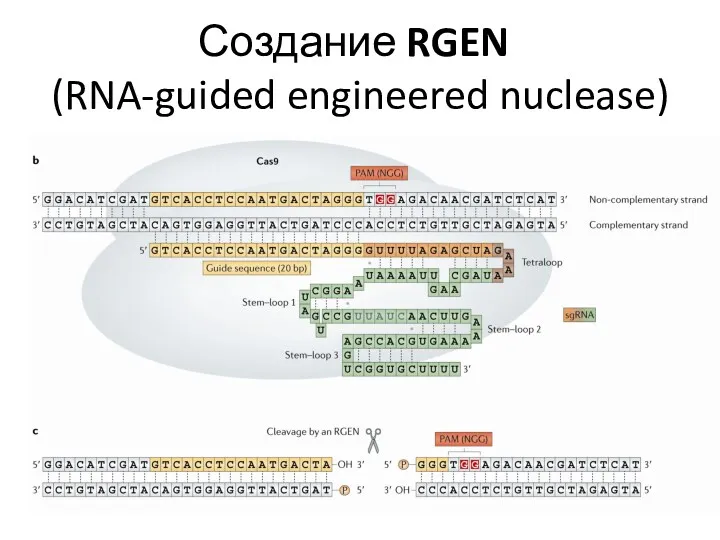
- 89. Создание RGEN (RNA-guided engineered nuclease)
- 90. Создание RGEN (RNA-guided engineered nuclease)
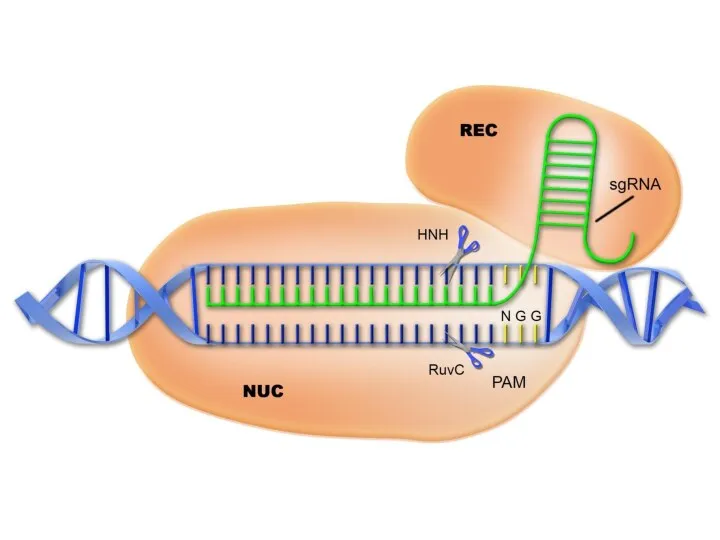
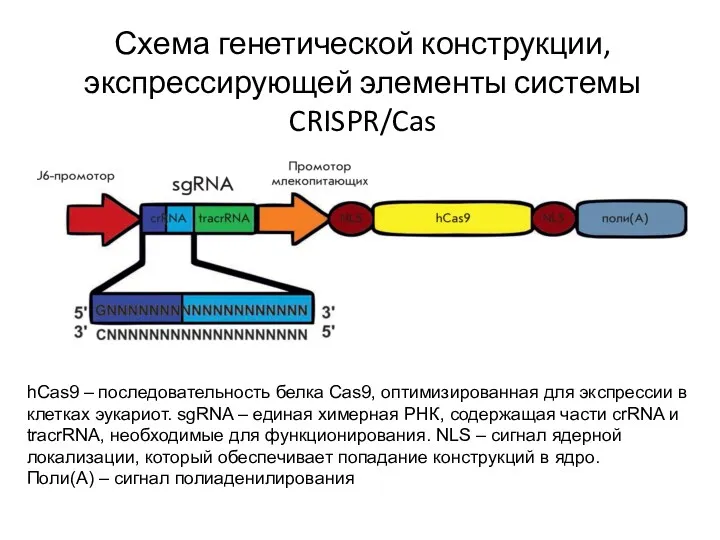
- 91. hCas9 – последовательность белка Cas9, оптимизированная для экспрессии в клетках эукариот. sgRNA – единая химерная РНК,
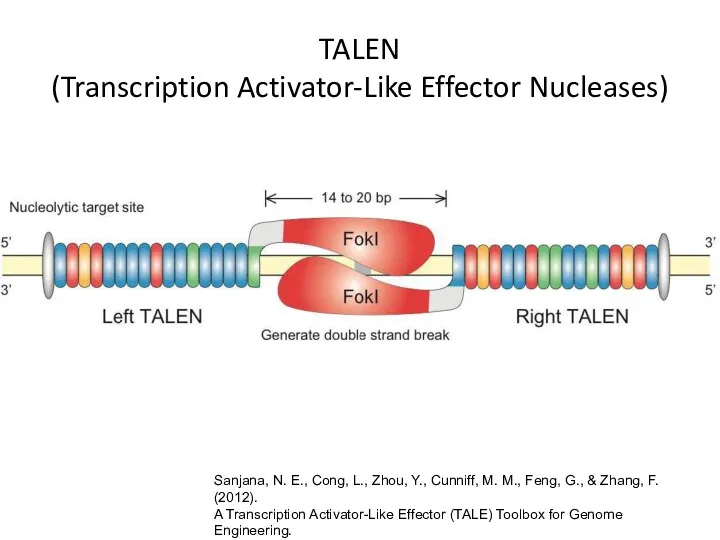
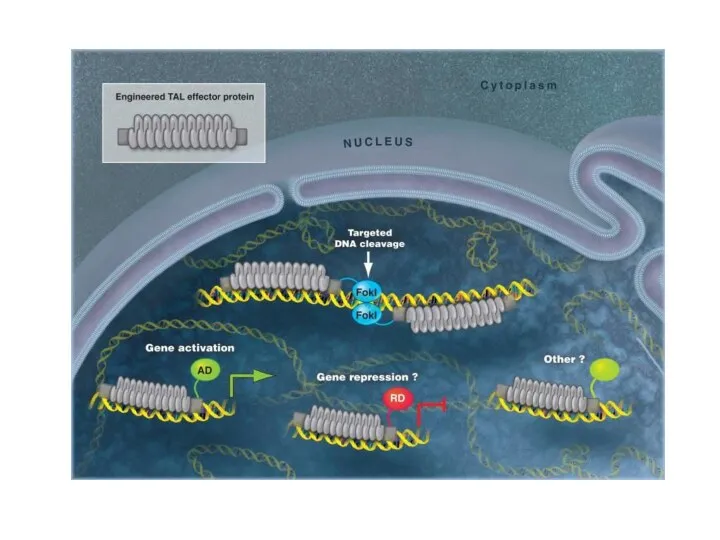
- 92. TALEN (Transcription Activator-Like Effector Nucleases; эффекторные нуклеазы, подобные активаторам транскрипции)
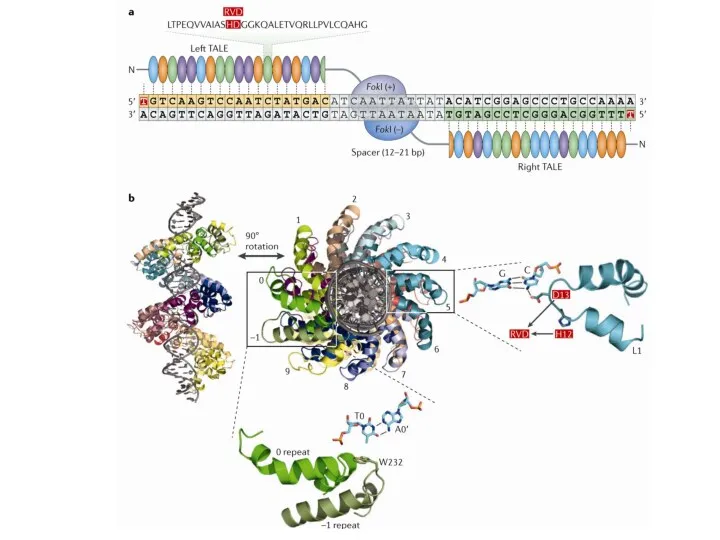
- 93. Transcription Activator-Like Effectors, TALE TALE белки были обнаружены у бактерий рода Xanthomonas поражающих рис, перец, хлопок
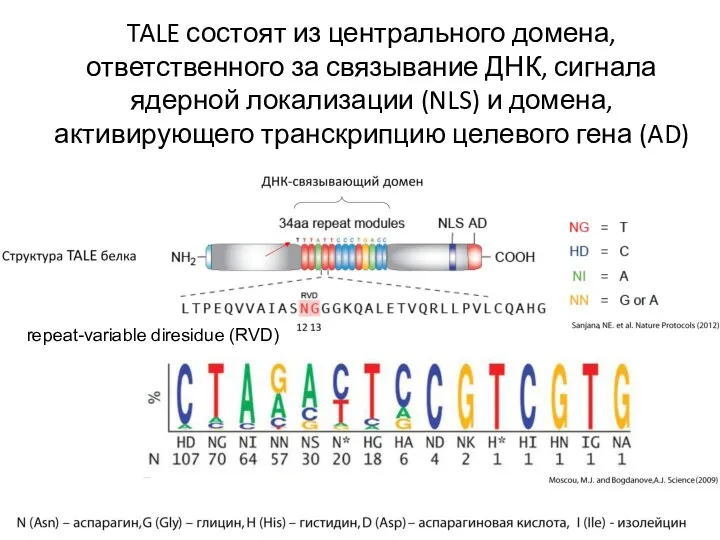
- 94. TALE состоят из центрального домена, ответственного за связывание ДНК, сигнала ядерной локализации (NLS) и домена, активирующего
- 95. TALEN (Transcription Activator-Like Effector Nucleases) Sanjana, N. E., Cong, L., Zhou, Y., Cunniff, M. M., Feng,
- 98. Транскрипционные факторы Транскрипционные факторы содержат следующие домены: ДНК-связывающий домен (DBD) — взаимодействует со специфичными последовательностями ДНК,
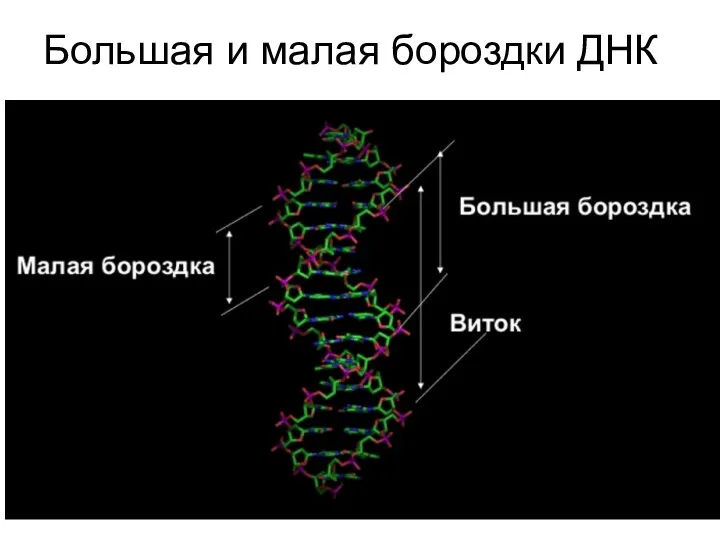
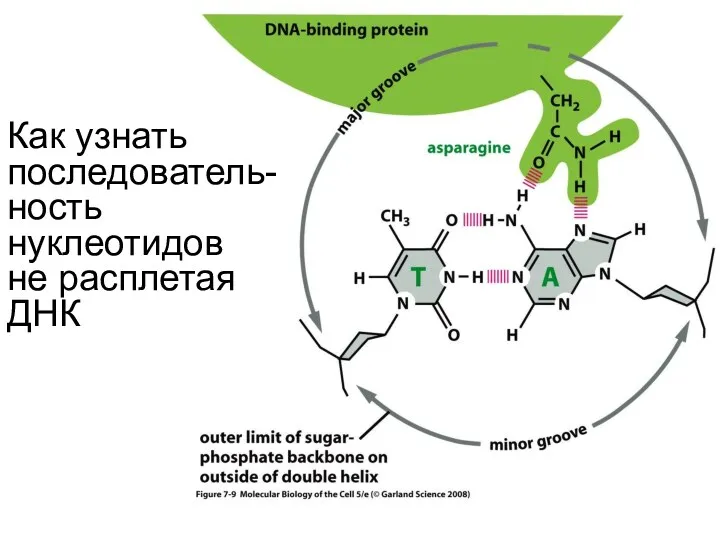
- 99. Большая и малая бороздки ДНК
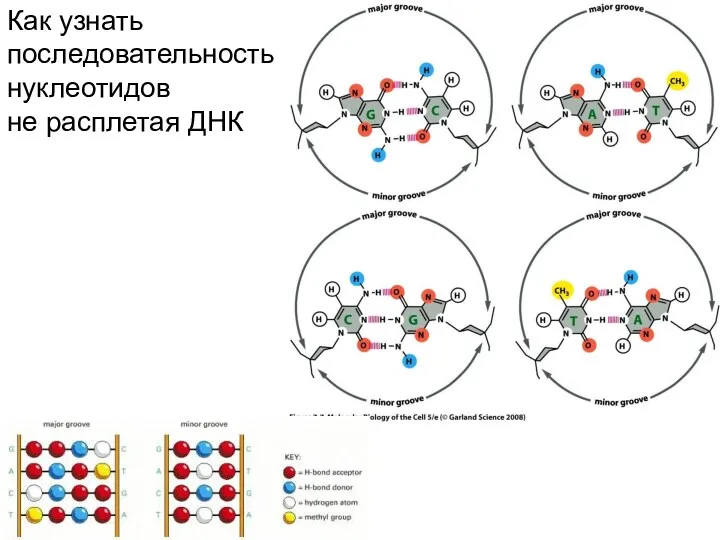
- 100. Как узнать последовательность нуклеотидов не расплетая ДНК
- 101. Как узнать последователь-ность нуклеотидов не расплетая ДНК
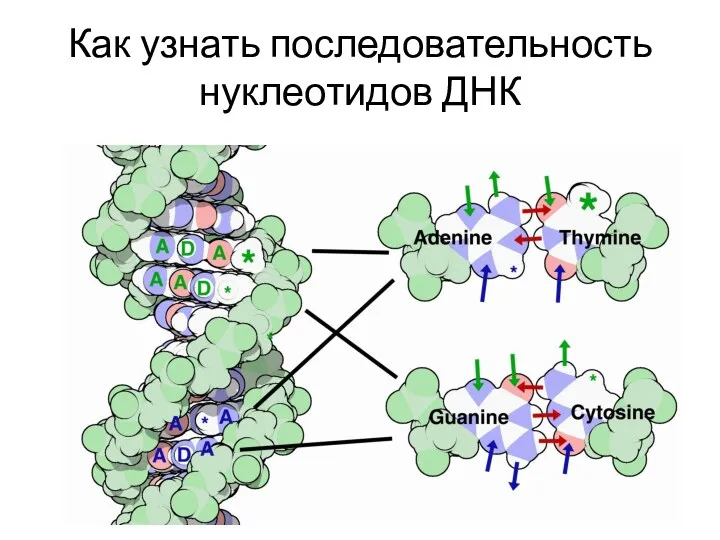
- 102. Как узнать последовательность нуклеотидов ДНК
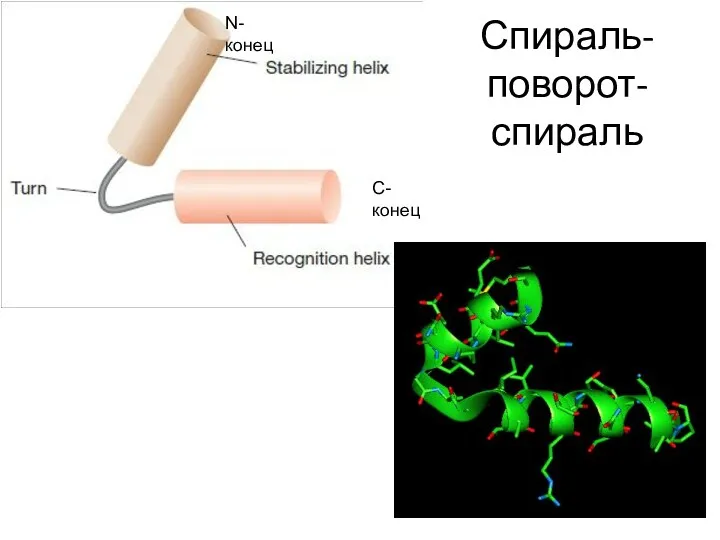
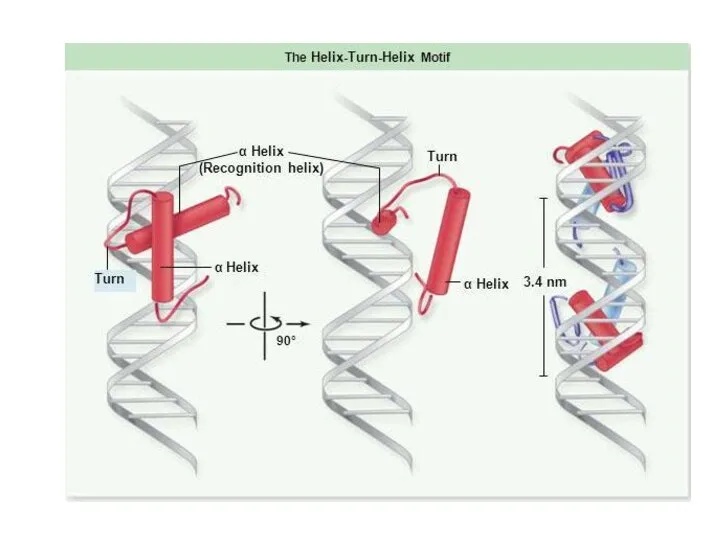
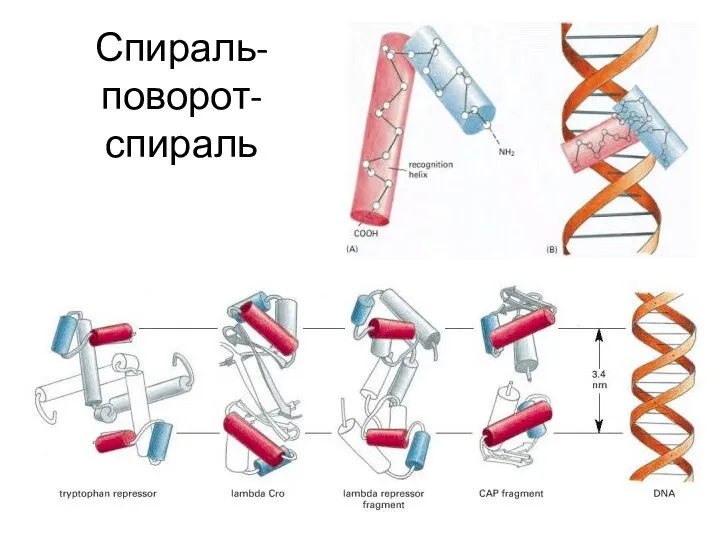
- 103. Спираль-поворот-спираль
- 104. Спираль-поворот-спираль N-конец С-конец
- 106. Спираль-поворот-спираль
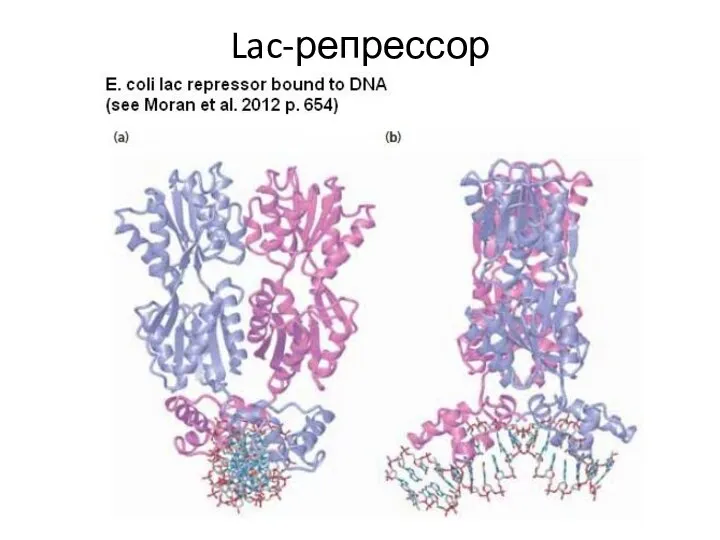
- 107. Lac-репрессор
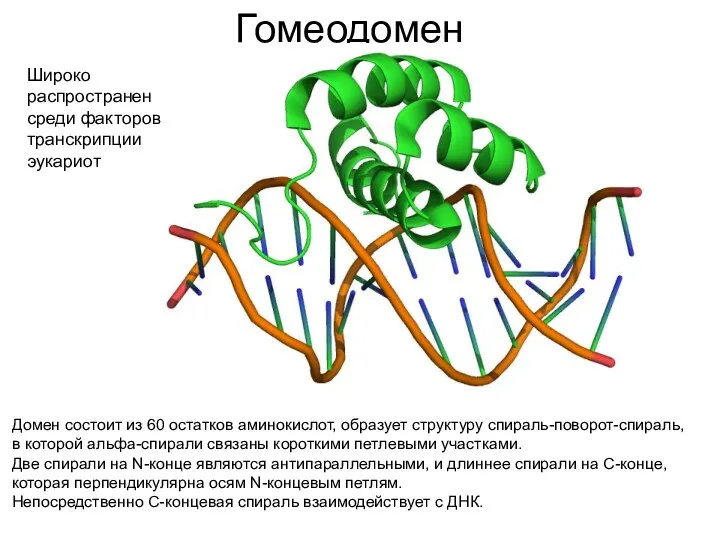
- 108. Гомеодомен Домен состоит из 60 остатков аминокислот, образует структуру спираль-поворот-спираль, в которой альфа-спирали связаны короткими петлевыми
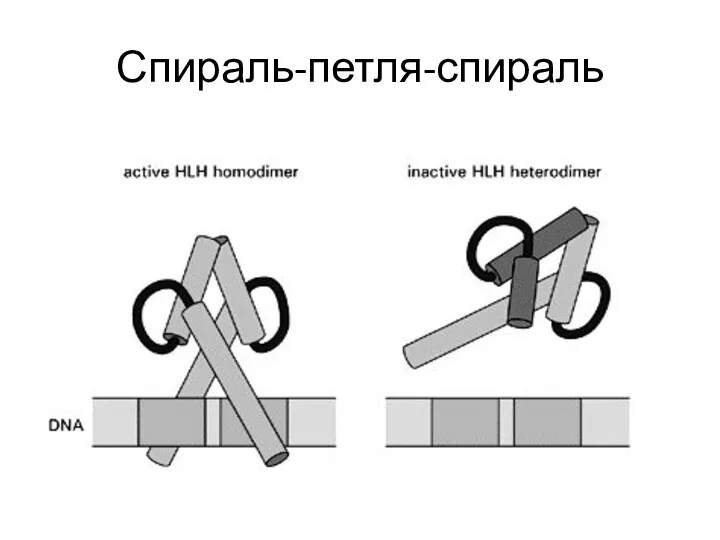
- 109. Спираль-петля-спираль
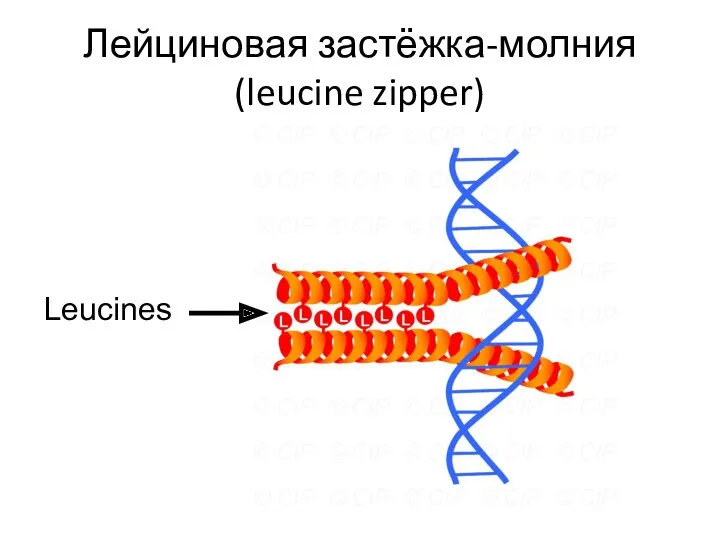
- 110. Лейциновая застёжка-молния (leucine zipper) Leucines
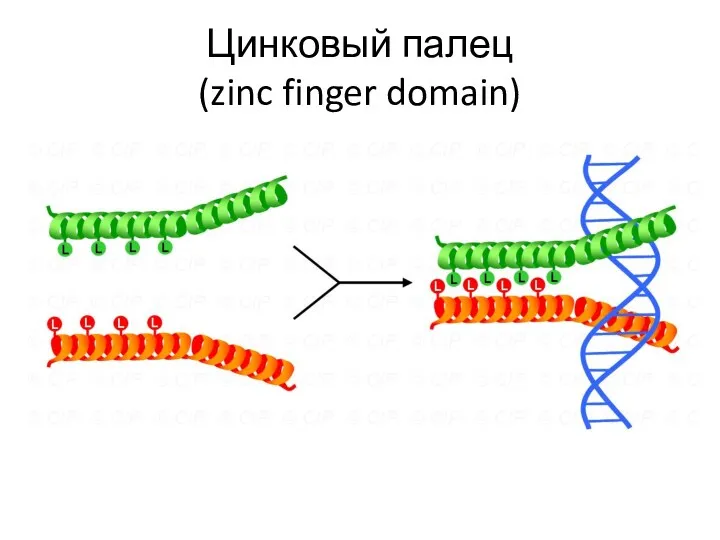
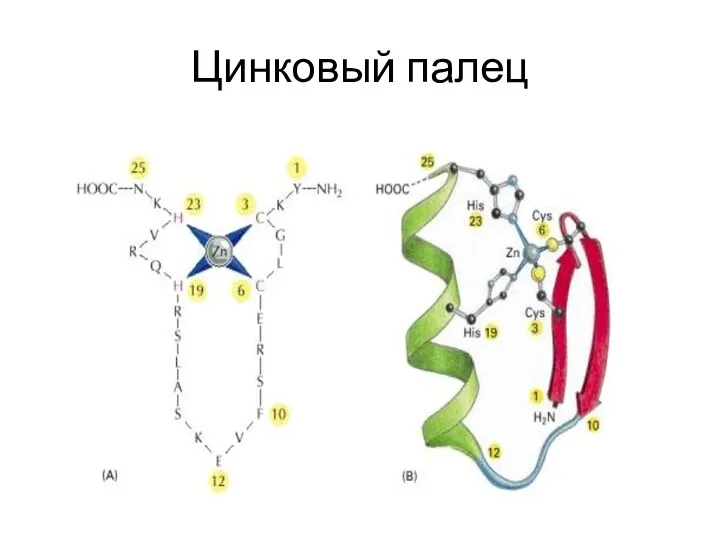
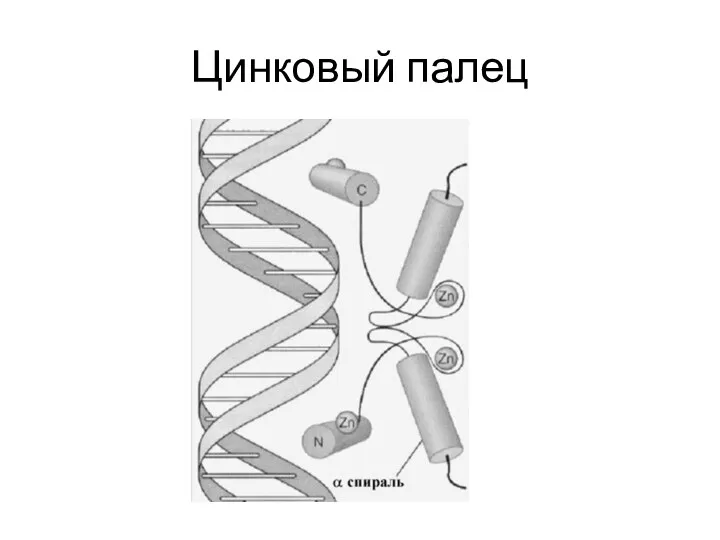
- 111. Цинковый палец (zinc finger domain)
- 112. Цинковый палец (zinc finger domain)
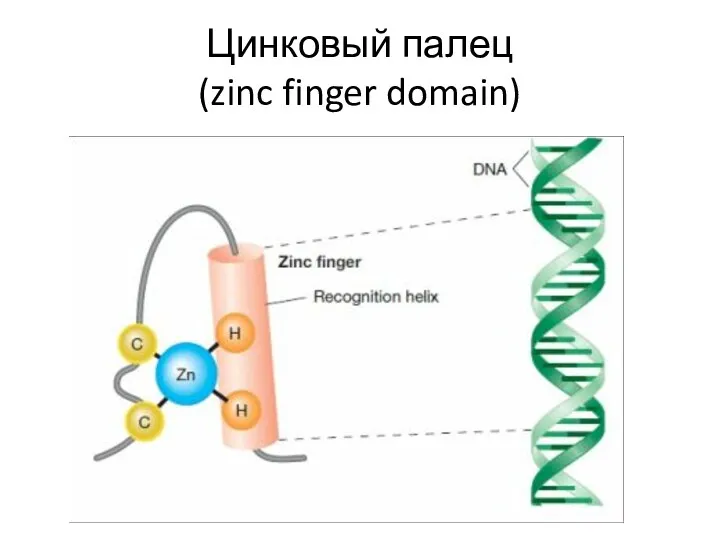
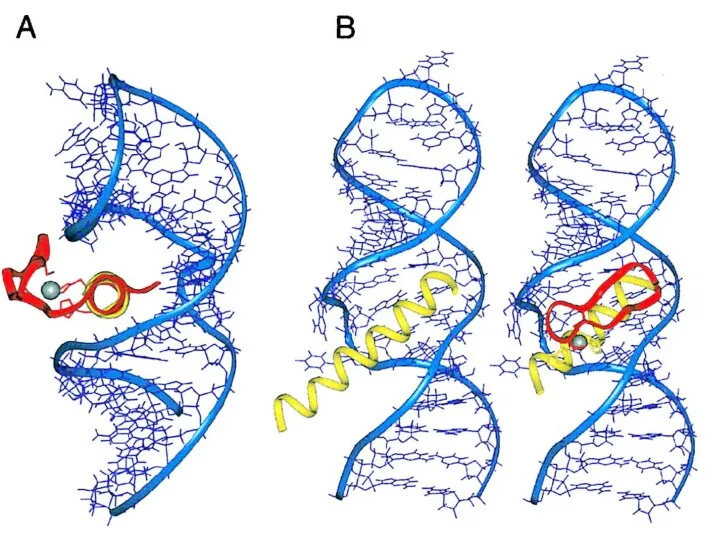
- 113. Цинковый палец
- 115. Цинковый палец
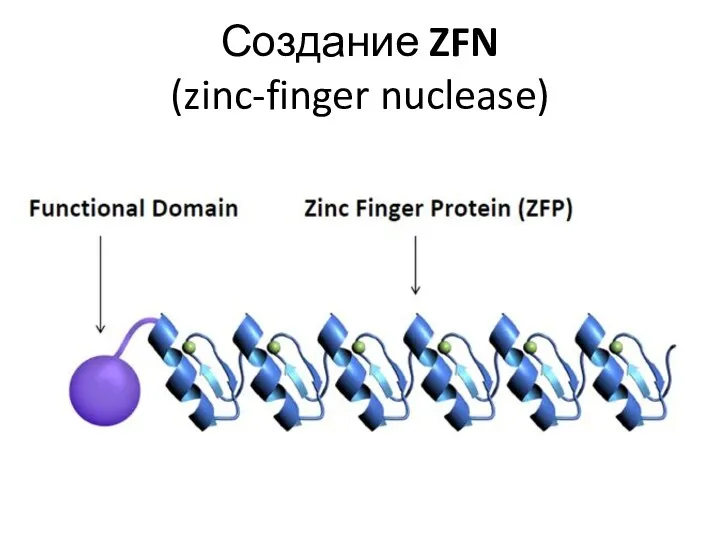
- 116. Создание ZFN (zinc-finger nuclease)
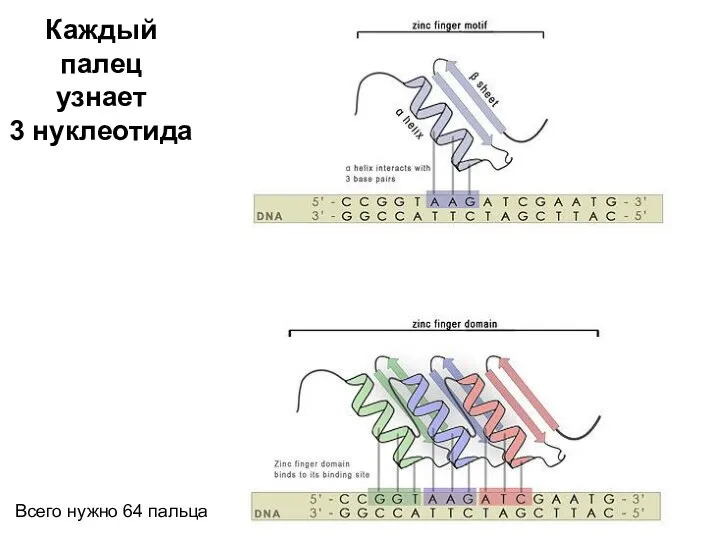
- 117. Каждый палец узнает 3 нуклеотида Всего нужно 64 пальца
- 119. Скачать презентацию



















 Методы исследования в микробиологии. Бактериологический метод исследования
Методы исследования в микробиологии. Бактериологический метод исследования Физиология микроорганизмов
Физиология микроорганизмов Общие свойства живых организмов
Общие свойства живых организмов Обмен веществ
Обмен веществ Семейство крестоцветные
Семейство крестоцветные Лишайники. Лихеноиндикация
Лишайники. Лихеноиндикация Микробы, друзья или враги
Микробы, друзья или враги Животные и птицы (фотографии)
Животные и птицы (фотографии) Викторина по биологии Что? Где? Когда?
Викторина по биологии Что? Где? Когда? Ель китайская
Ель китайская ВПР биология 6 класс 2018 год. №6 Систематика
ВПР биология 6 класс 2018 год. №6 Систематика Эволюция систем органов
Эволюция систем органов Вирусы. (11 класс)
Вирусы. (11 класс) Влияние стресса на продуктивность сельскохозяйственных животных
Влияние стресса на продуктивность сельскохозяйственных животных Физиология спорта. Физиологические основы классификации физических упражнений
Физиология спорта. Физиологические основы классификации физических упражнений Лишайники. Организм- сфинкс. 5 класс (1)
Лишайники. Организм- сфинкс. 5 класс (1) Деление клетки. Митоз
Деление клетки. Митоз История Московского зоопарка
История Московского зоопарка Сцепленное наследование
Сцепленное наследование Белок биосинтезінің негізгі сатылары: транскрипция және трансляция. Генетикалық код, оның қасиеттері
Белок биосинтезінің негізгі сатылары: транскрипция және трансляция. Генетикалық код, оның қасиеттері Головной мозг – конечный мозг
Головной мозг – конечный мозг Отбор как метод селекции
Отбор как метод селекции Презентация к уроку биологии на тему Витамины.
Презентация к уроку биологии на тему Витамины. Неорганические вещества клетки
Неорганические вещества клетки ПРЕЗЕНТАЦИЯ ДЛЯ ИНТЕРАКТИВНОЙ ДОСКИ. Цитология. строение и деление клетки 8 класс.
ПРЕЗЕНТАЦИЯ ДЛЯ ИНТЕРАКТИВНОЙ ДОСКИ. Цитология. строение и деление клетки 8 класс. Species and Taxonomy
Species and Taxonomy презентация для 7 класса Основные группы рыб
презентация для 7 класса Основные группы рыб Вода в природе и жизни человека
Вода в природе и жизни человека