Содержание
- 2. Парное выравнивание является самой фундаментальной операцией биоинформатики Определяет связаны ли структурно или функционально два белка (или
- 3. Парные выравнивания: белковые последовательности могут быть более информативными, чем ДНК Последовательность белка более информативна (20 против
- 4. Принятые однобуквенные коды нуклеиновых кислот 18.09.2019 Кафедра биоинформатики МБФ РНИМУ
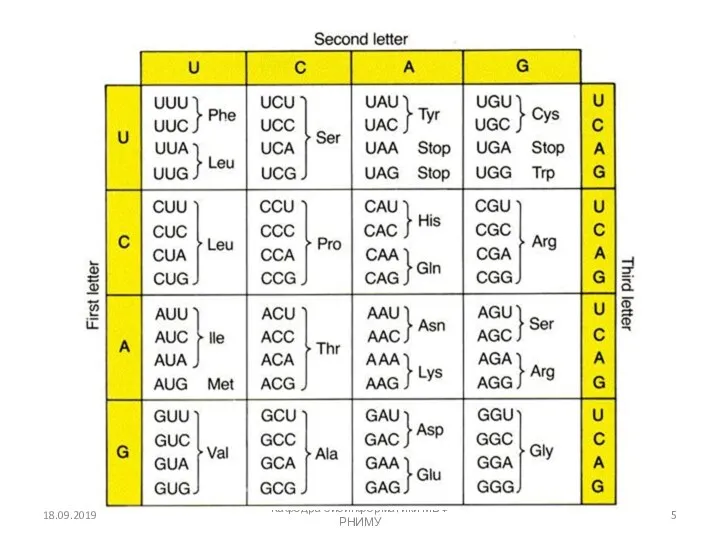
- 5. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ
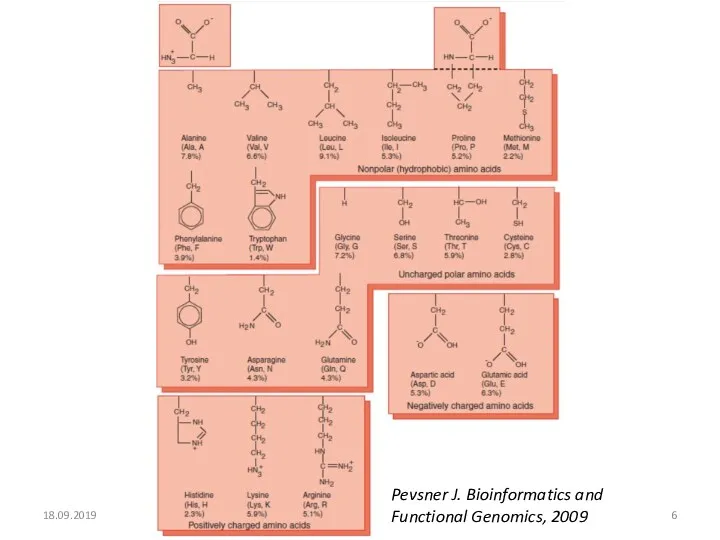
- 6. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Pevsner J. Bioinformatics and Functional Genomics, 2009
- 7. Принятые однобуквенные коды нуклеиновых кислот 18.09.2019 Кафедра биоинформатики МБФ РНИМУ
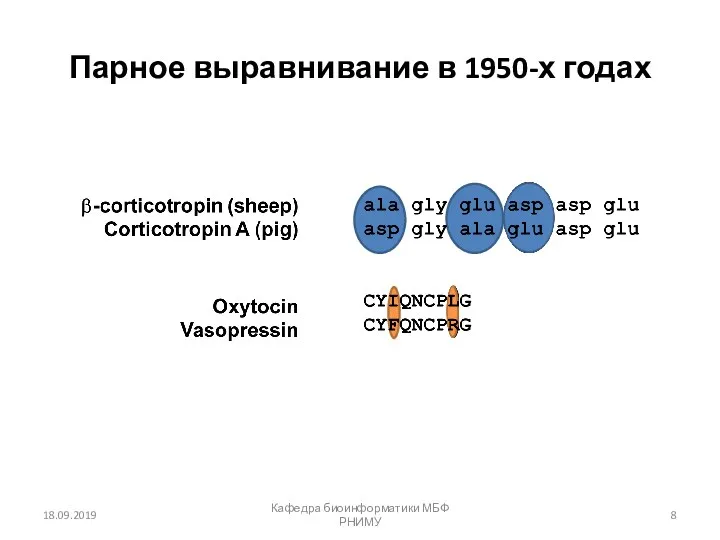
- 8. Парное выравнивание в 1950-х годах 18.09.2019 Кафедра биоинформатики МБФ РНИМУ
- 9. Парные выравнивания ДНК последовательностей полезны в следующих случаях: для подтверждения идентичности кДНК (комплементарная ДНК (кДНК, англ.
- 10. Определение парного выравнивания 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Процесс выравнивания двух последовательностей для достижения максимальных уровней
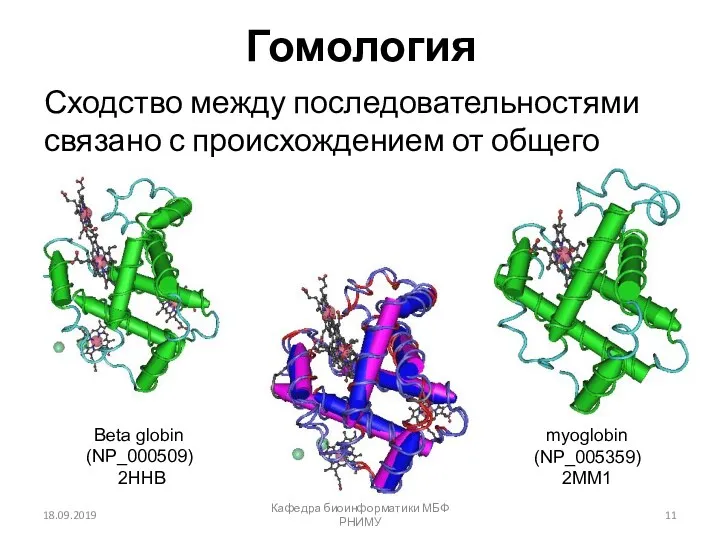
- 11. Гомология Сходство между последовательностями связано с происхождением от общего предка 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Beta
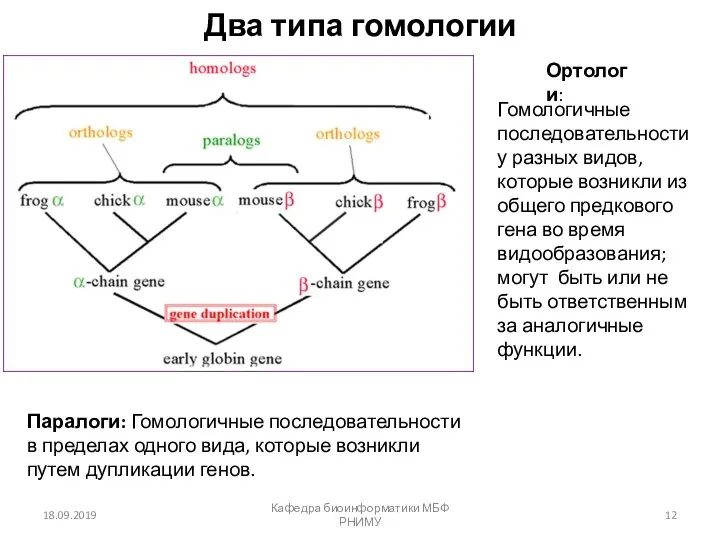
- 12. Два типа гомологии 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Ортологи: Гомологичные последовательности у разных видов, которые возникли
- 13. Общий подход к попарному выравниванию Выбрать две последовательности Выбрать алгоритм, который генерирует оценку сходства Определить условия
- 14. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Элементарное преобразование последовательности: замена буквы или удаление буквы или вставка буквы.
- 15. Расчёт оценки выравнивания (Score) 18.09.2019 Кафедра биоинформатики МБФ РНИМУ http://www.ncbi.nlm.nih.gov/Education/BLASTinfo/Alignment_Scores2.html Идентичность (identity) – остатки (аминокислоты) в
- 16. Сходство последовательностей (Similarity) Степень, в которой нуклеотидные или аминокислотные последовательности связаны между собой. Она основана на
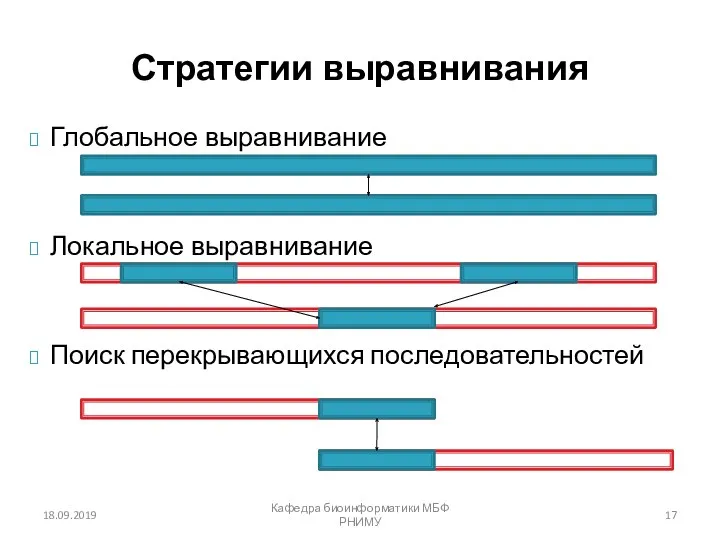
- 17. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Глобальное выравнивание Локальное выравнивание Поиск перекрывающихся последовательностей Стратегии выравнивания
- 18. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ
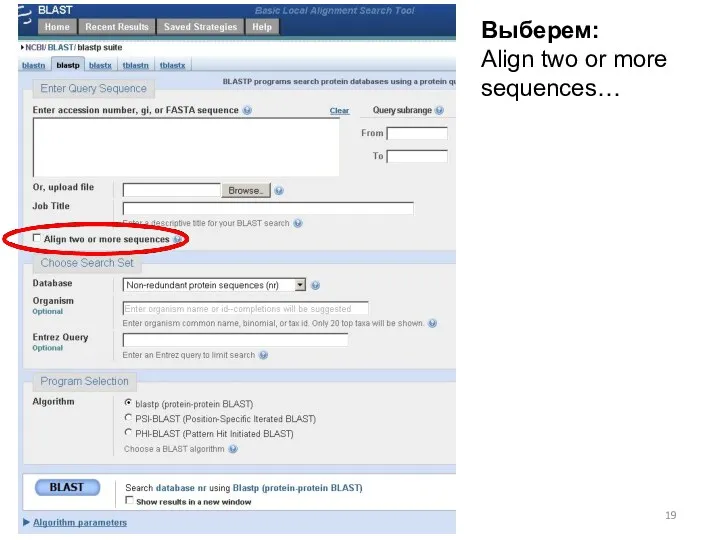
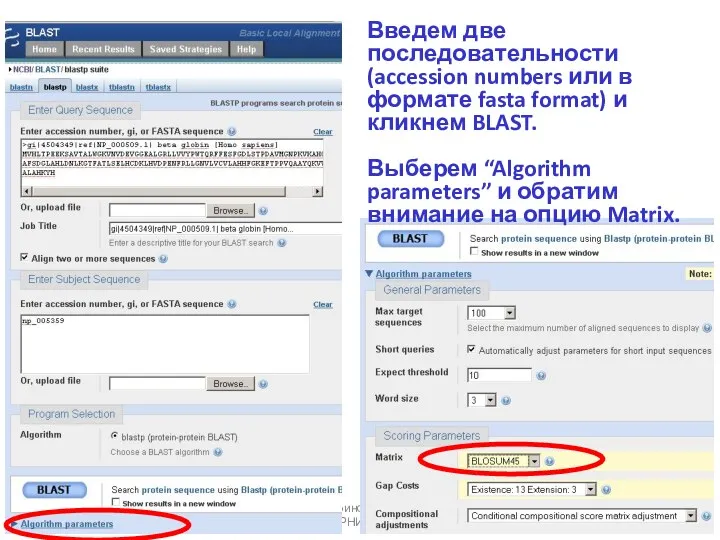
- 19. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Выберем: Аlign two or more sequences…
- 20. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Введем две последовательности (accession numbers или в формате fasta format) и
- 21. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Результаты парного выравнивания human beta globin и myoglobin Myoglobin RefSeq Query
- 22. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Результаты парного выравнивания human beta globin и myoglobin: Score = сумма
- 23. Пробелы (gaps) 18.09.2019 Кафедра биоинформатики МБФ РНИМУ First gap position scores -11 Second gap position scores
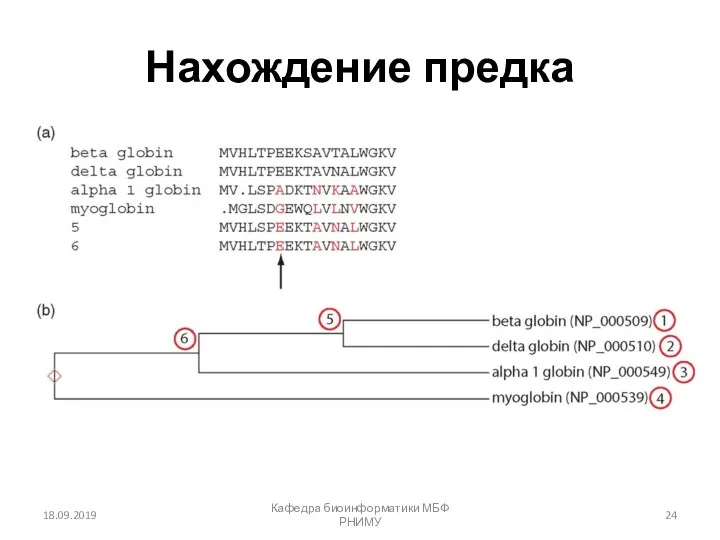
- 24. Нахождение предка 18.09.2019 Кафедра биоинформатики МБФ РНИМУ
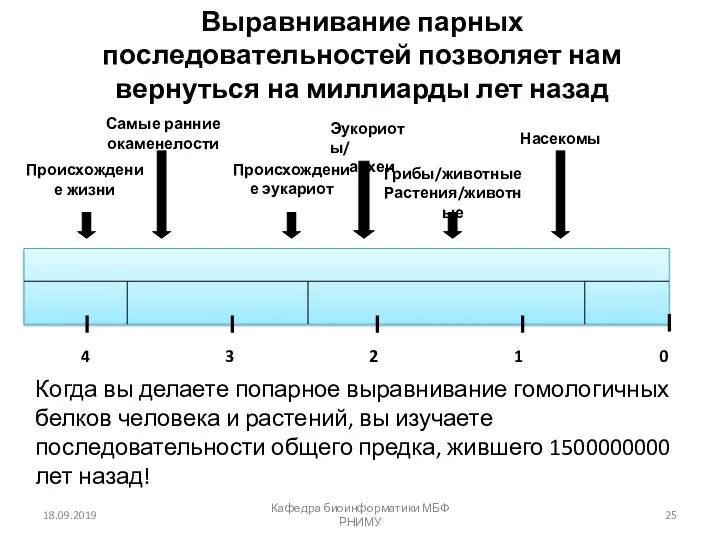
- 25. Выравнивание парных последовательностей позволяет нам вернуться на миллиарды лет назад 18.09.2019 Кафедра биоинформатики МБФ РНИМУ 4
- 26. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ fly GAKKVIISAP SAD.APM..F VCGVNLDAYK PDMKVVSNAS CTTNCLAPLA human GAKRVIISAP SAD.APM..F VMGVNHEKYD NSLKIISNAS
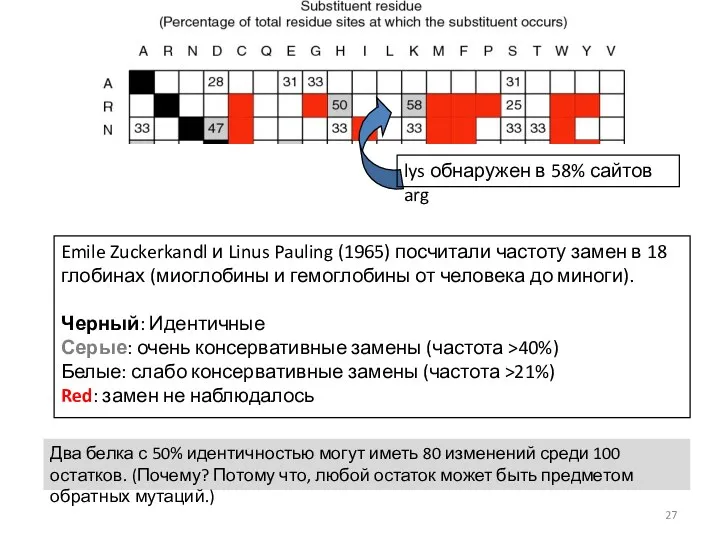
- 27. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Emile Zuckerkandl и Linus Pauling (1965) посчитали частоту замен в 18
- 28. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ
- 29. Матрицы замен Матрица замен содержит значения, пропорциональные вероятности того, что аминокислота i мутирует в аминокислоту j
- 30. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Основные матрицы замен, применяемые в исследованиях PAM (Percentage of Acceptable Point
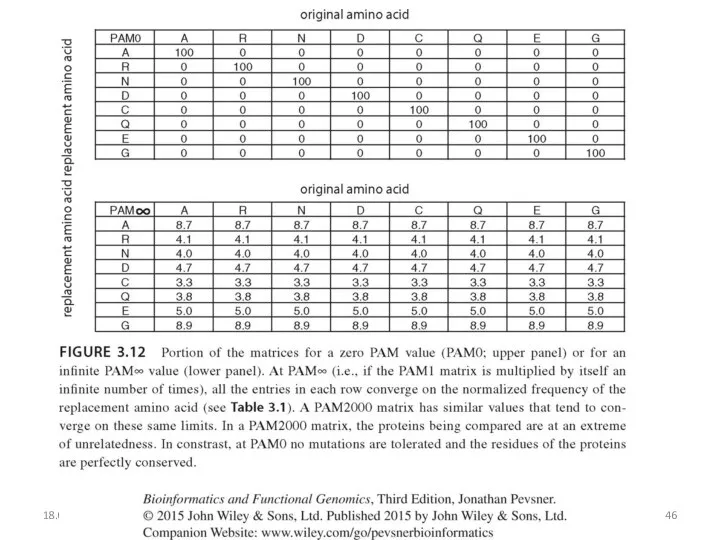
- 31. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Мы можем варьировать: от PAM250 = (PAM1)250, оценочная матрица, которая присваивает
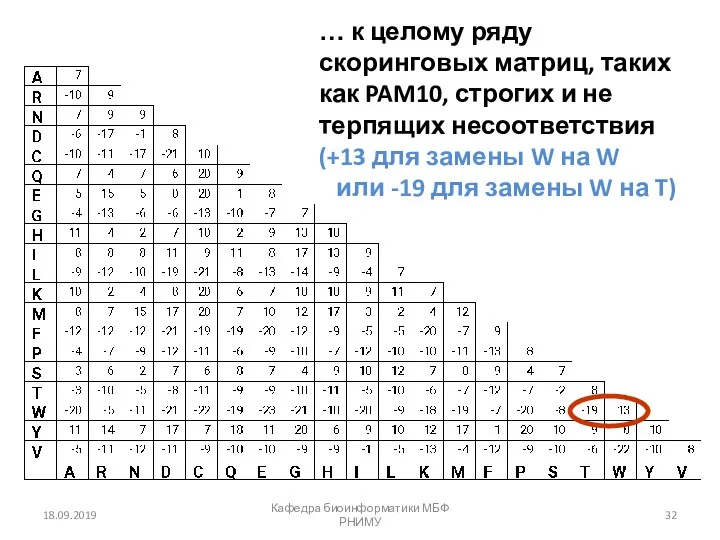
- 32. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ … к целому ряду скоринговых матриц, таких как PAM10, строгих и
- 33. 34 белковых надсемейства Dayhoff 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Белок PAMs за 100 млн. лет Ig
- 34. 34 белковых надсемейства Dayhoff 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Белок PAMs за 100 млн. лет Ig
- 35. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ 34 белковых надсемейства Dayhoff Белок PAMs за 100 млн. лет apolipoprotein
- 36. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ 34 белковых надсемейства Dayhoff Белок PAMs за 100 млн. лет triosephosphate
- 37. Парное выравнивание человеческого (NP_005203) и мышиного (NP_031812) убиквитина 18.09.2019 Кафедра биоинформатики МБФ РНИМУ
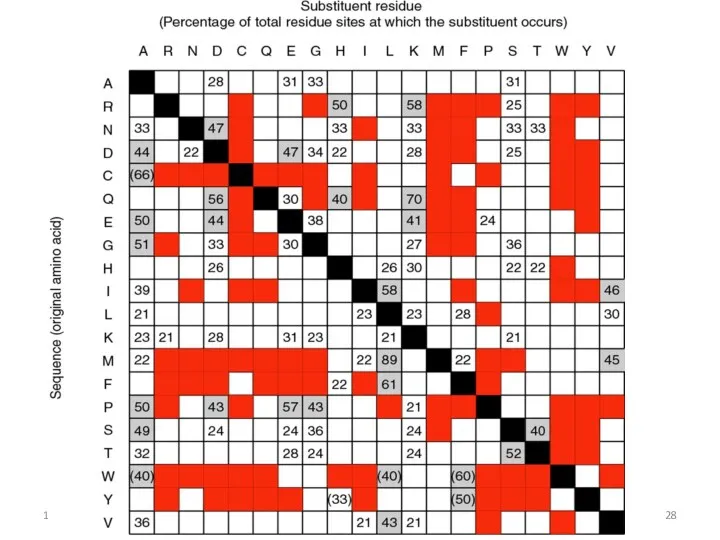
- 38. Подход Dayhoff позволяет посчитать оценку замены для любых двух выровненных аминокислотных остатков 18.09.2019 Кафедра биоинформатики МБФ
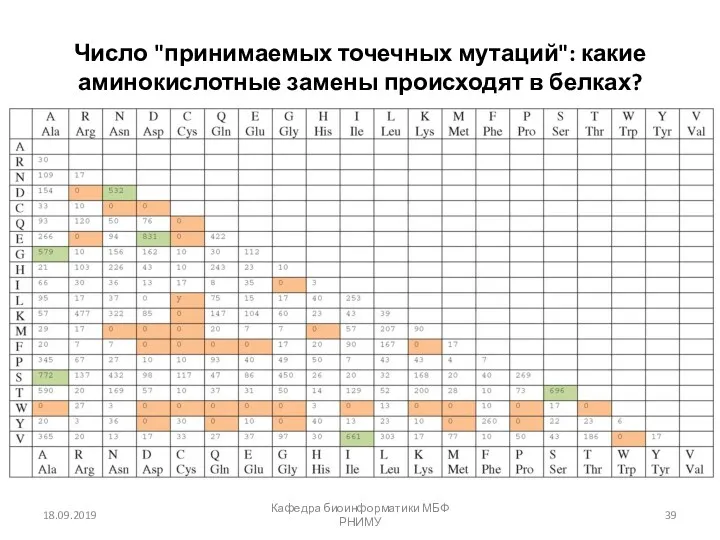
- 39. Число "принимаемых точечных мутаций": какие аминокислотные замены происходят в белках? 18.09.2019 Кафедра биоинформатики МБФ РНИМУ
- 40. Относительная мутабельность аминокислотных остатков 18.09.2019 Кафедра биоинформатики МБФ РНИМУ
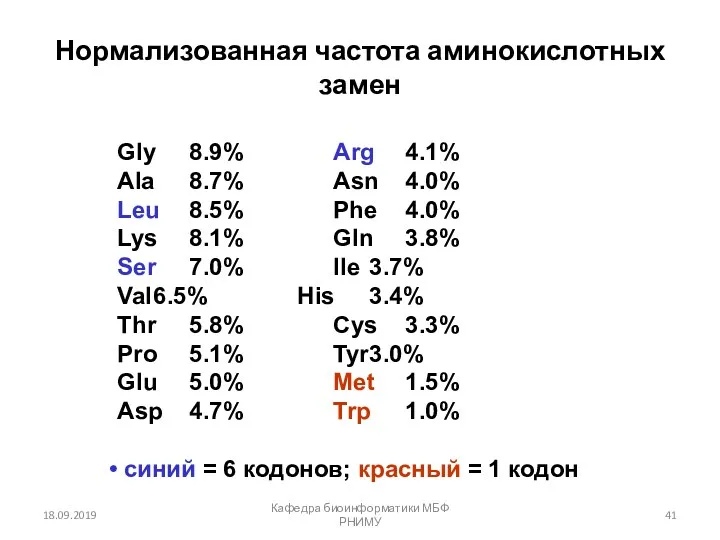
- 41. Нормализованная частота аминокислотных замен 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Gly 8.9% Arg 4.1% Ala 8.7% Asn
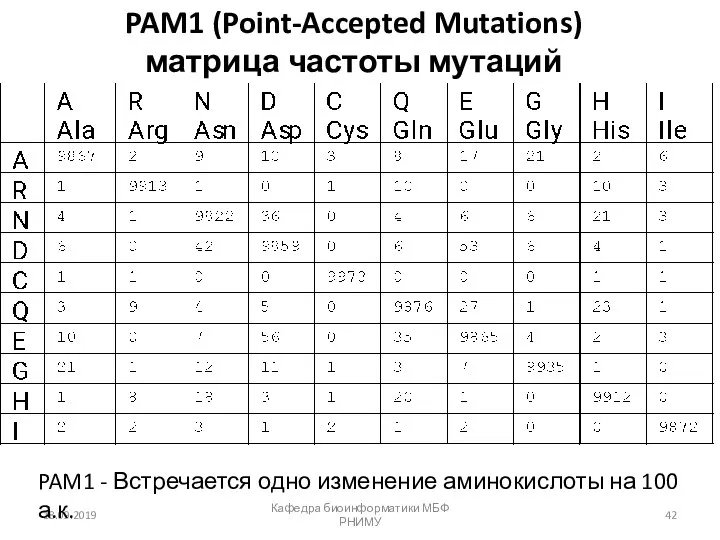
- 42. PAM1 (Point-Accepted Mutations) матрица частоты мутаций 18.09.2019 Кафедра биоинформатики МБФ РНИМУ PAM1 - Встречается одно изменение
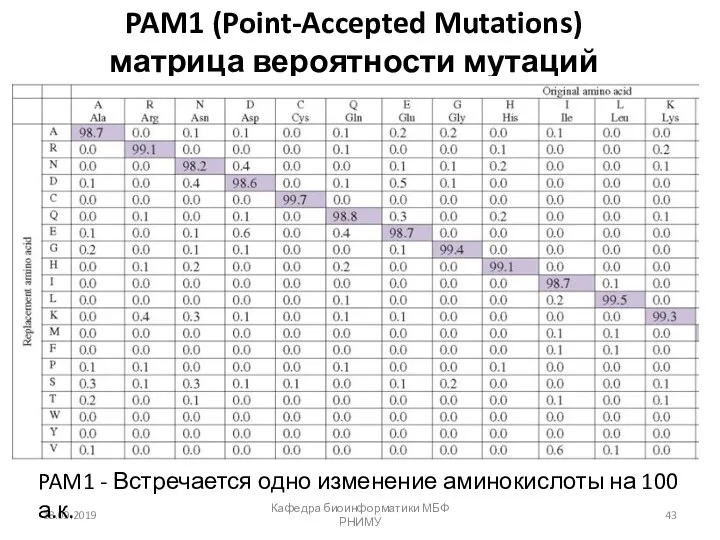
- 43. PAM1 (Point-Accepted Mutations) матрица вероятности мутаций 18.09.2019 Кафедра биоинформатики МБФ РНИМУ PAM1 - Встречается одно изменение
- 44. Множественное выравнивание последовательностей глицеральдегид 3-фосфат дегидрогеназ: колонки остатков могут иметь высокую или низкую консервативность 18.09.2019 Кафедра
- 45. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ
- 46. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ
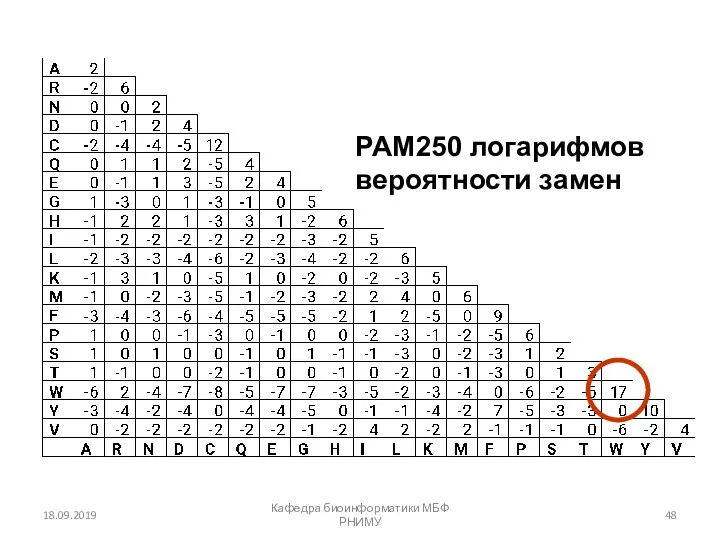
- 47. PAM250 матрица вероятности мутаций Встречается 250 изменений на 100 а.к. остатков 18.09.2019 Кафедра биоинформатики МБФ РНИМУ
- 48. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ PAM250 логарифмов вероятности замен
- 49. Почему мы используем вместо матрицы вероятностей мутаций, матрицу логарифмической вероятности мутаций? Оценочная матрица должна быть удобной
- 50. Переход от матрицы вероятности замен к логарифмической матрице Оценка S для выравнивания остатков a,b: S(a,b) =
- 51. Что означают числа логарифмической матрицы? Счет 2 показывает, что замена аминокислоты происходит в 1,6 раза чаще,
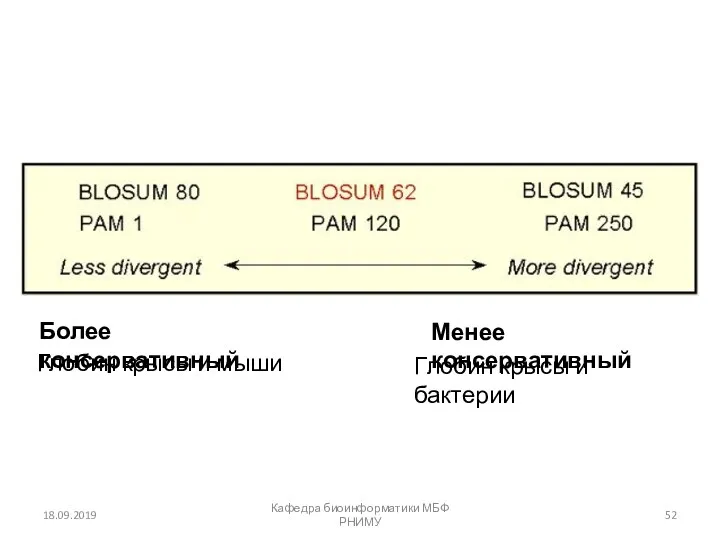
- 52. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Глобин крысы и мыши Глобин крысы и бактерии Более консервативный Менее
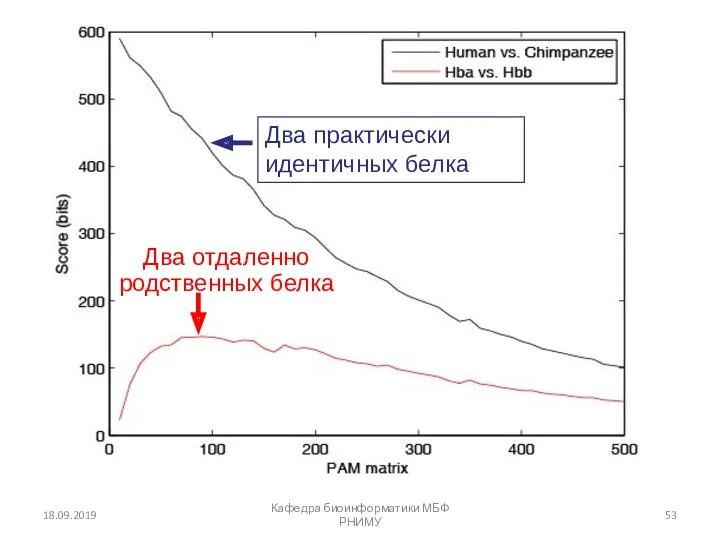
- 53. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Два практически идентичных белка Два отдаленно родственных белка
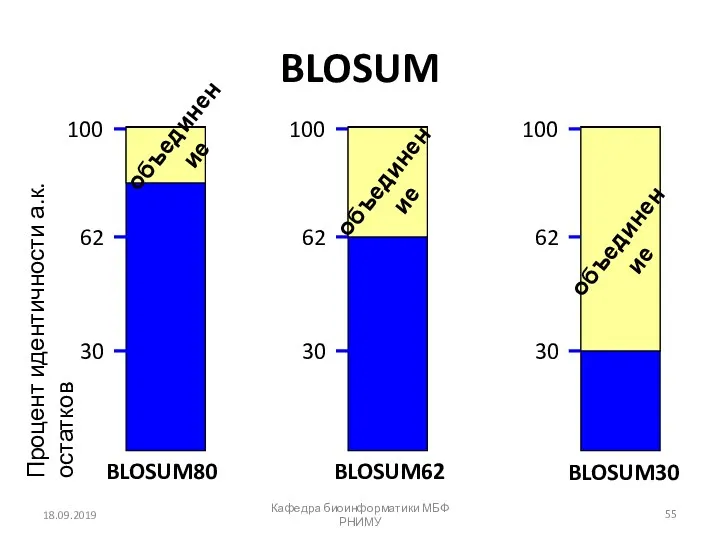
- 54. Матрица BLOSUM (Block substitution matrix) Основана на локальном выравнивании Основана на рассмотрении только консервативных участков (блоков)
- 55. BLOSUM 18.09.2019 Кафедра биоинформатики МБФ РНИМУ 100 62 30 Процент идентичности а.к. остатков BLOSUM62 100 62
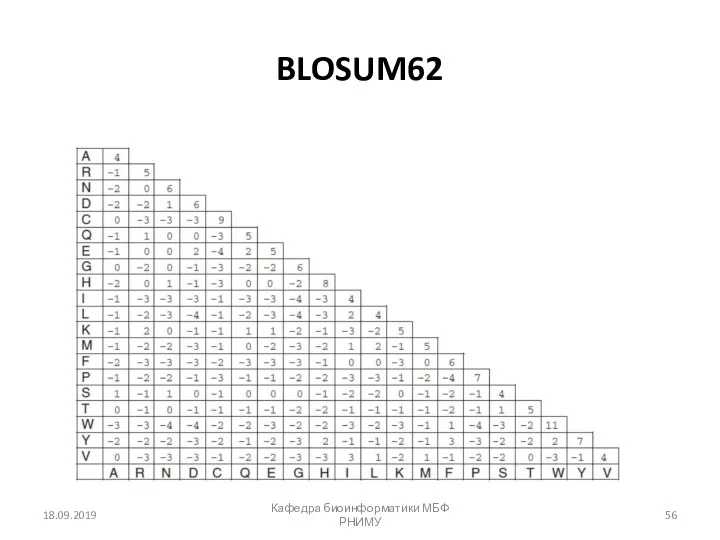
- 56. BLOSUM62 18.09.2019 Кафедра биоинформатики МБФ РНИМУ
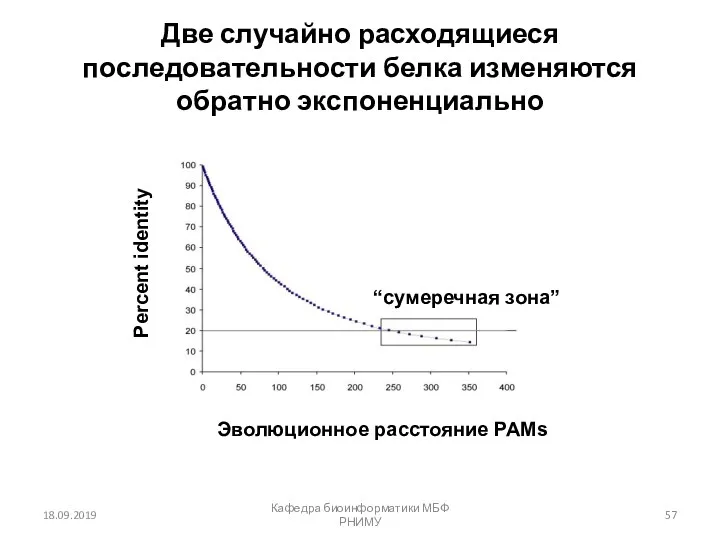
- 57. Две случайно расходящиеся последовательности белка изменяются обратно экспоненциально 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Percent identity Эволюционное
- 58. Алгоритмы выравнивания: Ниделмана-Вунша (Needleman-Wunsch) и Смита-Уотермана (Smith-Waterman) Алгоритм глобального выравнивания Ниделмана-Вунша (1970) Алгоритм локального выравнивания Смита-Уотермана
- 59. Алгоритм глобального выравнивания Ниделмана-Вунша Две последовательности сравниваются в матрице с осями Х и Y (каждая из
- 60. Три шага в глобальном выравнивании алгоритмом Ниделмана-Вунша Построить матрицу Оценка матрицы Выбрать оптимальное выравнивание 18.09.2019 Кафедра
- 61. Четыре возможных исхода при выравнивании двух последовательностей 18.09.2019 Кафедра биоинформатики МБФ РНИМУ [1] идентичность (оставаться вдоль
- 62. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ
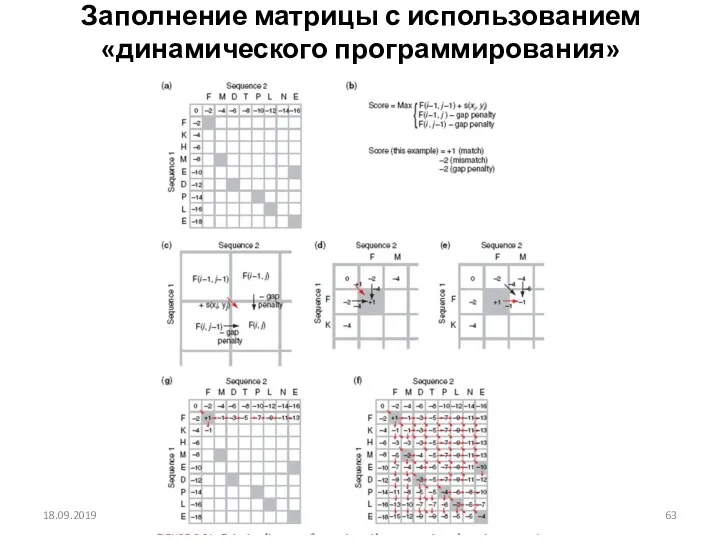
- 63. Заполнение матрицы с использованием «динамического программирования» 18.09.2019 Кафедра биоинформатики МБФ РНИМУ
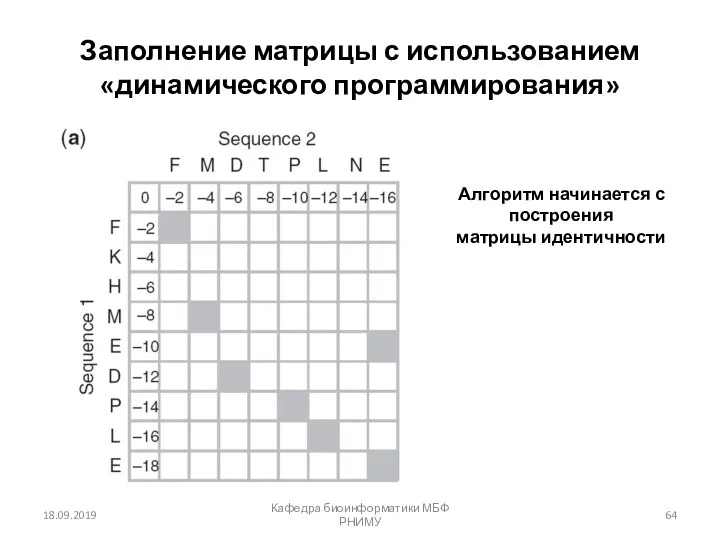
- 64. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Заполнение матрицы с использованием «динамического программирования» Алгоритм начинается с построения матрицы
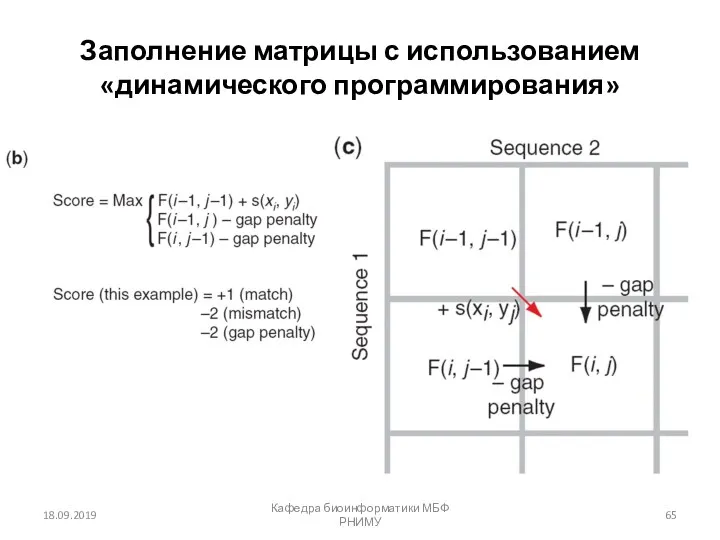
- 65. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Заполнение матрицы с использованием «динамического программирования»
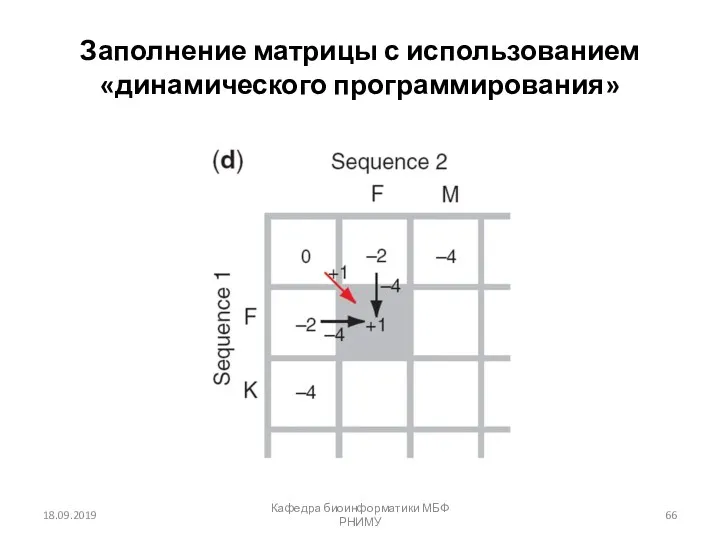
- 66. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Заполнение матрицы с использованием «динамического программирования»
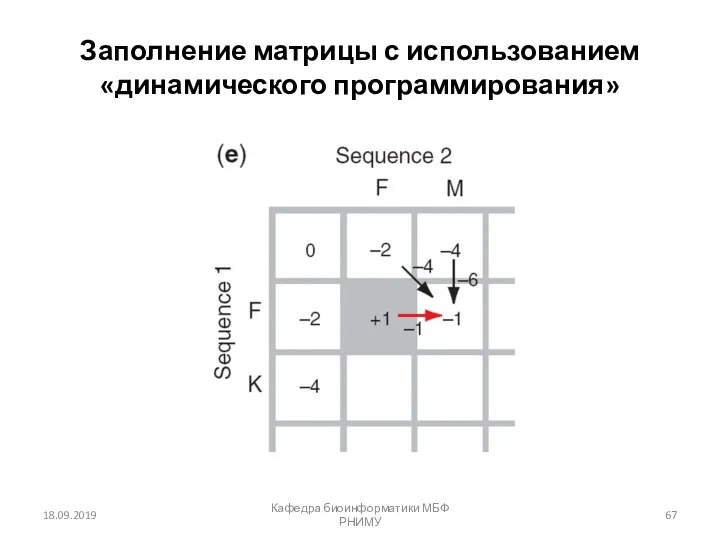
- 67. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Заполнение матрицы с использованием «динамического программирования»
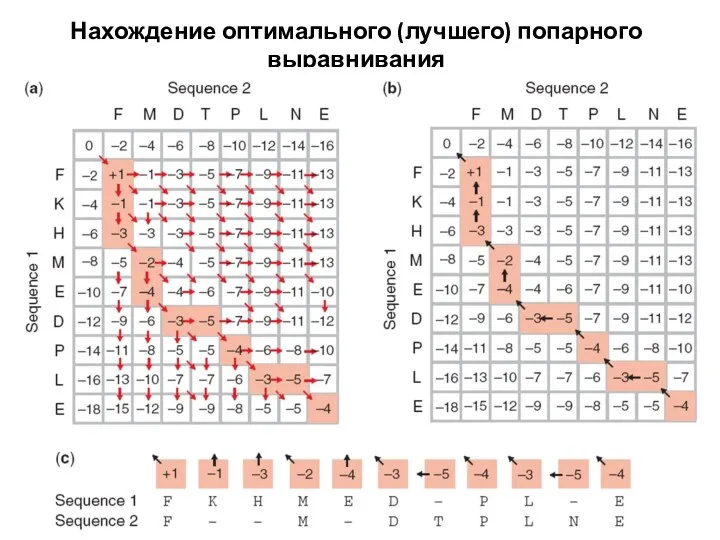
- 68. Нахождение оптимального (лучшего) попарного выравнивания 18.09.2019 Кафедра биоинформатики МБФ РНИМУ
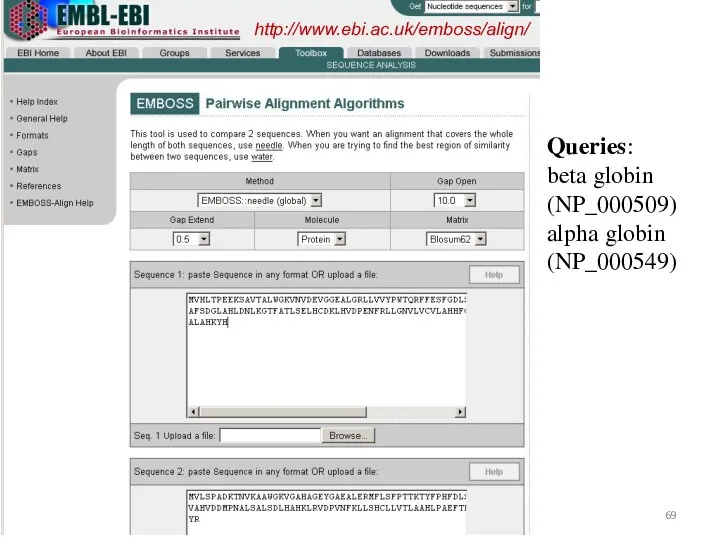
- 69. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Queries: beta globin (NP_000509) alpha globin (NP_000549) http://www.ebi.ac.uk/emboss/align/
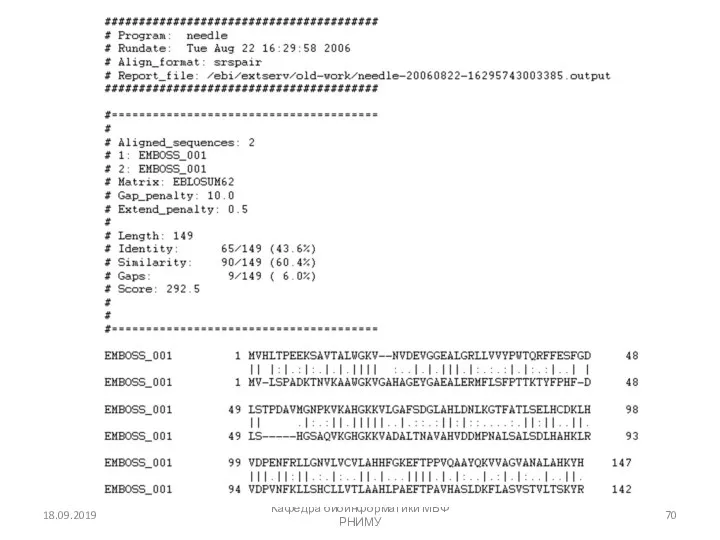
- 70. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ
- 71. Глобальное vs. локальное выравнивания Глобальное выравнивание (Ниделмана-Вунша) проходит от одного конца каждой последовательности к другому концу.
- 72. Глобальное выравнивание (верх) включает совпадения, игнорируемые локальным выравниванием (низ) 18.09.2019 Кафедра биоинформатики МБФ РНИМУ NP_824492, NP_337032
- 73. Алгоритм локального выравнивания Смита-Уотермана Создание матрицы между двумя белками (размер m + 1, n + 1)
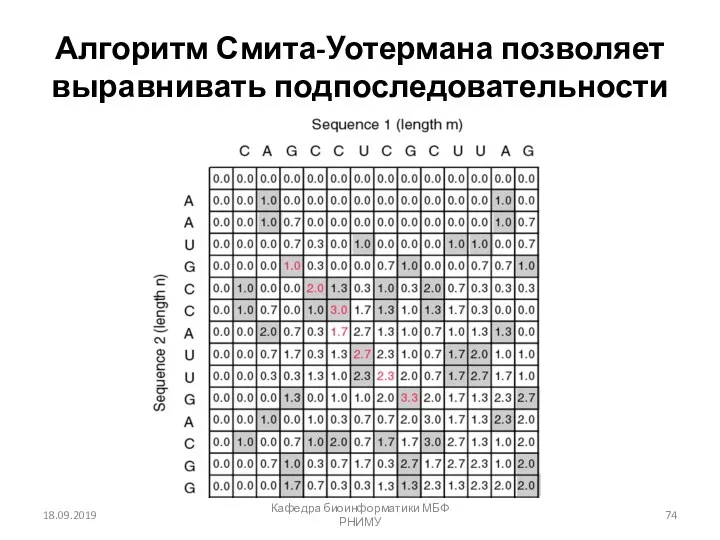
- 74. Алгоритм Смита-Уотермана позволяет выравнивать подпоследовательности 18.09.2019 Кафедра биоинформатики МБФ РНИМУ
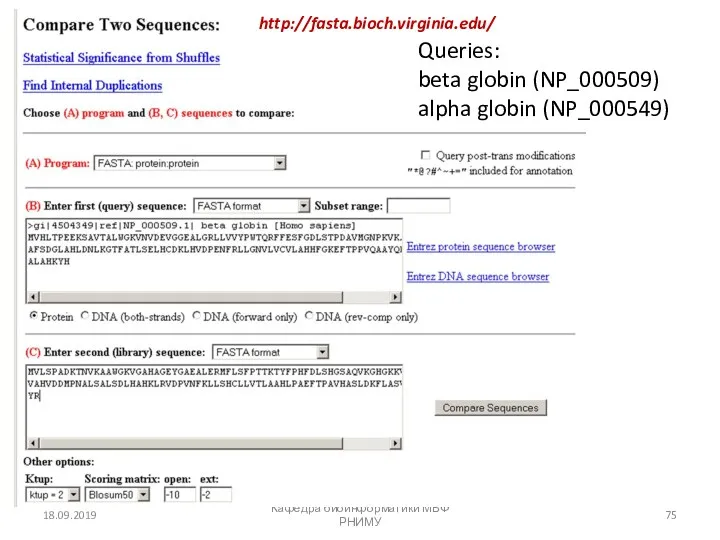
- 75. 18.09.2019 Кафедра биоинформатики МБФ РНИМУ Queries: beta globin (NP_000509) alpha globin (NP_000549) http://fasta.bioch.virginia.edu/
- 77. Скачать презентацию






































 Красная Книга Ростовской области
Красная Книга Ростовской области Механизмы биоэлектрогенеза
Механизмы биоэлектрогенеза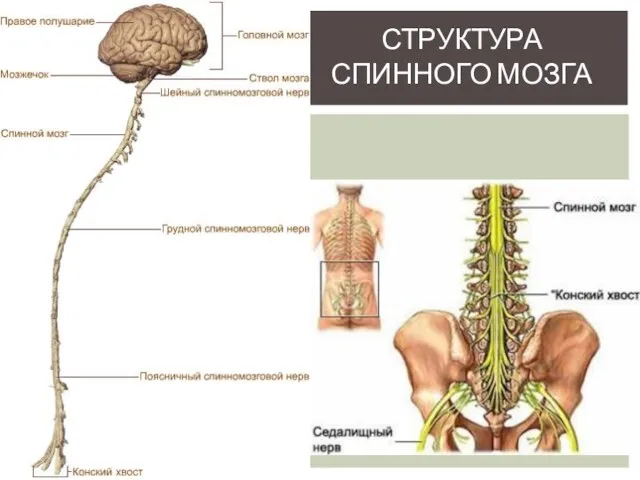 Структура спинного мозга
Структура спинного мозга Обеспечение клеток энергией
Обеспечение клеток энергией Строение и жизнедеятельность бактерий
Строение и жизнедеятельность бактерий Бактериология. Классификация бактерий. Морфология бактерий и методы её изучения. (Лекция 9)
Бактериология. Классификация бактерий. Морфология бактерий и методы её изучения. (Лекция 9) Физиология сердца. (Лекция 5.1)
Физиология сердца. (Лекция 5.1) Отряд Киты
Отряд Киты Межвидовые отношения организмов
Межвидовые отношения организмов Эволюция ОДС (опорно-двигательной системы)
Эволюция ОДС (опорно-двигательной системы) Строение семян однодольных и двудольных растений. Морфология проростка. Прорастание семян
Строение семян однодольных и двудольных растений. Морфология проростка. Прорастание семян Предмет и структура ихтиологической науки
Предмет и структура ихтиологической науки Цветы в легендах, поэзии, музыке
Цветы в легендах, поэзии, музыке ВНД человека. Вторая сигнальная система
ВНД человека. Вторая сигнальная система Морфология. Общие понятия
Морфология. Общие понятия Конструкции, обогрев и эксплуатация сооружений защищенного грунта
Конструкции, обогрев и эксплуатация сооружений защищенного грунта Жизнедеятельность клетки
Жизнедеятельность клетки Natural selection of human population. Darwin’s theory of evolution by natural selection
Natural selection of human population. Darwin’s theory of evolution by natural selection Антропогенез. Расы. Расизм. Часть 4
Антропогенез. Расы. Расизм. Часть 4 Бактерии
Бактерии Потребности и интересы. Уровень развития способностей человека
Потребности и интересы. Уровень развития способностей человека Динозавры
Динозавры Пищеварительная система
Пищеварительная система Физиология спорта. Физиологические основы классификации физических упражнений
Физиология спорта. Физиологические основы классификации физических упражнений Презентация к выступлению на научной конференции Совершенствование естественнонаучного школьного образования
Презентация к выступлению на научной конференции Совершенствование естественнонаучного школьного образования Клубеньковые долгоносики. Гороховая зерновка
Клубеньковые долгоносики. Гороховая зерновка Типы кожи лица. Уход за сухой кожей
Типы кожи лица. Уход за сухой кожей Как животные пустынь получают воду
Как животные пустынь получают воду