Содержание
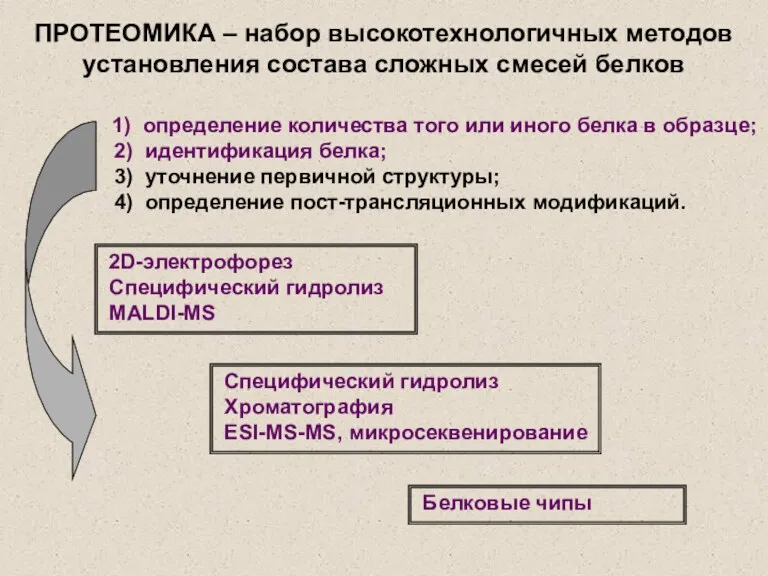
- 2. 1) определение количества того или иного белка в образце; 2) идентификация белка; 3) уточнение первичной структуры;
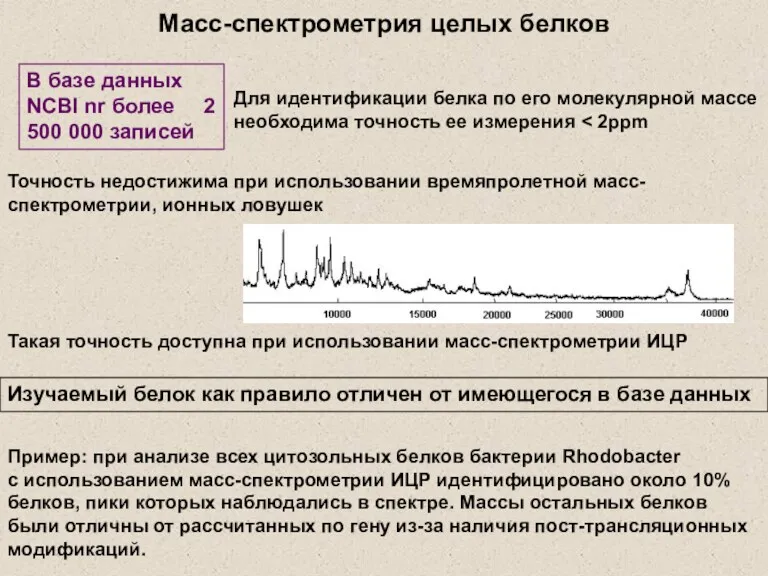
- 3. Масс-спектрометрия целых белков Точность недостижима при использовании времяпролетной масс-спектрометрии, ионных ловушек Изучаемый белок как правило отличен
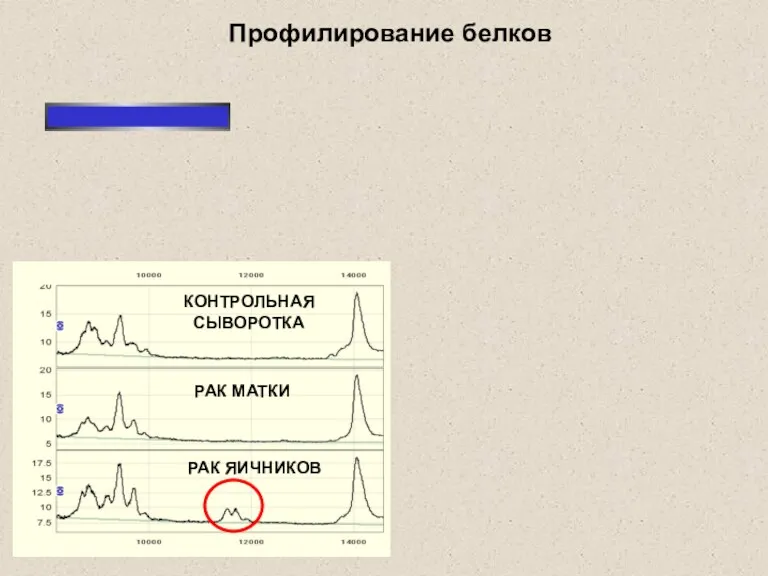
- 4. Профилирование белков КОНТРОЛЬНАЯ СЫВОРОТКА РАК МАТКИ РАК ЯИЧНИКОВ Подложки с разными типами поверхности: обращенная фаза, анионообменная,
- 5. 2D электрофорез белков Компьютерный анализ изображений гелей Вырезание и трипсинолиз белков в фрагментах геля Экстракция пептидов
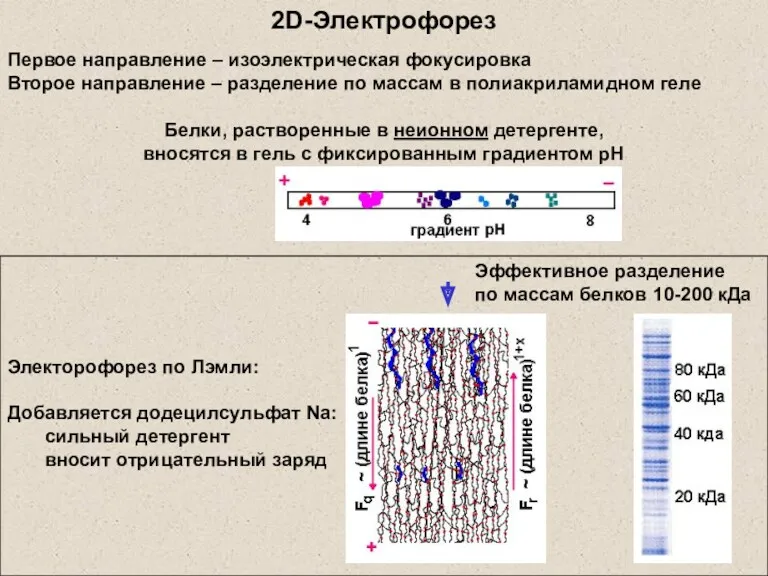
- 6. 2D-Электрофорез Электорофорез по Лэмли: Добавляется додецилсульфат Na: сильный детергент вносит отрицательный заряд Эффективное разделение по массам
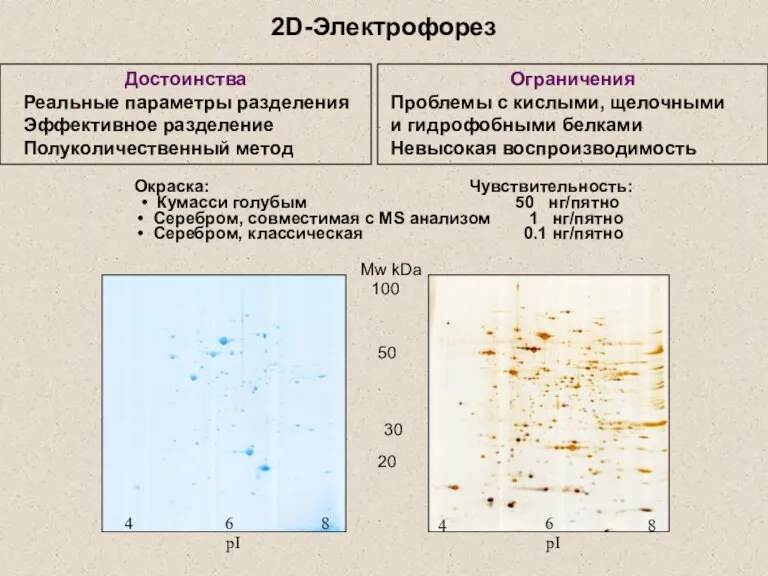
- 7. 2D-Электрофорез 30 Достоинства Реальные параметры разделения Эффективное разделение Полуколичественный метод Ограничения Проблемы с кислыми, щелочными и
- 8. 2D-Электрофорез Окраска DIGE Cy3/Cy5 Электрофорез 2-х предварительно окрашенных белков Mycoplasma gallisepticum проводится на одном геле. Зеленым
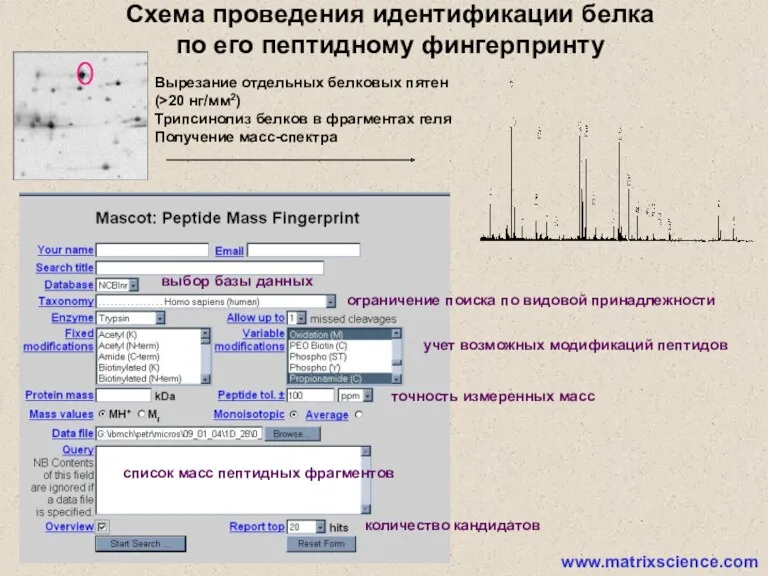
- 9. ограничение поиска по видовой принадлежности выбор базы данных учет возможных модификаций пептидов точность измеренных масс список
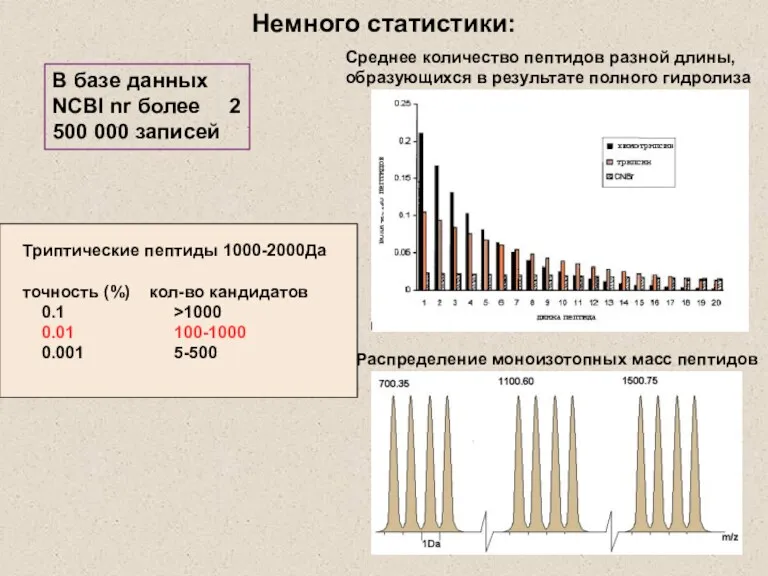
- 10. Немного статистики: Среднее количество пептидов разной длины, образующихся в результате полного гидролиза Распределение моноизотопных масс пептидов
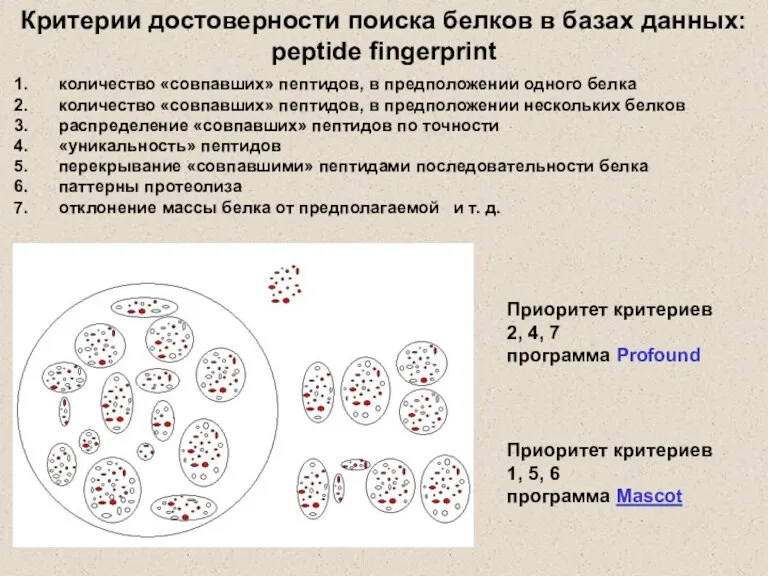
- 11. Критерии достоверности поиска белков в базах данных: peptide fingerprint количество «совпавших» пептидов, в предположении одного белка
- 12. N Масса Вероятность Описание gi|8486123 27875 88 reading frame [Influenza A virus] gi|4996872 26451 74 M1
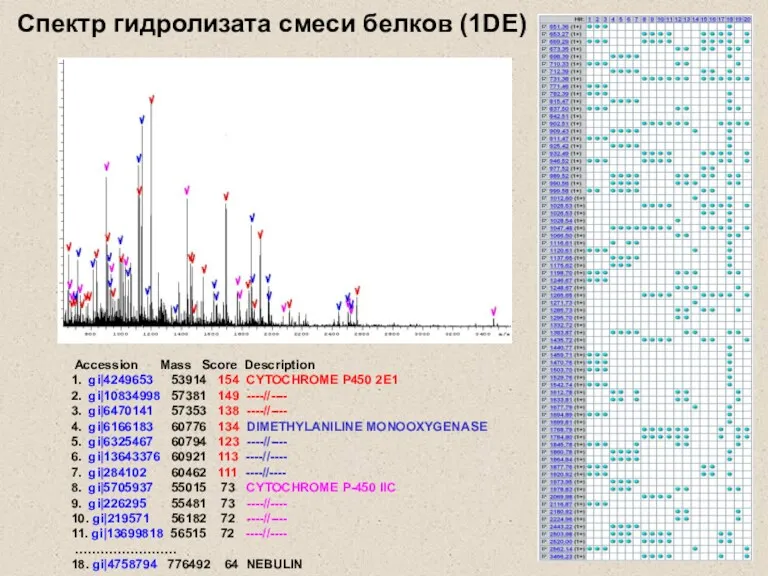
- 13. Accession Mass Score Description 1. gi|4249653 53914 154 CYTOCHROME P450 2E1 2. gi|10834998 57381 149 ----//----
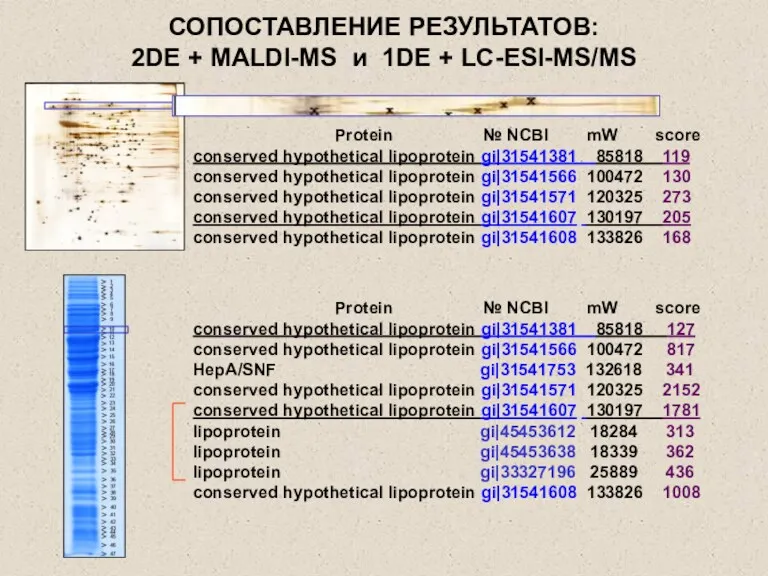
- 14. 2DE + MALDI-MS - результаты Белковых пятен > 400 Проанализировано >150 Индивидуальных белков Mycoplasma gallisepticum 105
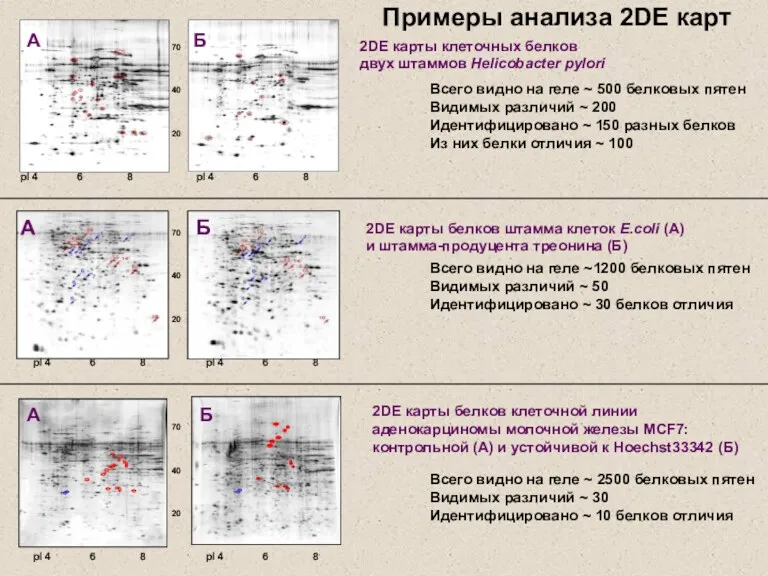
- 15. 70 40 20 pI 4 6 8 А Б 2DЕ карты белков клеточной линии аденокарциномы молочной
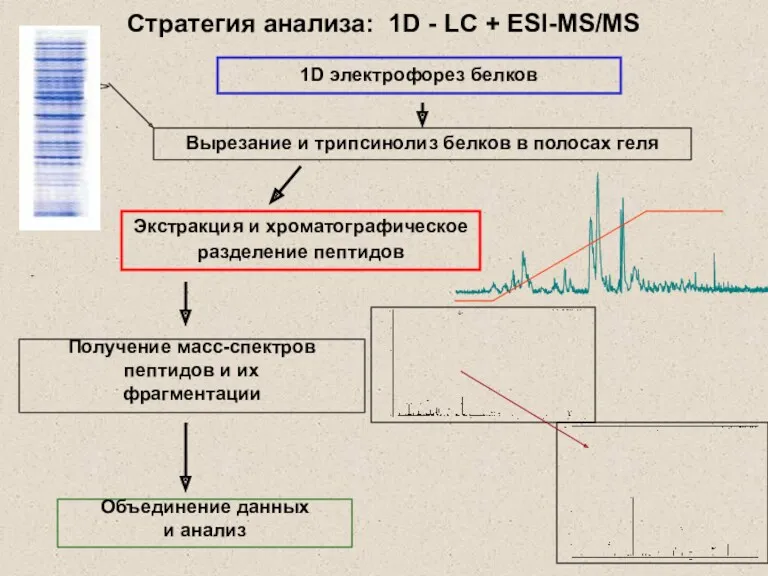
- 16. Стратегия анализа: 1D - LC + ESI-MS/MS Получение масс-спектров пептидов и их фрагментации Объединение данных и
- 17. Способы фрагментации пептидов LID – фрагментация, индуцированная лазером CID – фрагментация, индуцированная столкновениями ECD – фрагментация,
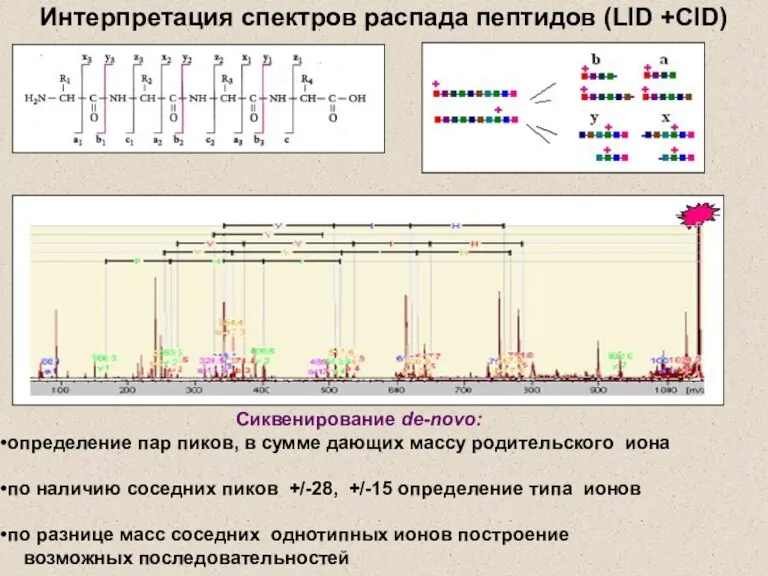
- 18. Интерпретация спектров распада пептидов TIGTHPSSSAGLK [MH]2+
- 19. Отбор из базы данных всех кандидатов, содержащих триптические пептиды указанных m/z 2. «Примерка» спектров фрагментации (MS/MS)
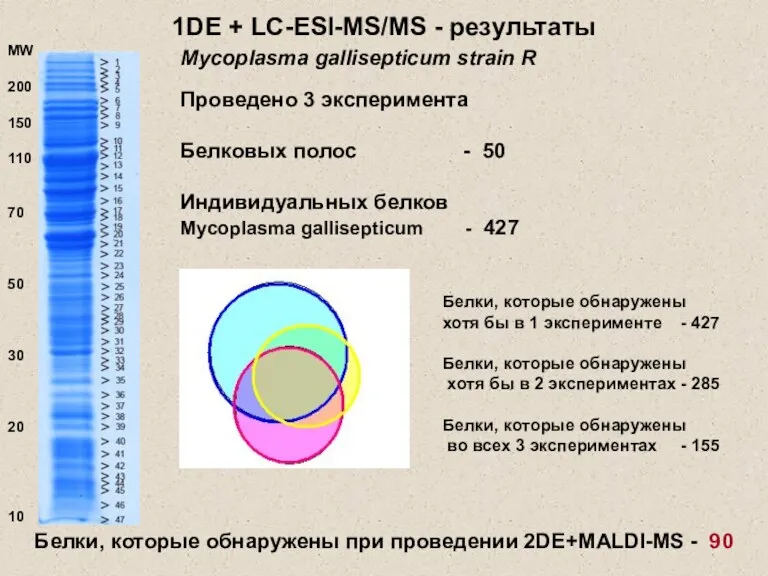
- 20. 1DE + LC-ESI-MS/MS - результаты Проведено 3 эксперимента Белковых полос - 50 Индивидуальных белков Mycoplasma gallisepticum
- 21. СОПОСТАВЛЕНИЕ РЕЗУЛЬТАТОВ: 2DE + MALDI-MS и 1DE + LC-ESI-MS/MS Protein № NCBI mW score conserved hypothetical
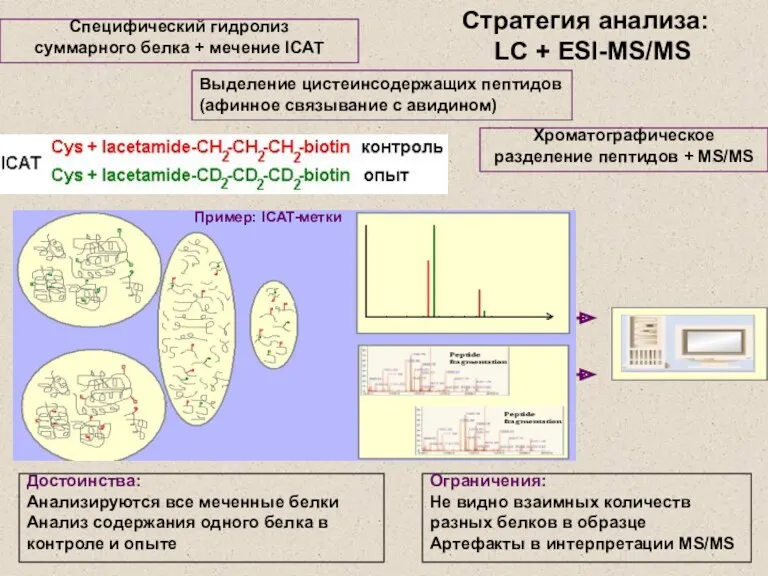
- 22. Стратегия анализа: LC + ESI-MS/MS Специфический гидролиз суммарного белка + мечение ICAT Хроматографическое разделение пептидов +
- 23. Интерпретация спектров распада пептидов (LID +CID) Сиквенирование de-novo: определение пар пиков, в сумме дающих массу родительского
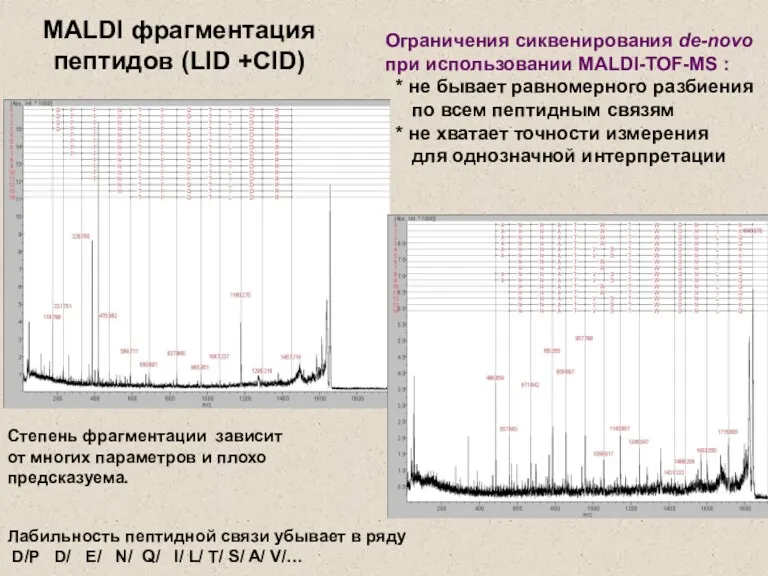
- 24. Лабильность пептидной связи убывает в ряду D/P D/ E/ N/ Q/ l/ L/ T/ S/ A/
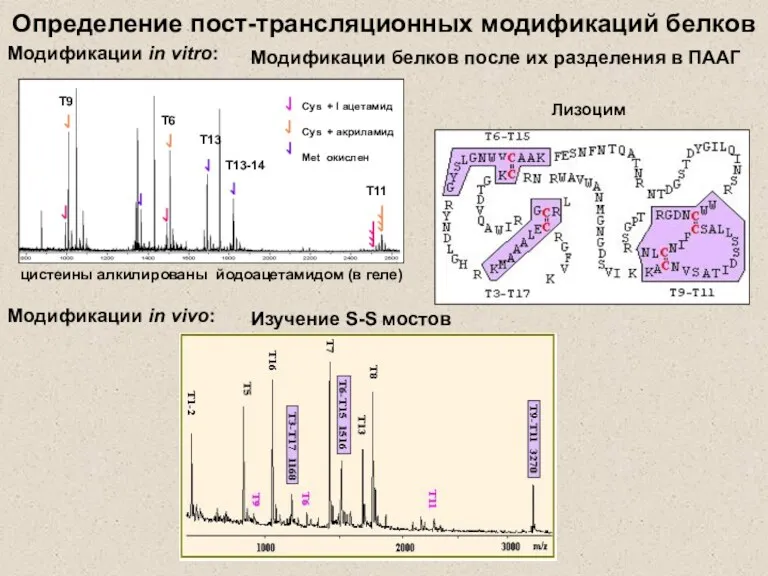
- 25. Определение пост-трансляционных модификаций белков Модификации in vitro: Модификации белков после их разделения в ПААГ цистеины алкилированы
- 26. О-гликозилированный пептид SAPASTTQPIGSTTSTTTK сахарный остаток галактоза (верх) либо фукоза (низ) Сахарный остаток локализован на N-концевом серине
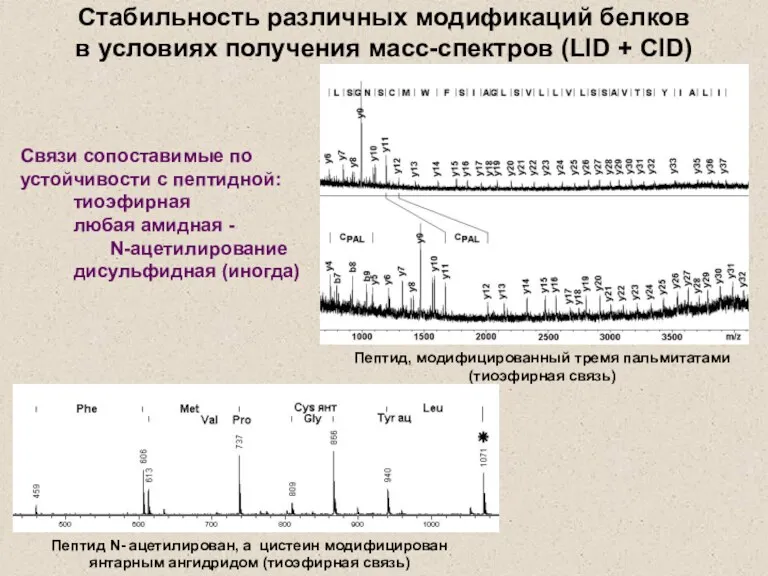
- 27. Стабильность различных модификаций белков в условиях получения масс-спектров (LID + CID) Связи сопоставимые по устойчивости с
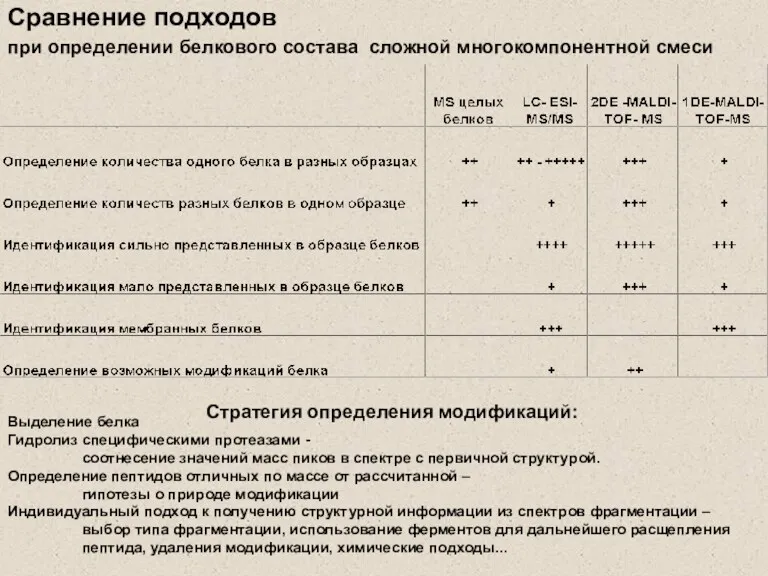
- 28. Сравнение подходов при определении белкового состава сложной многокомпонентной смеси Выделение белка Гидролиз специфическими протеазами - соотнесение
- 30. Скачать презентацию





![Интерпретация спектров распада пептидов TIGTHPSSSAGLK [MH]2+](/_ipx/f_webp&q_80&fit_contain&s_1440x1080/imagesDir/jpg/437335/slide-17.jpg)


 Элементы специальной теории относительности (СТО). Релятивистская кинематика
Элементы специальной теории относительности (СТО). Релятивистская кинематика Электромагнитная индукция. Явление электромагнитной индукции. Правило Ленца
Электромагнитная индукция. Явление электромагнитной индукции. Правило Ленца Атмосферное давление
Атмосферное давление Энергия. Потенциальная и кинетическая энергия тел
Энергия. Потенциальная и кинетическая энергия тел Материя, темная материя, вещество, антивещество
Материя, темная материя, вещество, антивещество Вращение твердого тела вокруг неподвижной оси
Вращение твердого тела вокруг неподвижной оси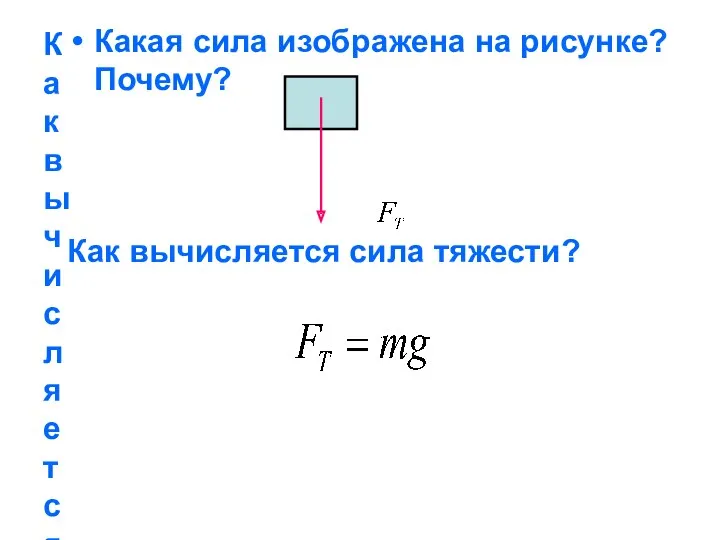 Чему равна сила упругости?
Чему равна сила упругости? Электрическое явление
Электрическое явление Проектирование участка диагностики и обслуживания ходовой части автомобиля
Проектирование участка диагностики и обслуживания ходовой части автомобиля Дозиметрия ионизирующих излучений. Лекция 2
Дозиметрия ионизирующих излучений. Лекция 2 Подшипники скольжения (ПС)
Подшипники скольжения (ПС) Презентация к уроку Диффузия. Движение молекул. (7 класс)
Презентация к уроку Диффузия. Движение молекул. (7 класс) 11кл слайд 3-Меруерт
11кл слайд 3-Меруерт Презентация к уроку физики по теме Напряжённость
Презентация к уроку физики по теме Напряжённость Передачи. Ременные, цепные, фрикционные передачи
Передачи. Ременные, цепные, фрикционные передачи Lektsia_7_PolyarizatsiaMalyus-Bryuster
Lektsia_7_PolyarizatsiaMalyus-Bryuster Силовые агрегаты и двигатели
Силовые агрегаты и двигатели Теплотехника и тепловые установки
Теплотехника и тепловые установки Холодильные машины
Холодильные машины Презентация-игра, 7-8 класс
Презентация-игра, 7-8 класс Датчик температуры охлаждающей жидкости
Датчик температуры охлаждающей жидкости Ustroystvo_reaktora_RTs-1
Ustroystvo_reaktora_RTs-1 Цели и задачи технической диагностики. Основные понятия, термины и определения диагностики
Цели и задачи технической диагностики. Основные понятия, термины и определения диагностики Техническое обслуживание и текущий ремонт переднего моста и рулевого управления автомобиля
Техническое обслуживание и текущий ремонт переднего моста и рулевого управления автомобиля Резистор, конденсатор, катушка индуктивности в цепи переменного тока
Резистор, конденсатор, катушка индуктивности в цепи переменного тока Background radiation
Background radiation Основы радиационной дозиметрии
Основы радиационной дозиметрии История освоения электричества
История освоения электричества