Содержание
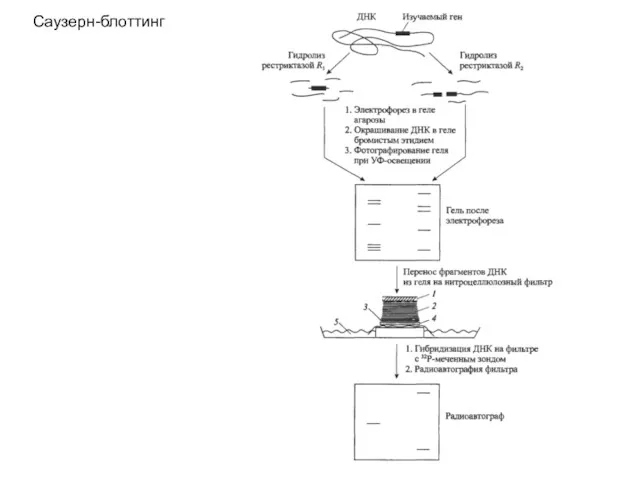
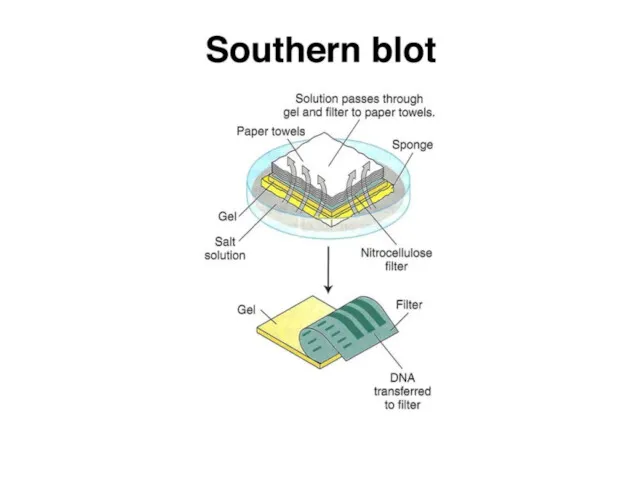
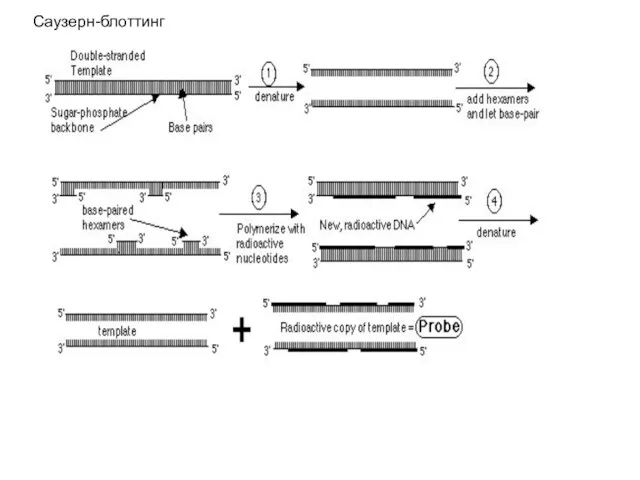
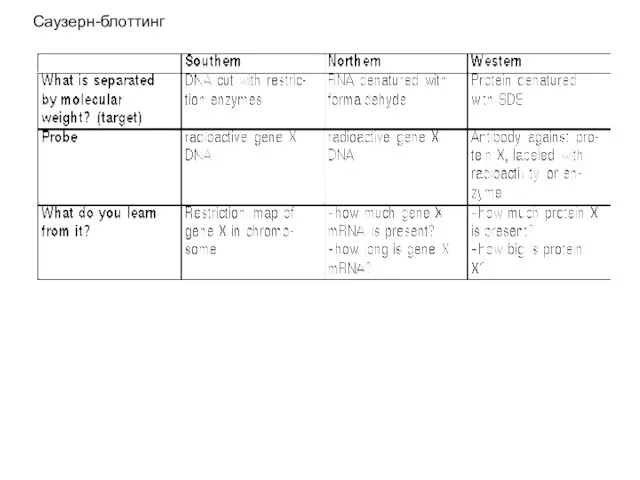
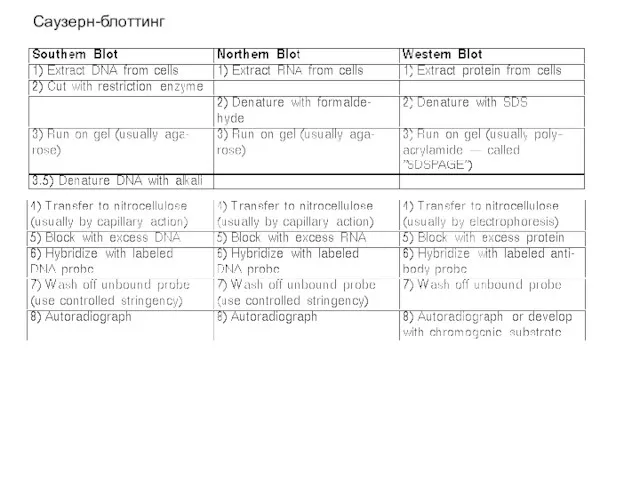
- 2. Саузерн-блоттинг
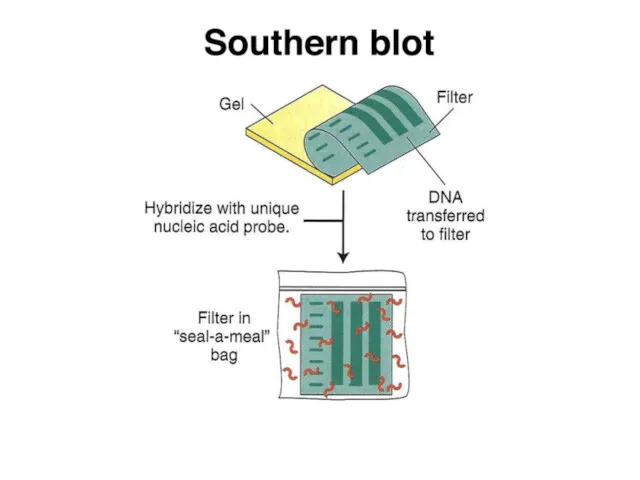
- 3. Саузерн-блоттинг
- 4. Саузерн-блоттинг
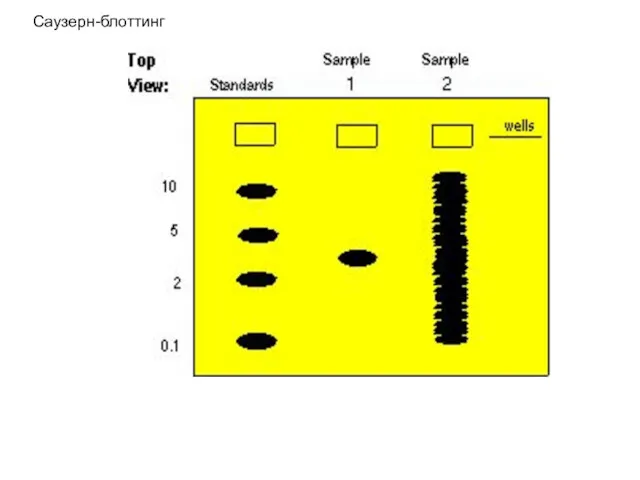
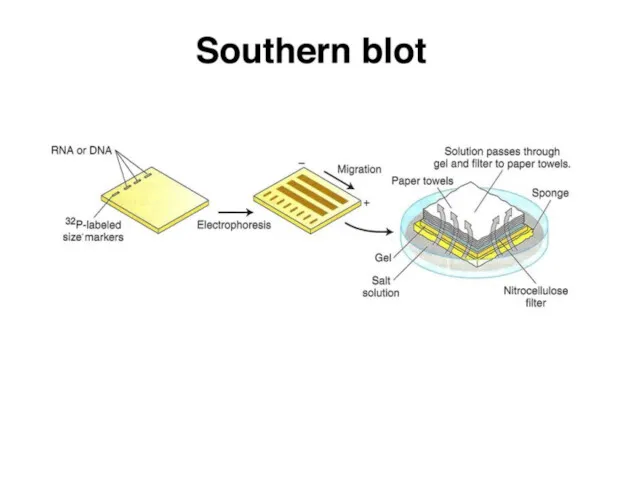
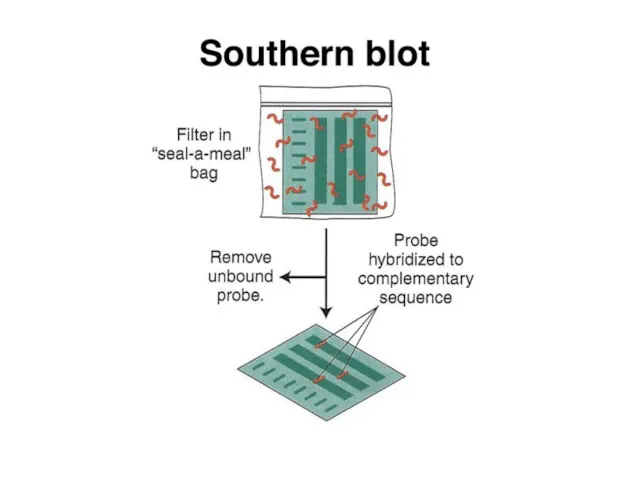
- 10. Саузерн-блоттинг
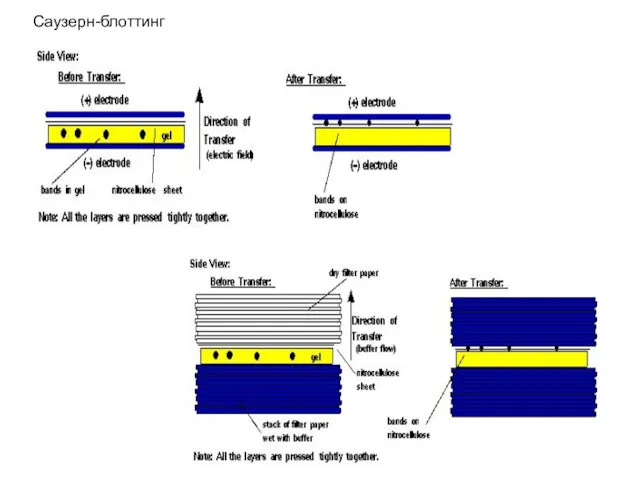
- 11. Саузерн-блоттинг
- 12. Саузерн-блоттинг
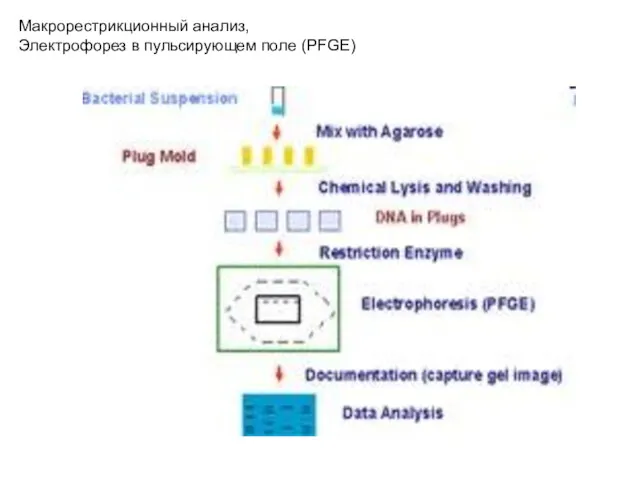
- 13. Макрорестрикционный анализ, Электрофорез в пульсирующем поле (PFGE)
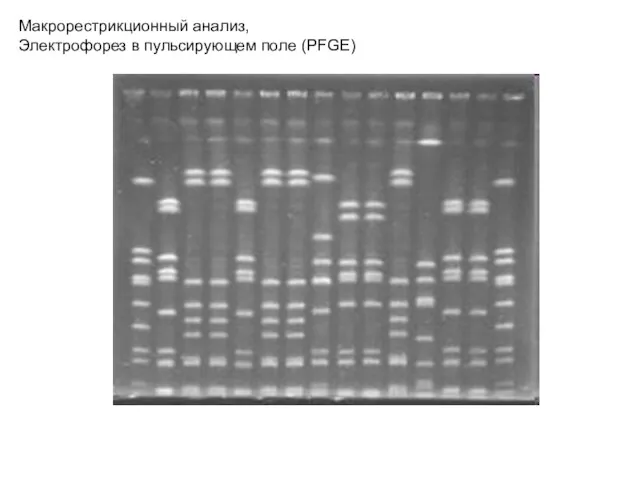
- 14. Макрорестрикционный анализ, Электрофорез в пульсирующем поле (PFGE)
- 15. Макрорестрикционный анализ, Электрофорез в пульсирующем поле Assignment of Staphylococcus Isolates to Groups by spa Typing, SmaI
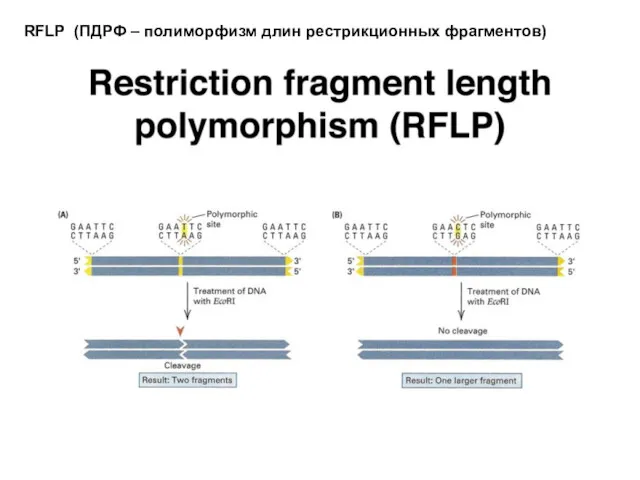
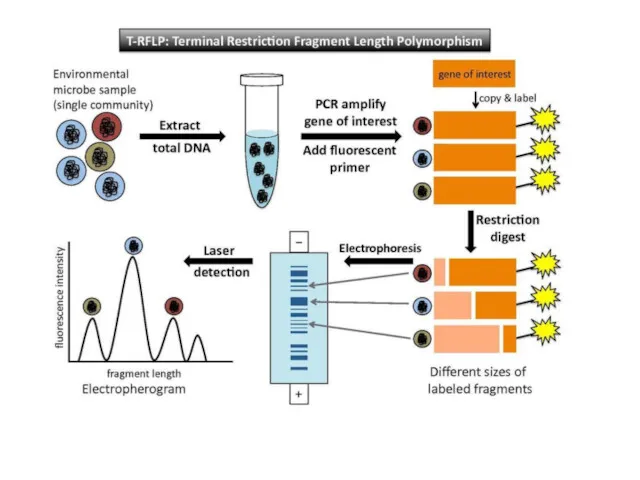
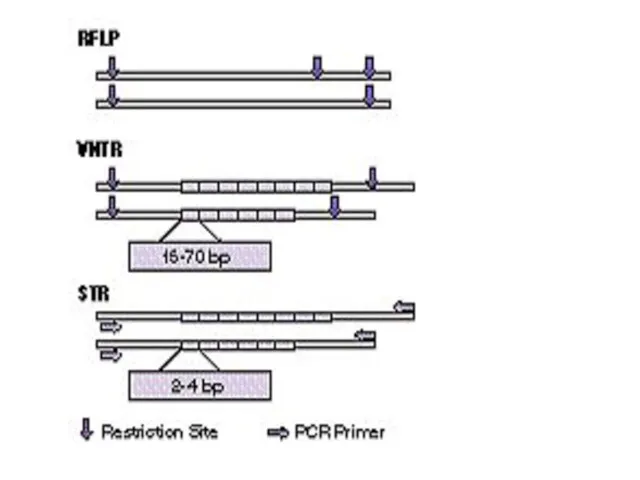
- 16. RFLP (ПДРФ – полиморфизм длин рестрикционных фрагментов)
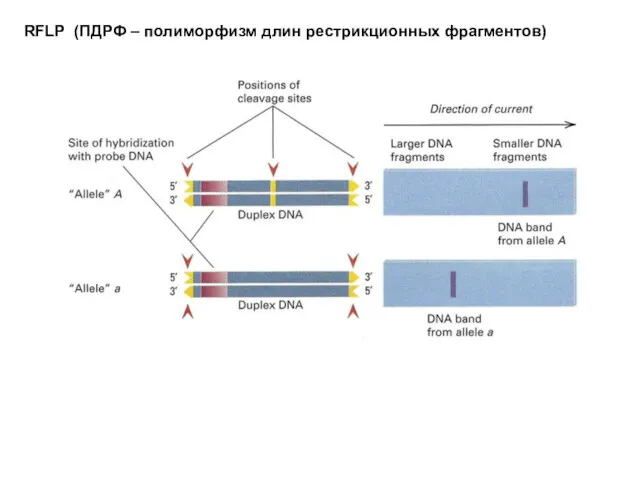
- 17. RFLP (ПДРФ – полиморфизм длин рестрикционных фрагментов)
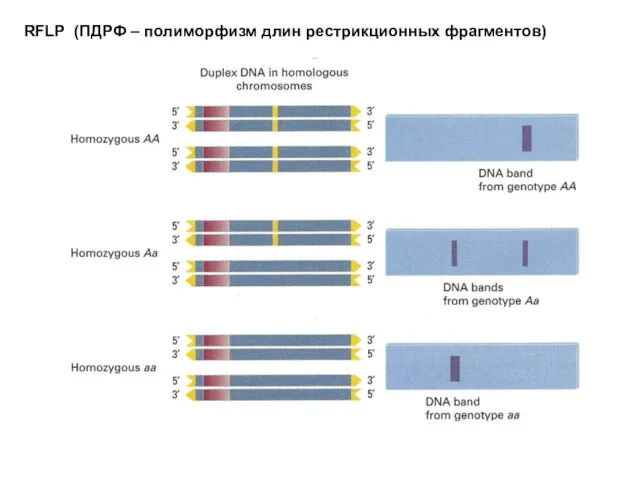
- 18. RFLP (ПДРФ – полиморфизм длин рестрикционных фрагментов)
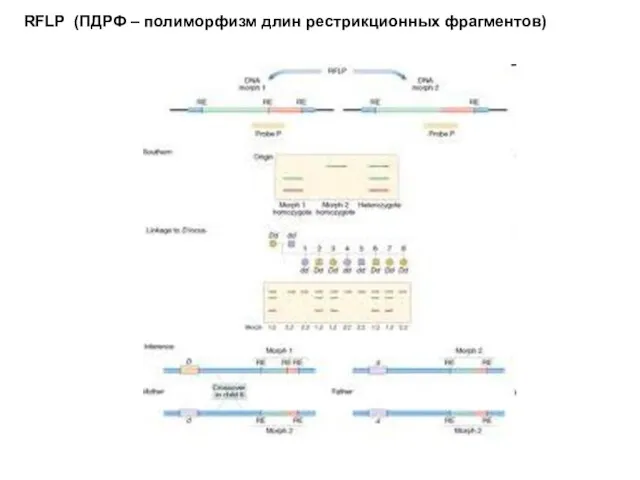
- 19. RFLP (ПДРФ – полиморфизм длин рестрикционных фрагментов)
- 20. Подбор праймеров, специфичных к участкам гена dnaA Примечание: В скобках указан номер нуклеотида в последовательности гена,
- 21. Типирование методом локус-специфичной ПЦР-ПДРФ Семейство Xanthomonadaceae Профили фрагментов рестрикции амплификата гена dnaA С использованием рестриктазы Aat2
- 22. Семейство Xanthomonadaceae Профили фрагментов рестрикции амплификата гена dnaA С использованием рестриктазы Sac1 Дорожки 1 – 16
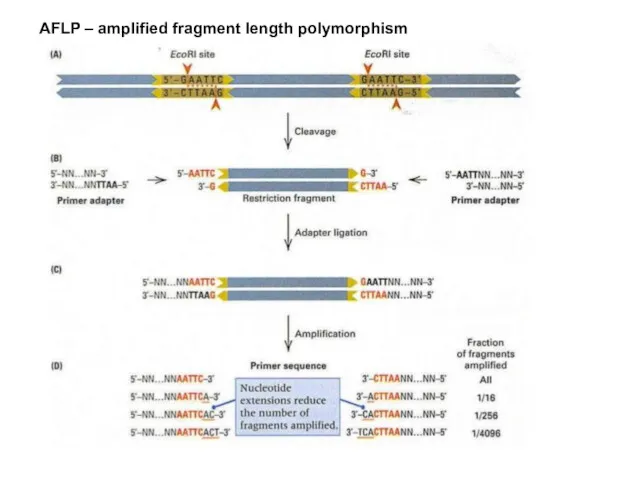
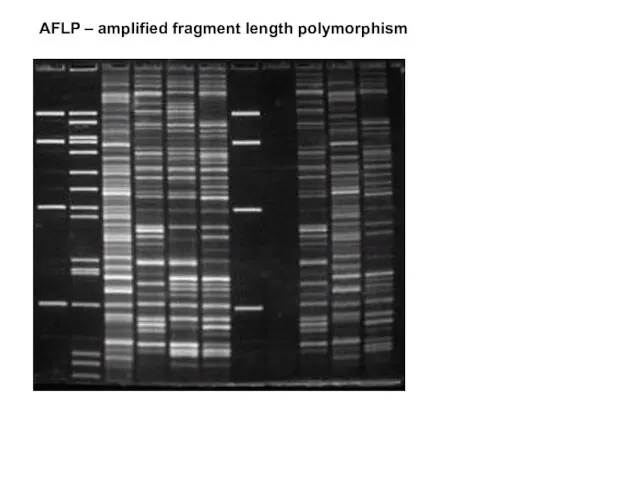
- 24. AFLP – amplified fragment length polymorphism
- 25. AFLP – amplified fragment length polymorphism
- 26. AFLP – amplified fragment length polymorphism
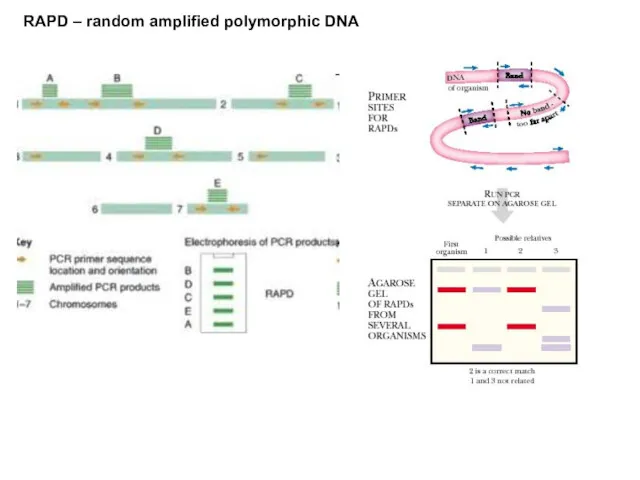
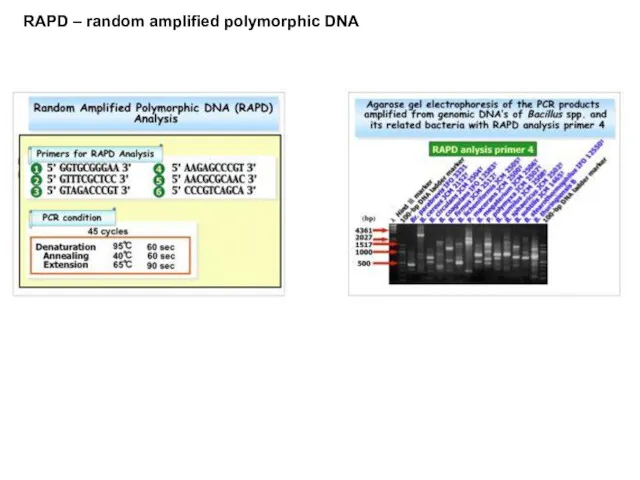
- 27. RAPD – random amplified polymorphic DNA
- 28. RAPD – random amplified polymorphic DNA
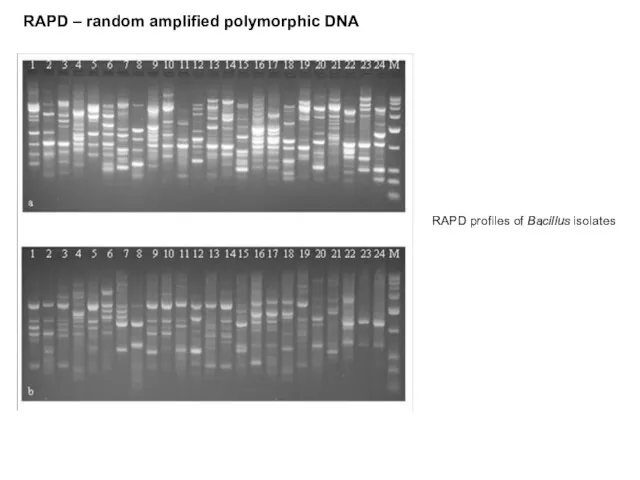
- 29. RAPD – random amplified polymorphic DNA RAPD profiles of Bacillus isolates
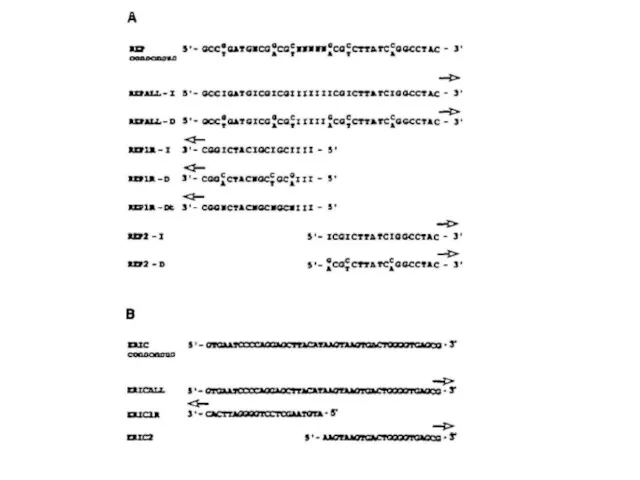
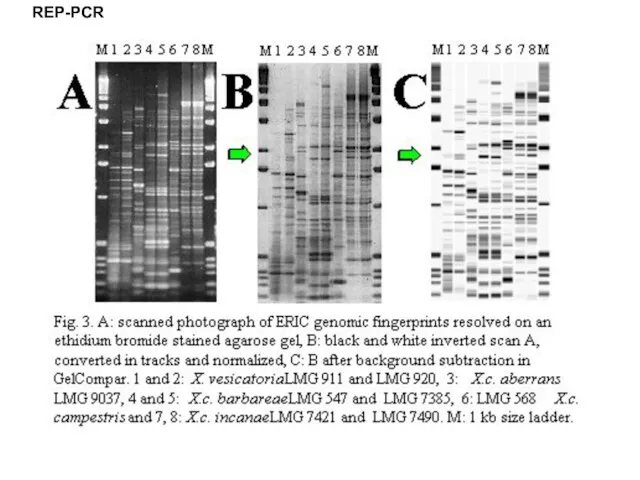
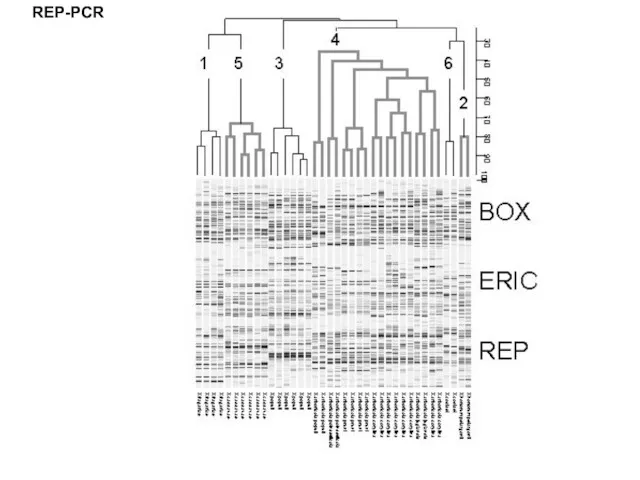
- 30. REP-PCR
- 32. REP-PCR
- 33. REP-PCR
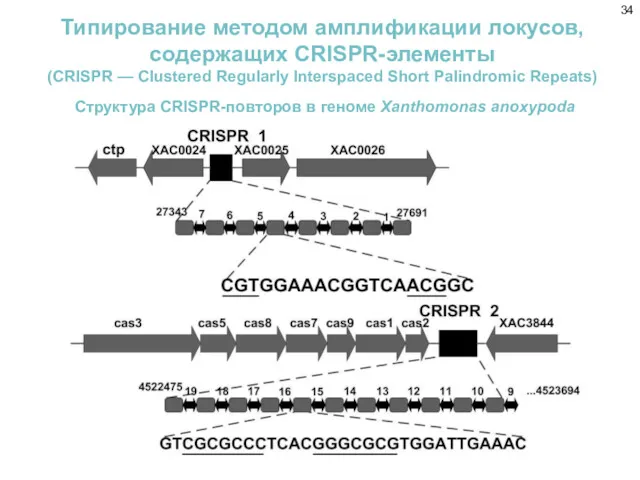
- 34. Типирование методом амплификации локусов, содержащих CRISPR-элементы (CRISPR — Clustered Regularly Interspaced Short Palindromic Repeats) Структура CRISPR-повторов
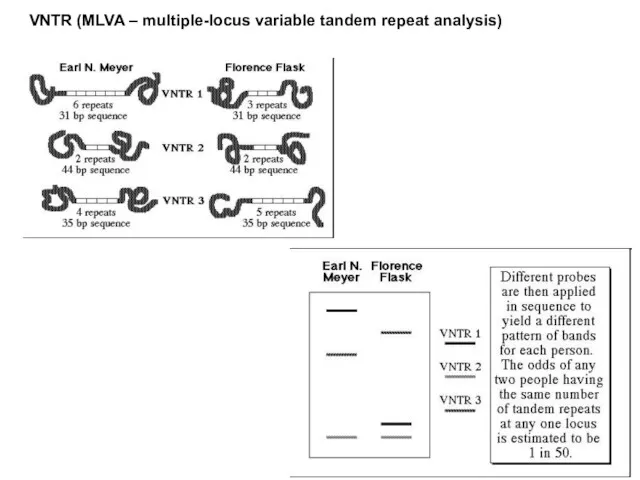
- 35. VNTR (MLVA – multiple-locus variable tandem repeat analysis)
- 36. Alignment of Lpms37 repeats and flanking sequences in the three reference strains and three unrelated isolates
- 38. Методы генотипирования микроорганизмов
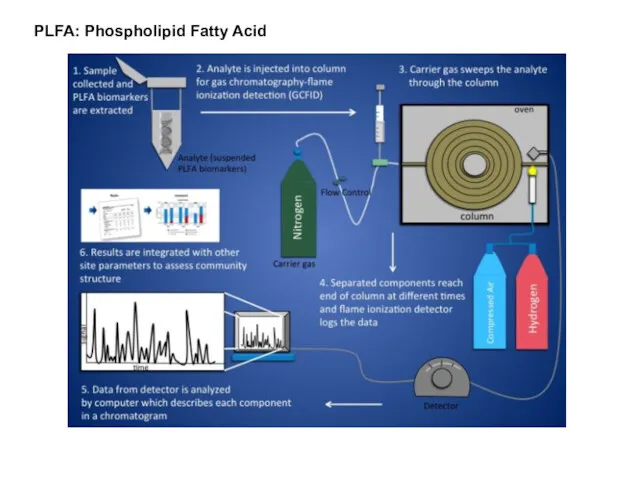
- 39. PLFA: Phospholipid Fatty Acid
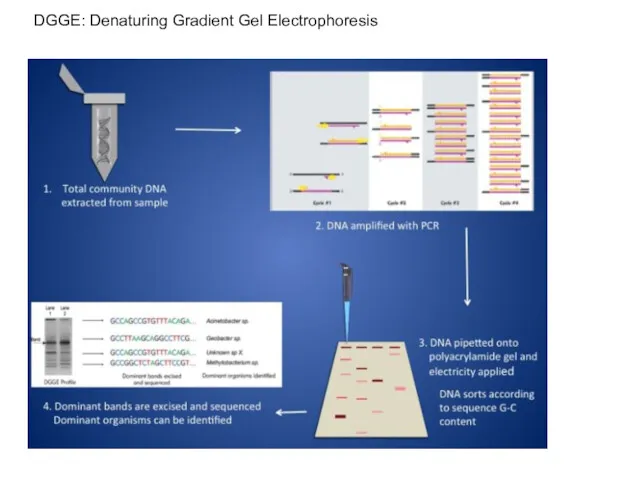
- 40. DGGE: Denaturing Gradient Gel Electrophoresis
- 42. Скачать презентацию









 Скелет – опора организма
Скелет – опора организма Будова тварин: тканини
Будова тварин: тканини Ядовитые грибы
Ядовитые грибы Взаимодействие неаллельных генов
Взаимодействие неаллельных генов Динозавры
Динозавры Животные. Суслики
Животные. Суслики Гиацинт (лат. Hyacínthus)
Гиацинт (лат. Hyacínthus) Естественный отбор – главная движущая сила эволюции
Естественный отбор – главная движущая сила эволюции Бархатцы. Описание растения
Бархатцы. Описание растения Пищеварение в тонком кишечнике
Пищеварение в тонком кишечнике Биология білімінің мақсаттары және мазмұны
Биология білімінің мақсаттары және мазмұны Ферменты: строение, свойства, функции
Ферменты: строение, свойства, функции Для начинающих улитководов
Для начинающих улитководов Общая характеристика грибов
Общая характеристика грибов Презентация по биологии Тайны рождения
Презентация по биологии Тайны рождения Анамнії та амніоти
Анамнії та амніоти Ас қорытудың маңызы
Ас қорытудың маңызы Обмен липидов - 2
Обмен липидов - 2 Парша яблони и меры борьбы с ней в условиях Уйгурского района Алматинской области
Парша яблони и меры борьбы с ней в условиях Уйгурского района Алматинской области Молекулярные основы реализации генетической информации. Лекция 2
Молекулярные основы реализации генетической информации. Лекция 2 Вестибулярный анализатор
Вестибулярный анализатор Черепно-мозговые нервы
Черепно-мозговые нервы Химический состав растений
Химический состав растений Болотные птицы
Болотные птицы Отряды млекопитающих: Китообразные, Ластоногие, Хоботные, Хищные
Отряды млекопитающих: Китообразные, Ластоногие, Хоботные, Хищные Получение продуцентов с помощью генной инженерии
Получение продуцентов с помощью генной инженерии Презентация Скелет и мускулатура птиц
Презентация Скелет и мускулатура птиц Цитологія. Загальні поняття про цитологію
Цитологія. Загальні поняття про цитологію