Содержание
- 2. План 6 лекций Методы определения размера, массы, олигомерного состояния и гидродинамических свойств белков (EM, AFM, DLS,
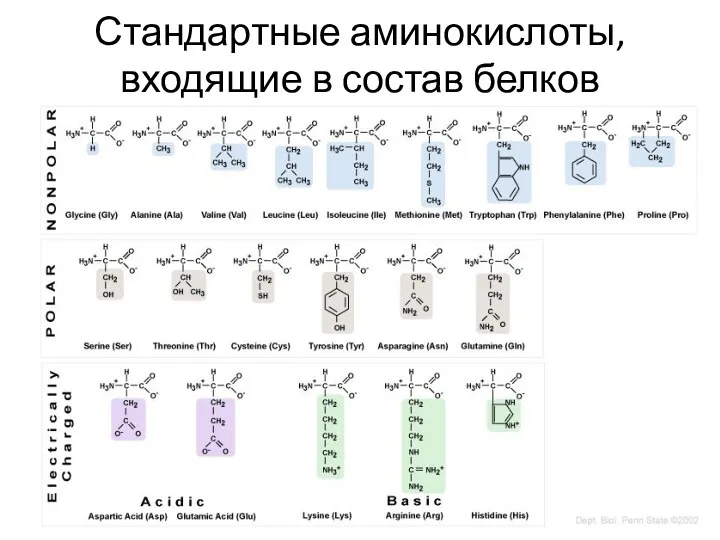
- 3. Стандартные аминокислоты, входящие в состав белков
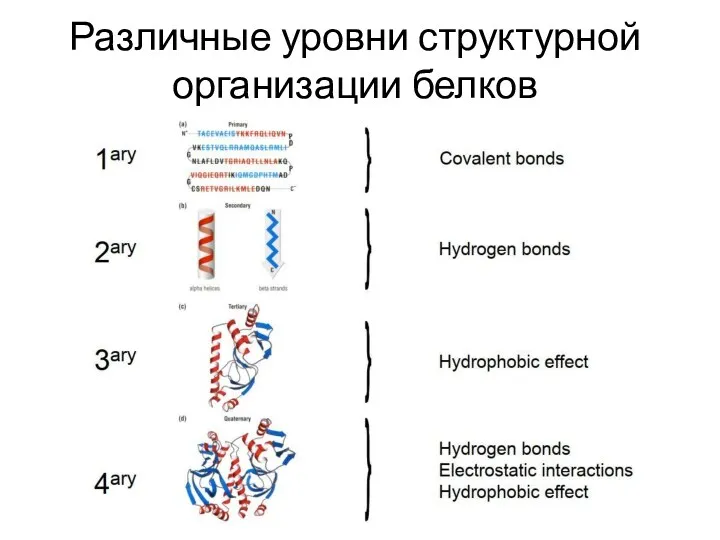
- 4. Различные уровни структурной организации белков
- 5. Мы выделили и очистили рекомбинантный белок, знаем, какая должна быть его последовательность, что дальше? Как ее
- 6. Mass-spectrometry A toolkit of methods to accurately determine masses in a sample Required steps: Ionization is
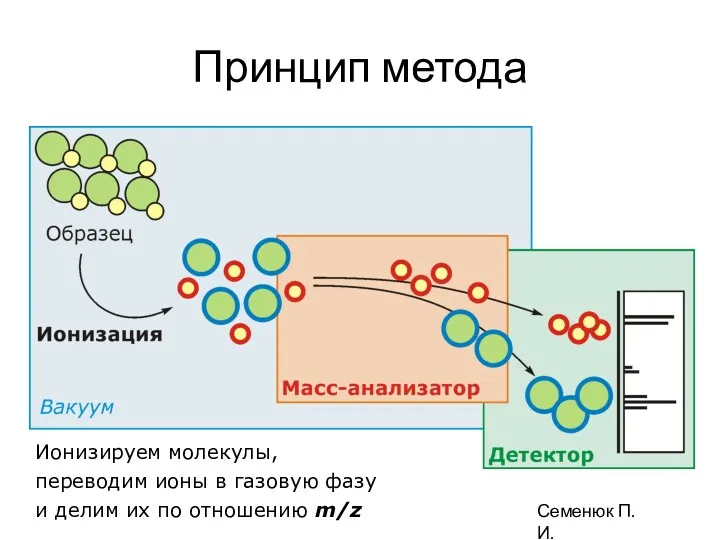
- 7. Принцип метода Ионизируем молекулы, переводим ионы в газовую фазу и делим их по отношению m/z Семенюк
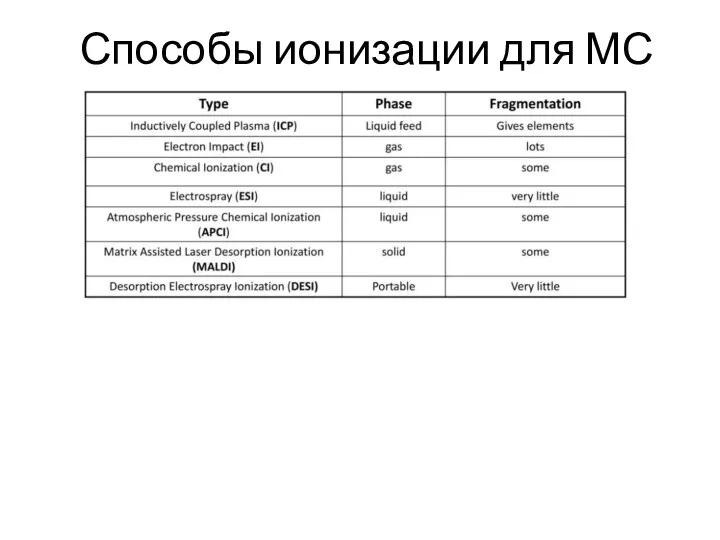
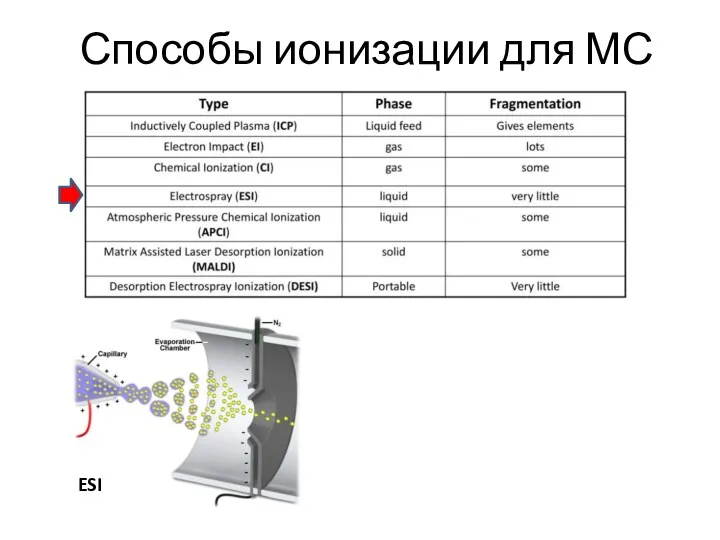
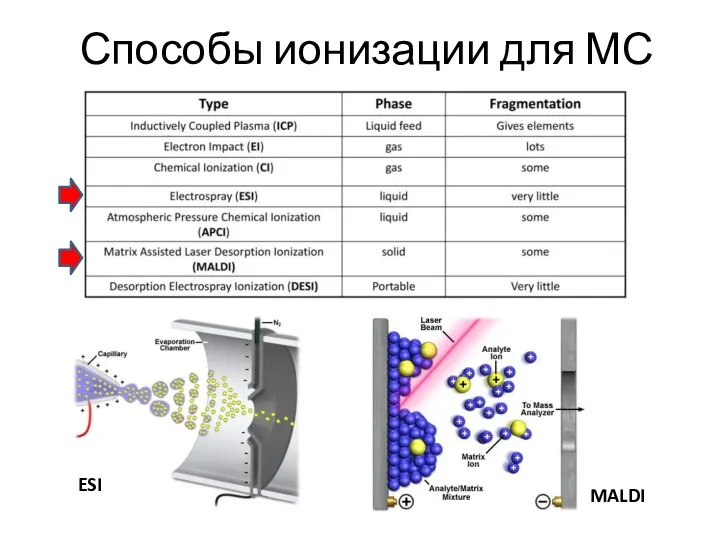
- 8. Способы ионизации для МС
- 9. Способы ионизации для МС ESI
- 10. Способы ионизации для МС ESI MALDI
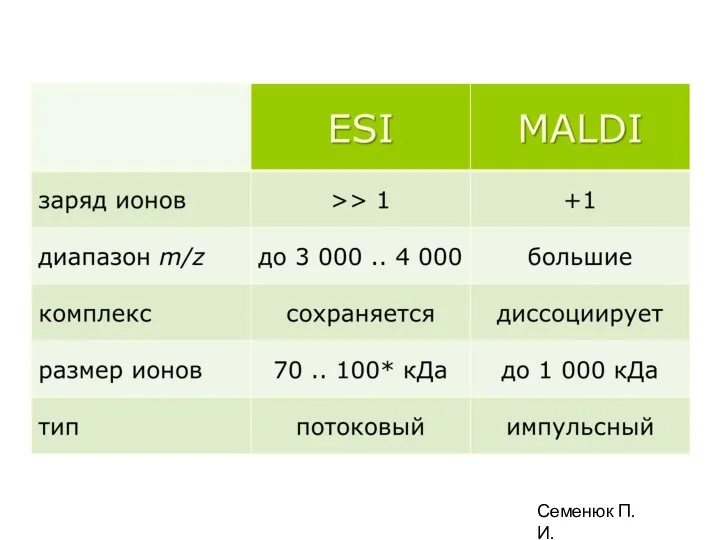
- 11. Семенюк П.И.
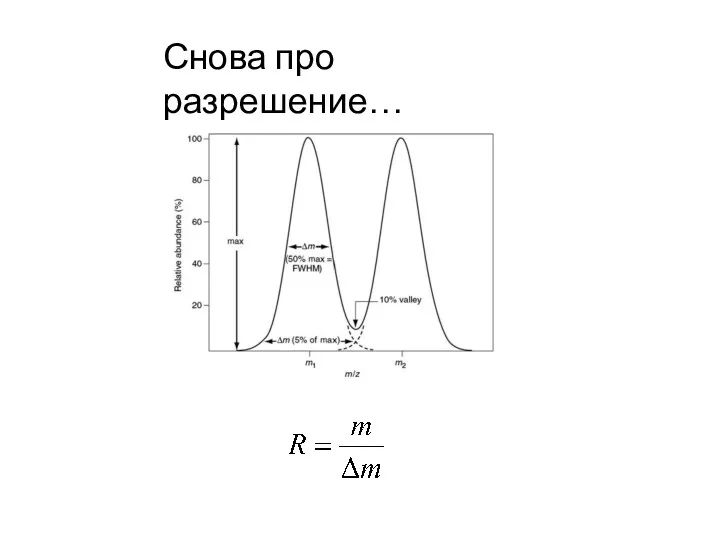
- 12. Снова про разрешение…
- 14. Как посчитать массу иона, зная соседние пики m/z ? Пример
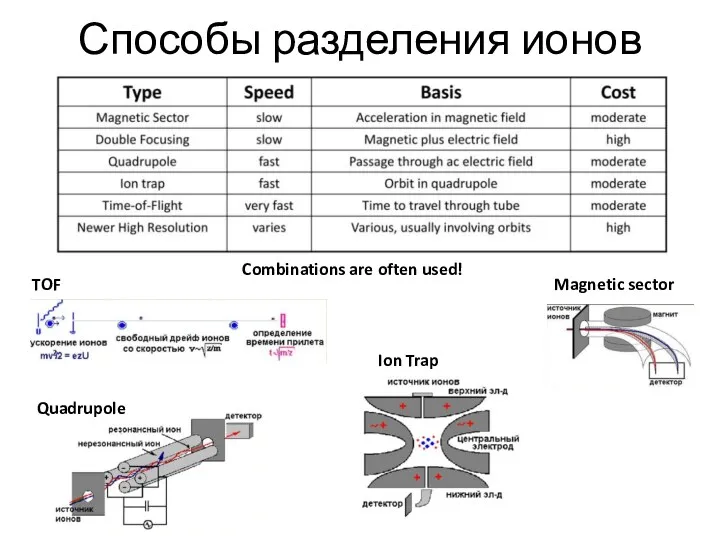
- 15. Способы разделения ионов TOF Quadrupole Ion Trap Magnetic sector Combinations are often used!
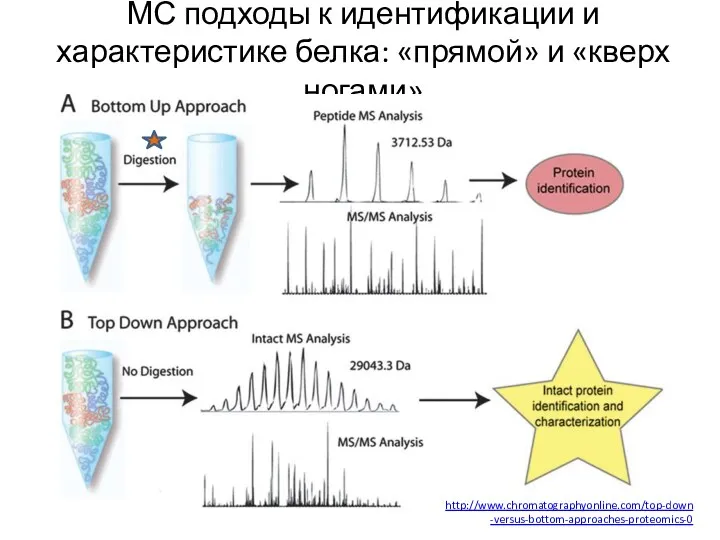
- 16. МС подходы к идентификации и характеристике белка: «прямой» и «кверх ногами» http://www.chromatographyonline.com/top-down-versus-bottom-approaches-proteomics-0
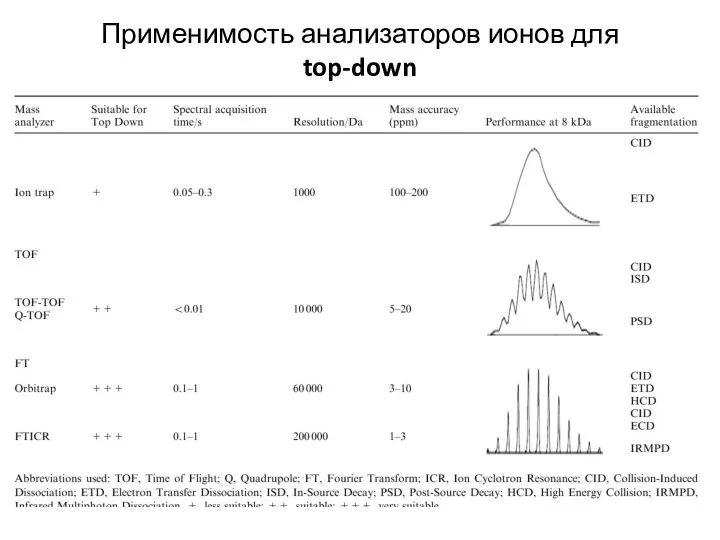
- 17. Применимость анализаторов ионов для top-down
- 18. Two types of the “bottom-up” protein identification Peptide mass fingerprinting after proteolytic digestion and comparison with
- 19. Как правило, однозарядные ионы -> тривиальное определение их массы из m/z
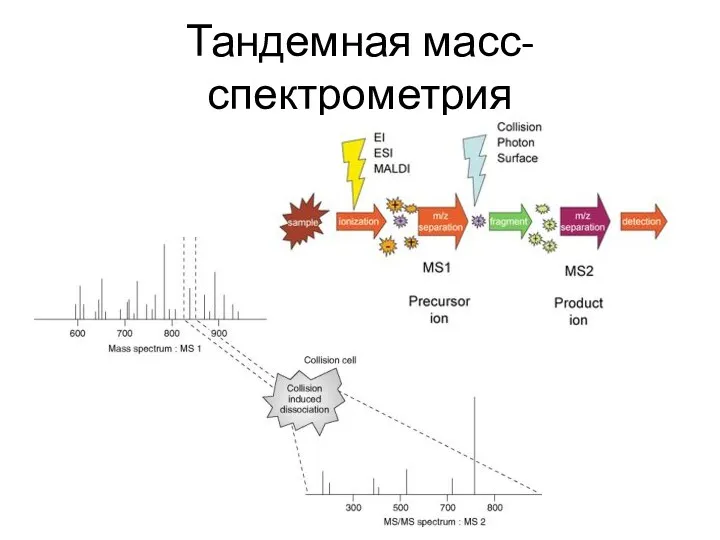
- 20. Тандемная масс-спектрометрия
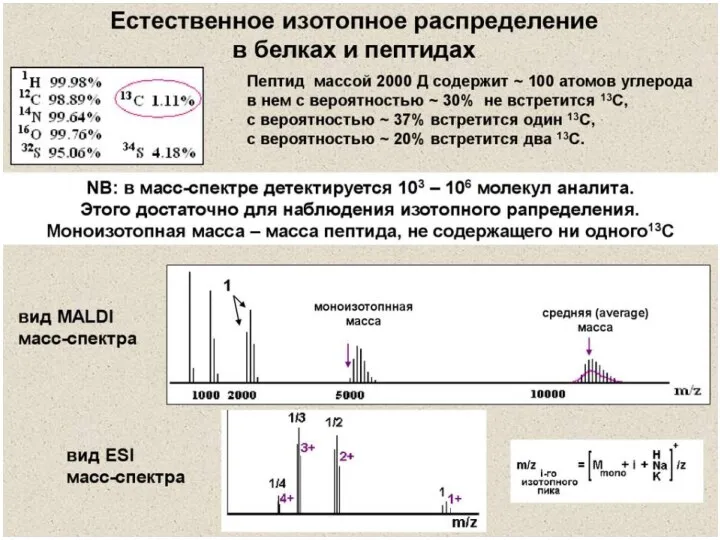
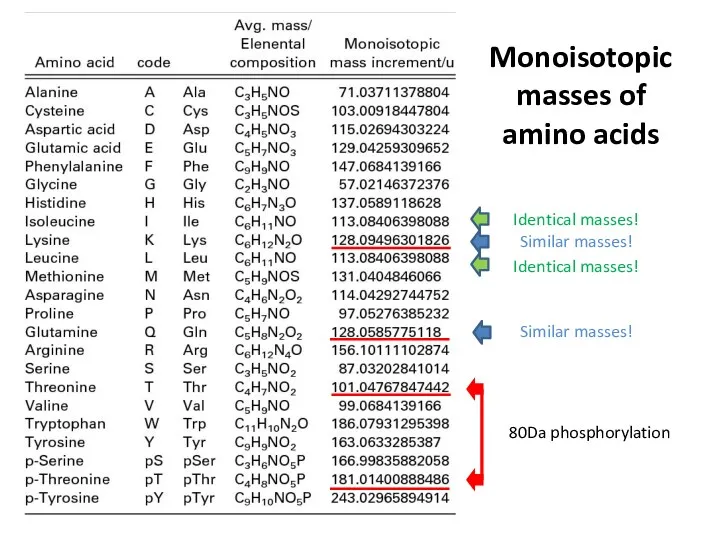
- 21. Monoisotopic masses of amino acids 80Da phosphorylation Similar masses! Identical masses! Identical masses! Similar masses!
- 22. Тандемная масс-спектрометрия
- 23. MS/MS for indentifying terminal truncations in protein sequence >sp|O14558|1-160 MEIPVPVQPSWLRRASAPLPGLSAPGRLFDQRFGEGLLEAELAALCPTTLAPYYLRAPSV ALPVAQVPTDPGHFSVLLDVKHFSPEEIAVKVVGEHVEVHARHEERPDEHGFVAREFHRR YRLPPGVDPAAVTSALSPEGVLSIQAAPASAQAPPPAAAK >sp|O14558|1-160 MEIPVPVQPSWLRRASAPLPGLSAPGRLFDQRFGEGLLEAELAALCPTTLAPYYLRAPSV ALPVAQVPTDPGHFSVLLDVKHFSPEEIAVKVVGEHVEVHARHEERPDEHGFVAREFHRR YRLPPGVDPAAVTSALSPEGVLSIQAAPASAQA
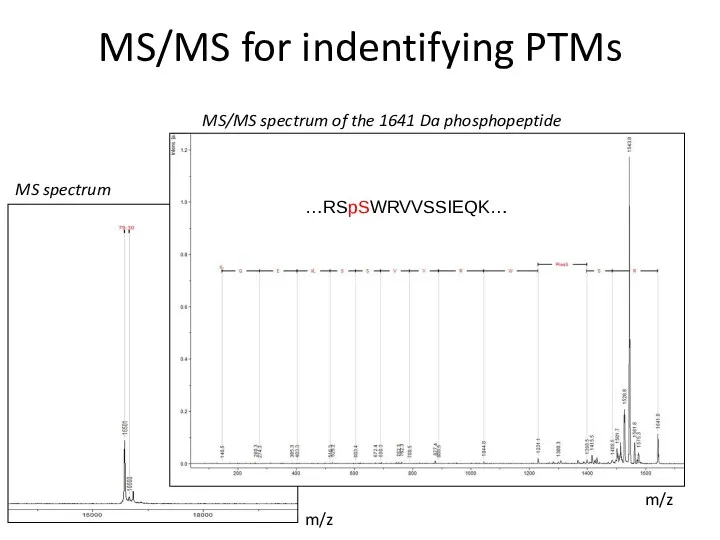
- 24. MS/MS for indentifying PTMs m/z m/z MS spectrum MS/MS spectrum of the 1641 Da phosphopeptide …RSpSWRVVSSIEQK…
- 25. Конец лекции 18.11.19
- 26. Secondary structure elements α-helix β-strand Turns and loops Random coil Protein conformation is stabilized largely by
- 27. Предсказание вторичной структуры белка по его последовательности http://www.compbio.dundee.ac.uk/jpred4/index_up.html
- 28. Secondary structure prediction based on STARD1 protein sequence alpha helix ('H'), beta sheet ('E') or not
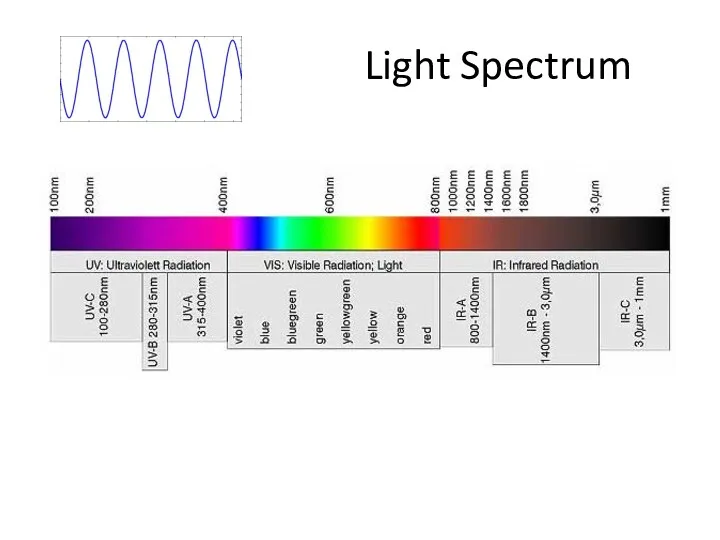
- 29. Light Spectrum
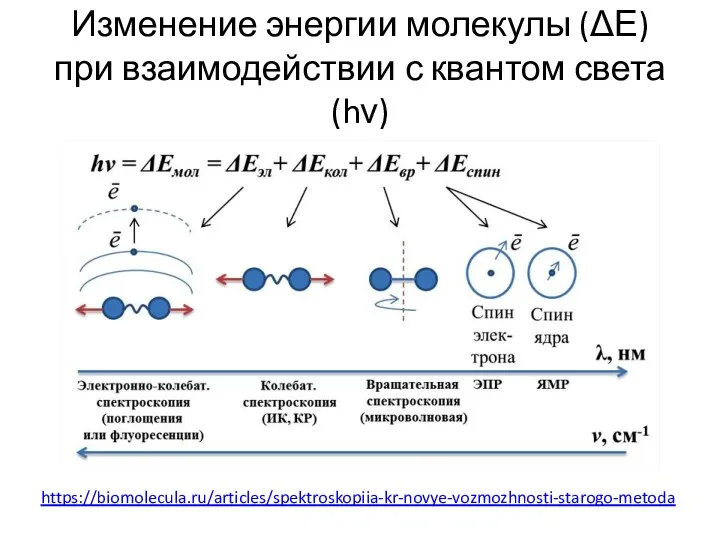
- 30. Изменение энергии молекулы (ΔЕ) при взаимодействии с квантом света (hν) https://biomolecula.ru/articles/spektroskopiia-kr-novye-vozmozhnosti-starogo-metoda
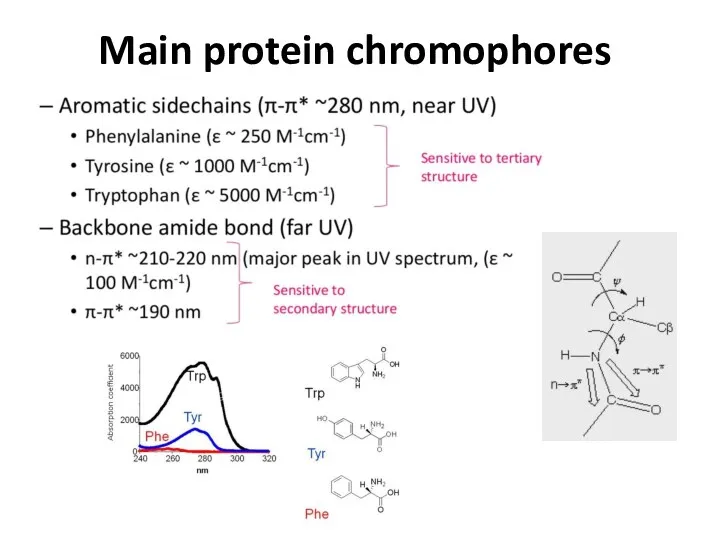
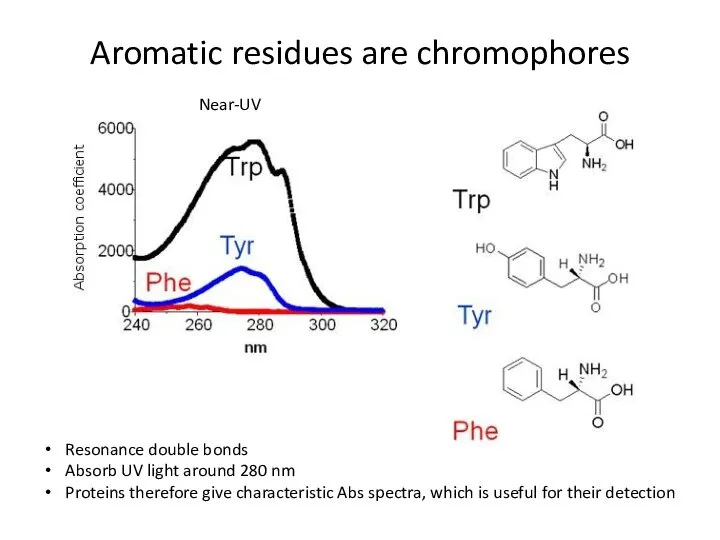
- 31. Main protein chromophores
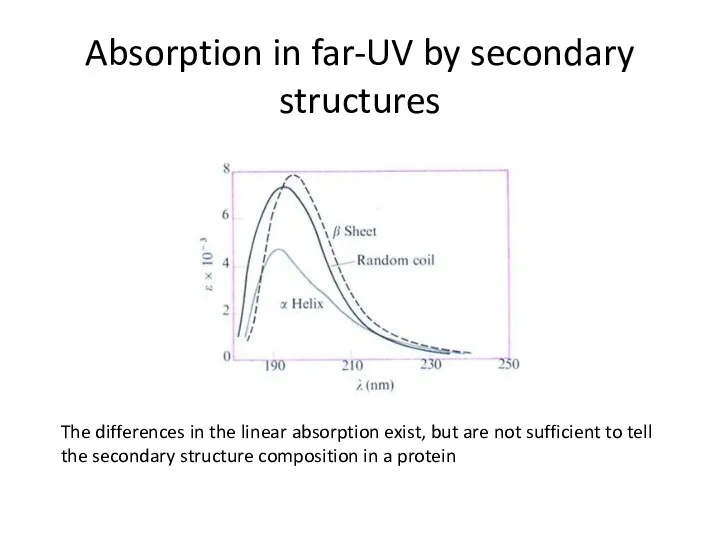
- 32. Absorption in far-UV by secondary structures The differences in the linear absorption exist, but are not
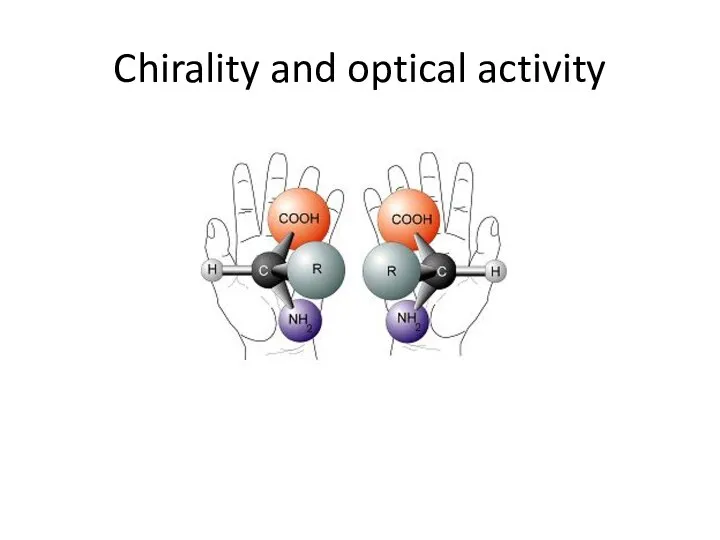
- 33. Chirality and optical activity
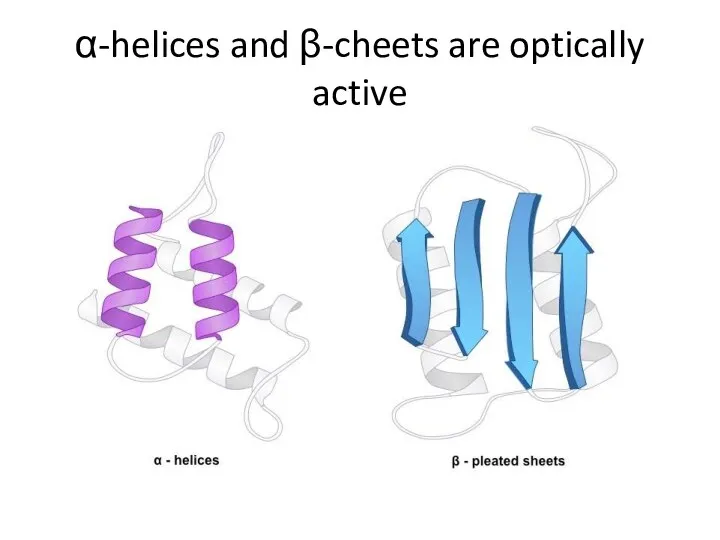
- 34. α-helices and β-cheets are optically active
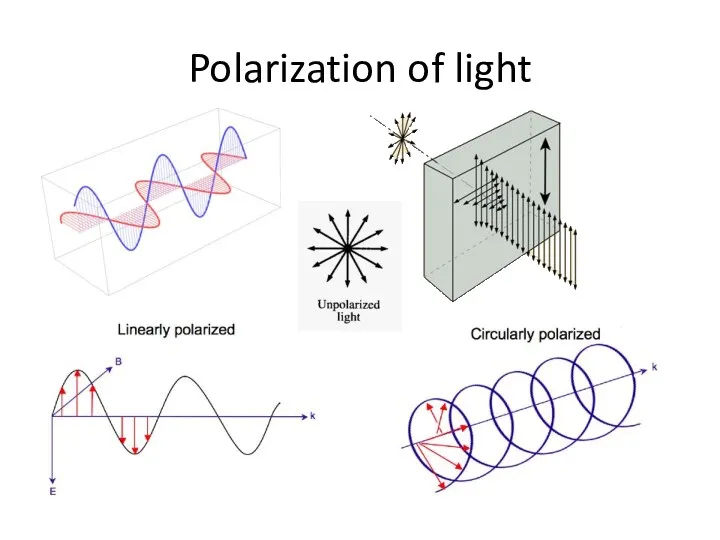
- 35. Polarization of light
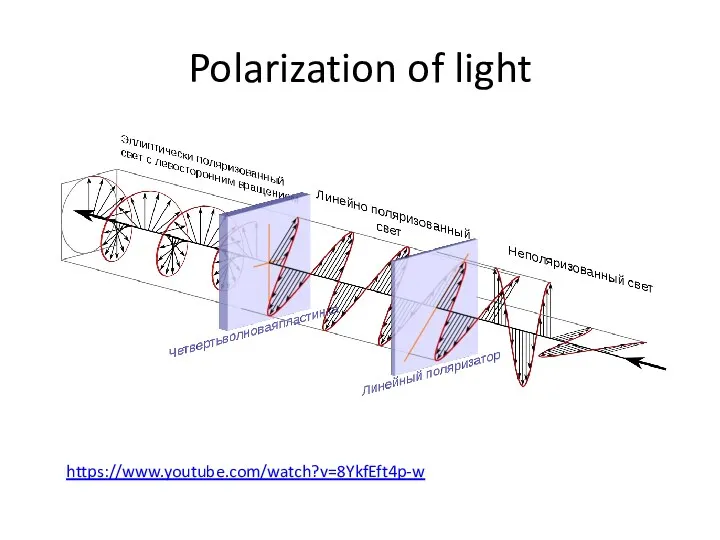
- 36. Polarization of light https://www.youtube.com/watch?v=8YkfEft4p-w
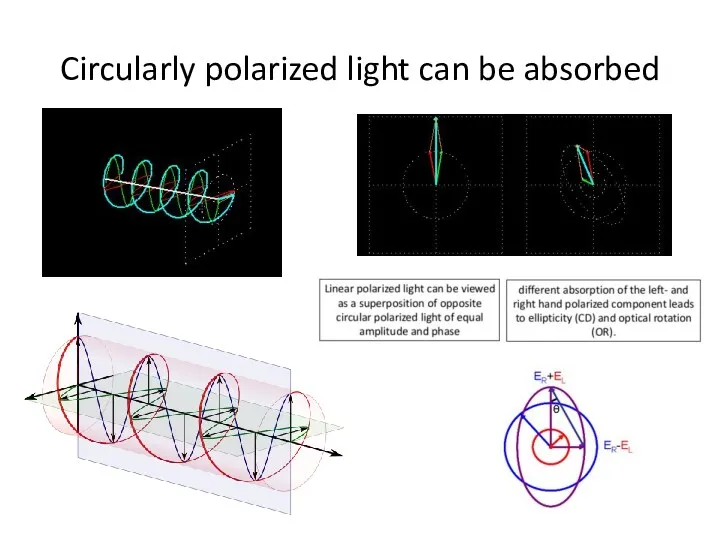
- 37. Circularly polarized light can be absorbed
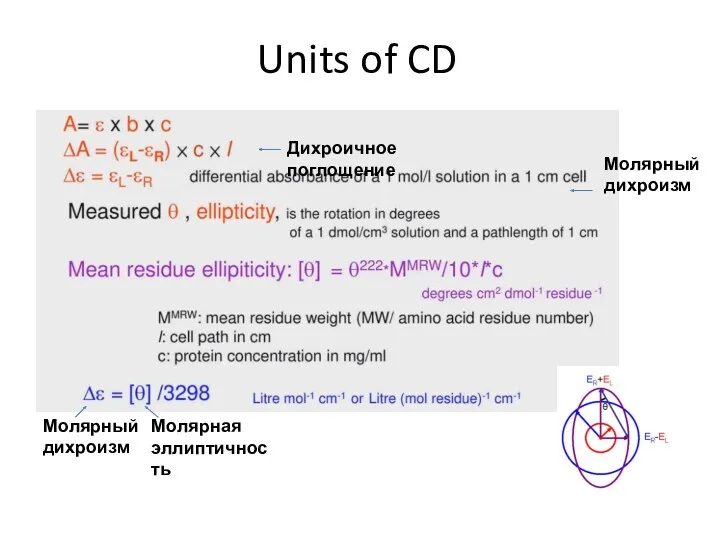
- 38. Units of CD Дихроичное поглощение Молярный дихроизм Молярный дихроизм Молярная эллиптичность
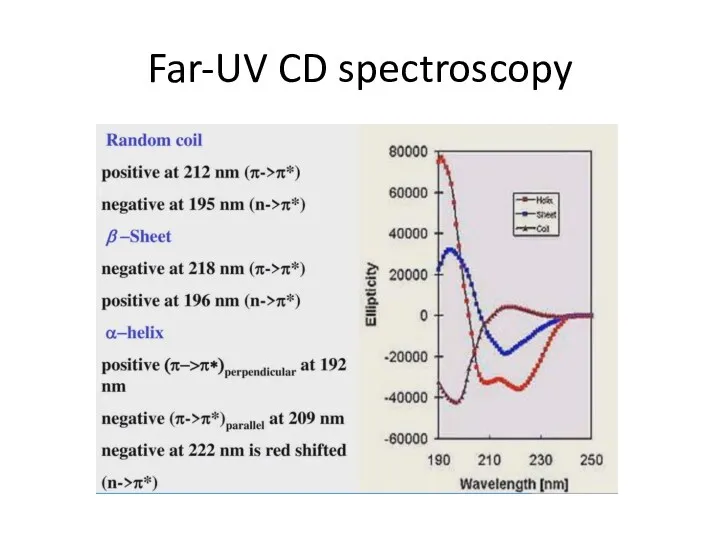
- 39. Far-UV CD spectroscopy
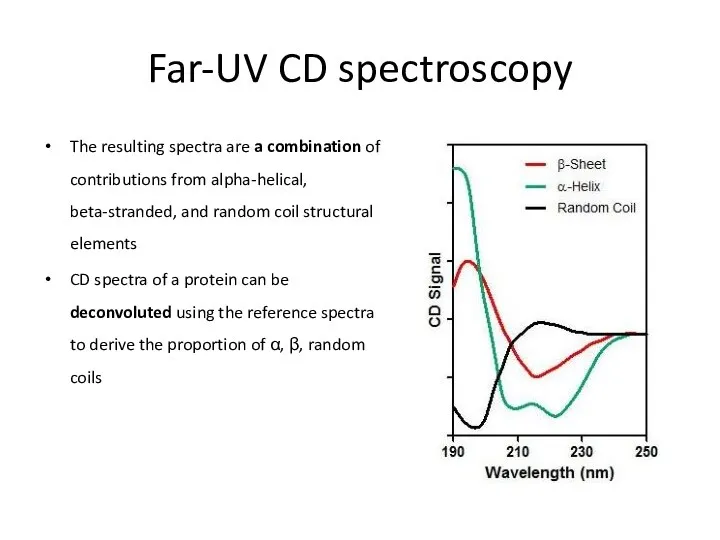
- 40. Far-UV CD spectroscopy The resulting spectra are a combination of contributions from alpha-helical, beta-stranded, and random
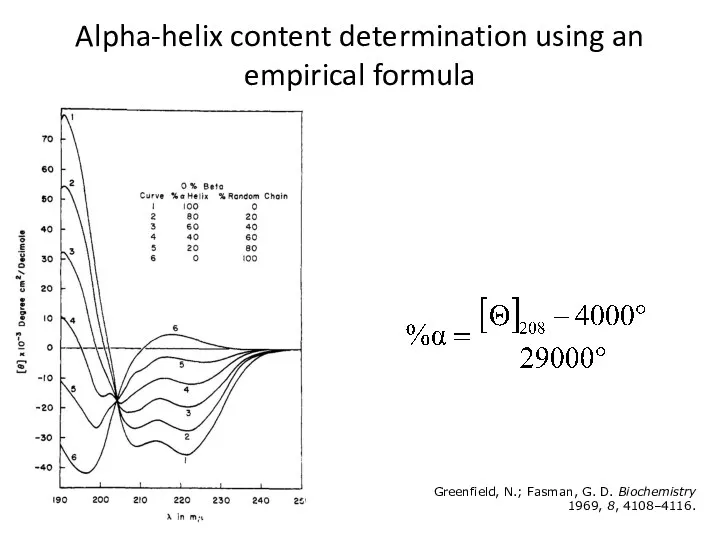
- 41. Alpha-helix content determination using an empirical formula Greenfield, N.; Fasman, G. D. Biochemistry 1969, 8, 4108–4116.
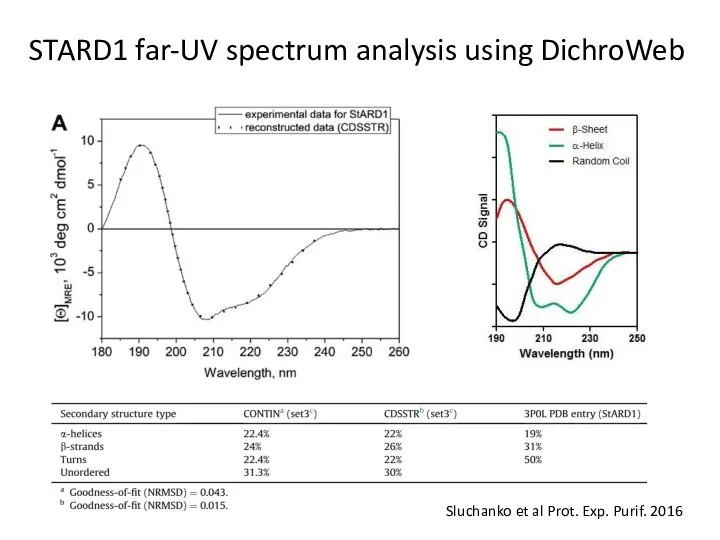
- 43. STARD1 far-UV spectrum analysis using DichroWeb Sluchanko et al Prot. Exp. Purif. 2016
- 44. Applications Determination of 2° structure content in a protein of interest The effect of ligand binding
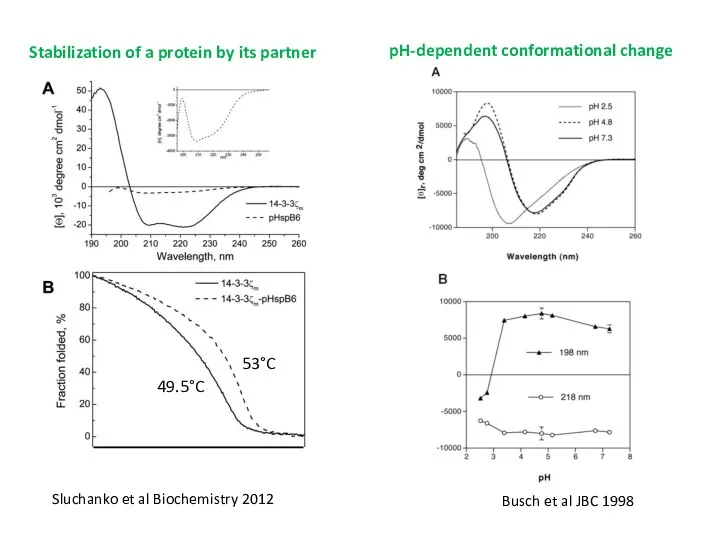
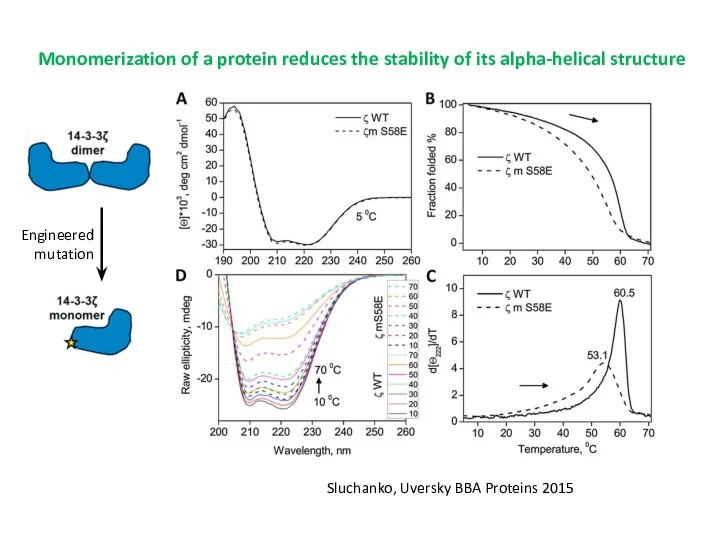
- 45. Stabilization of a protein by its partner Sluchanko et al Biochemistry 2012 49.5°C 53°C pH-dependent conformational
- 46. Monomerization of a protein reduces the stability of its alpha-helical structure Sluchanko, Uversky BBA Proteins 2015
- 47. Far-UV CD Very convenient, sensitive, non-invasive technique Small sample consumption (50-100 µl, 0.5-1 mg/ml), sample can
- 48. Aromatic residues are chromophores Resonance double bonds Absorb UV light around 280 nm Proteins therefore give
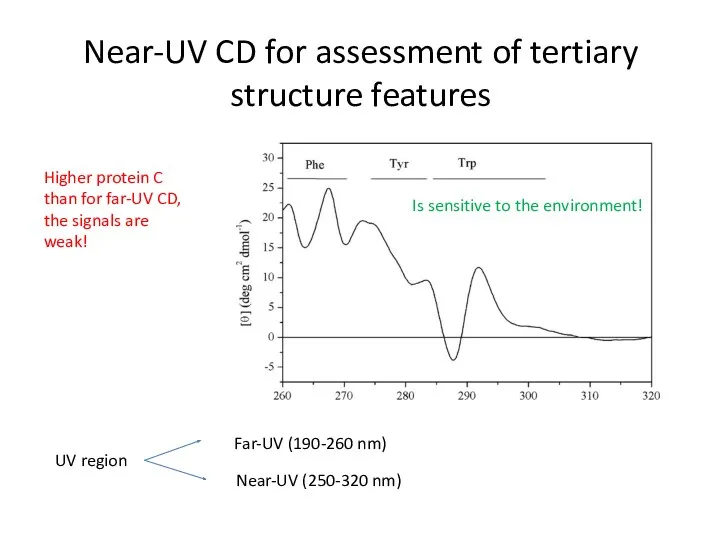
- 49. Near-UV CD for assessment of tertiary structure features Is sensitive to the environment! Higher protein C
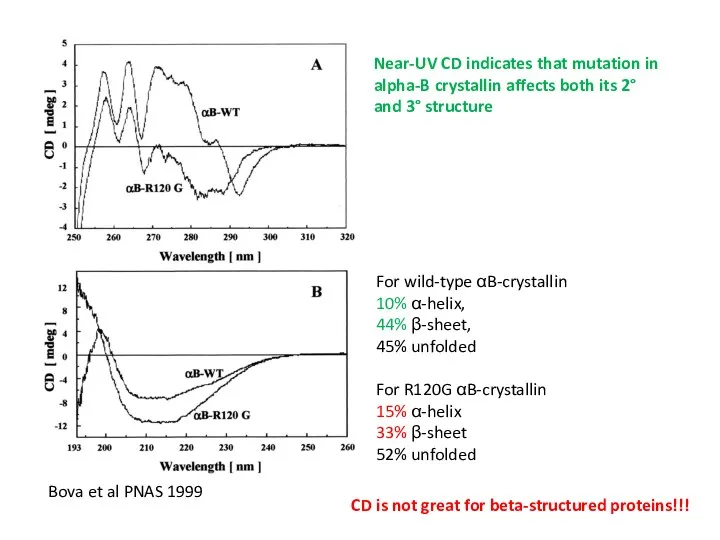
- 50. Bova et al PNAS 1999 For wild-type αB-crystallin 10% α-helix, 44% β-sheet, 45% unfolded For R120G
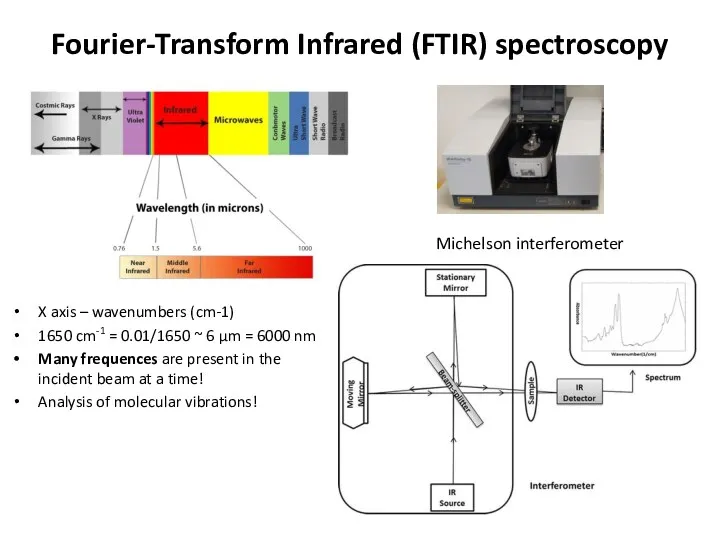
- 51. Fourier-Transform Infrared (FTIR) spectroscopy X axis – wavenumbers (cm-1) 1650 cm-1 = 0.01/1650 ~ 6 µm
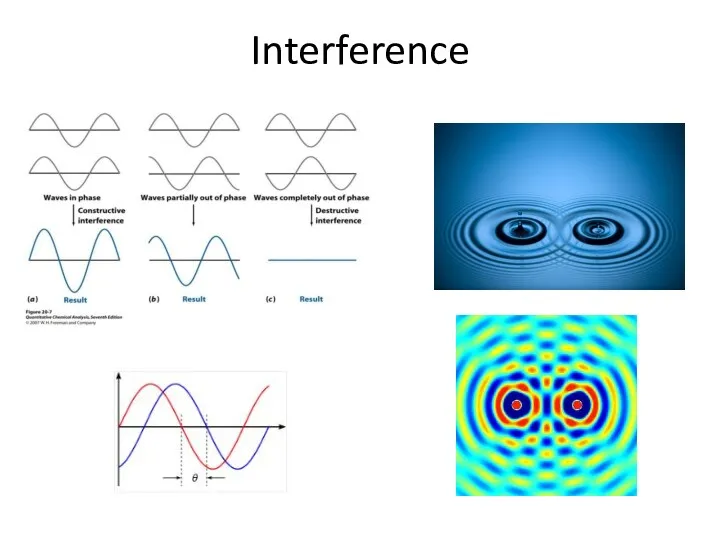
- 52. Interference
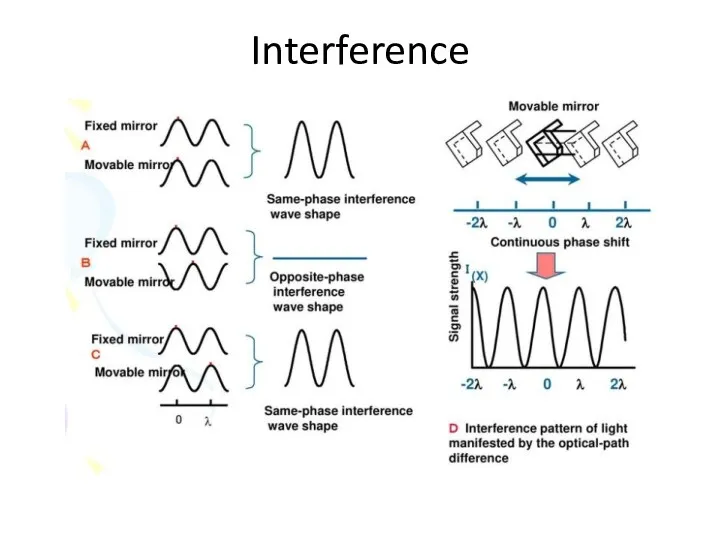
- 53. Interference
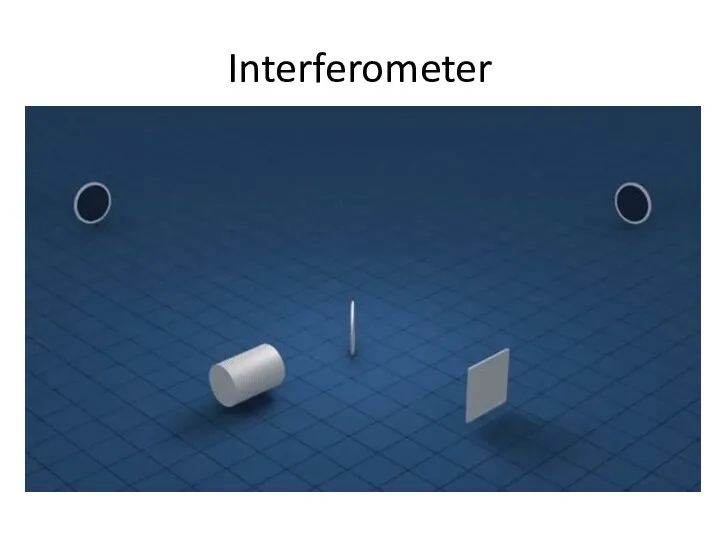
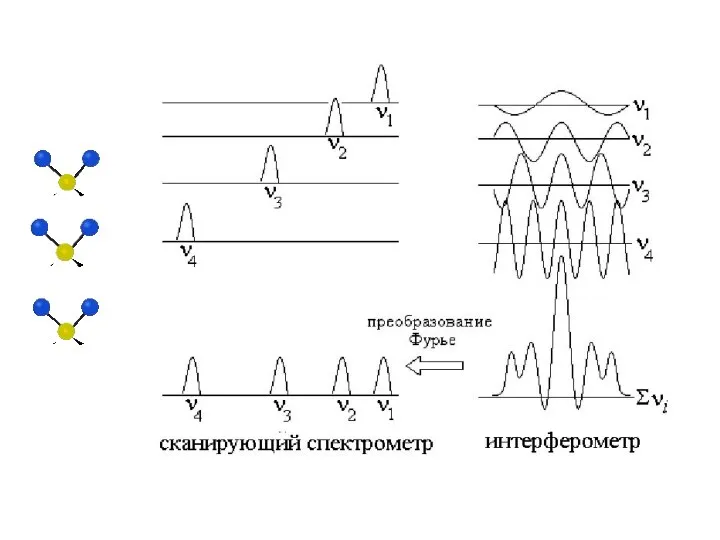
- 54. Interferometer
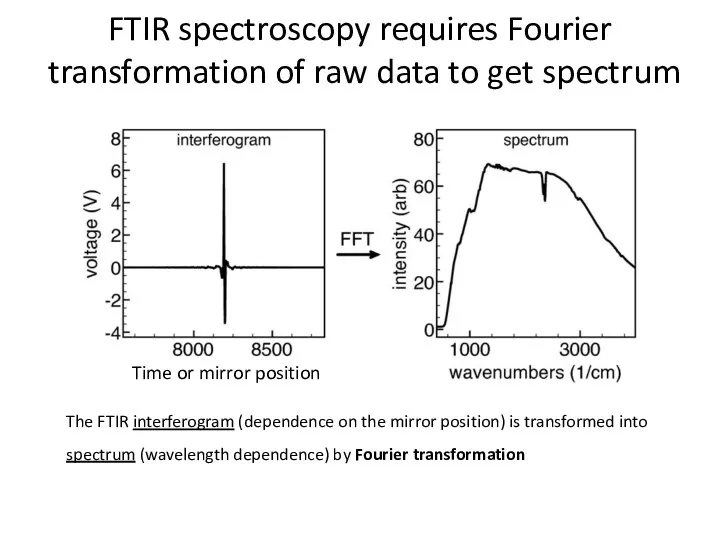
- 55. FTIR spectroscopy requires Fourier transformation of raw data to get spectrum The FTIR interferogram (dependence on
- 56. Fourier transform Signal in time domain Signal in frequency domain
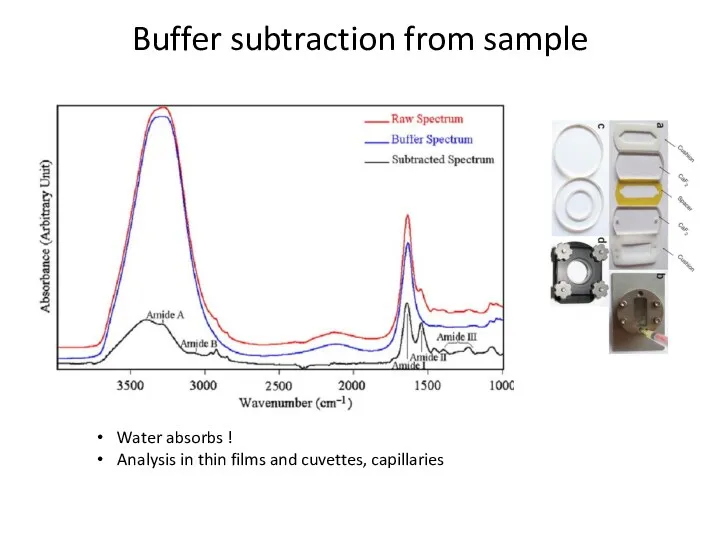
- 58. Buffer subtraction from sample Water absorbs ! Analysis in thin films and cuvettes, capillaries
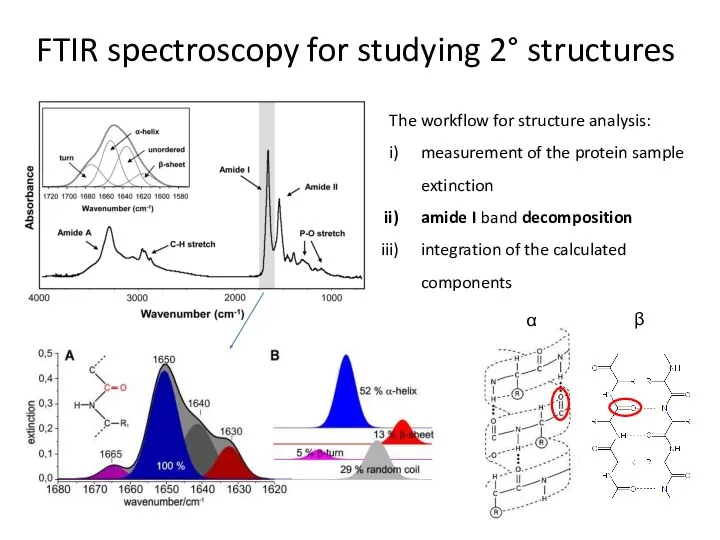
- 59. FTIR spectroscopy for studying 2° structures The workflow for structure analysis: measurement of the protein sample
- 60. Protein bands in a FTIR spectrum
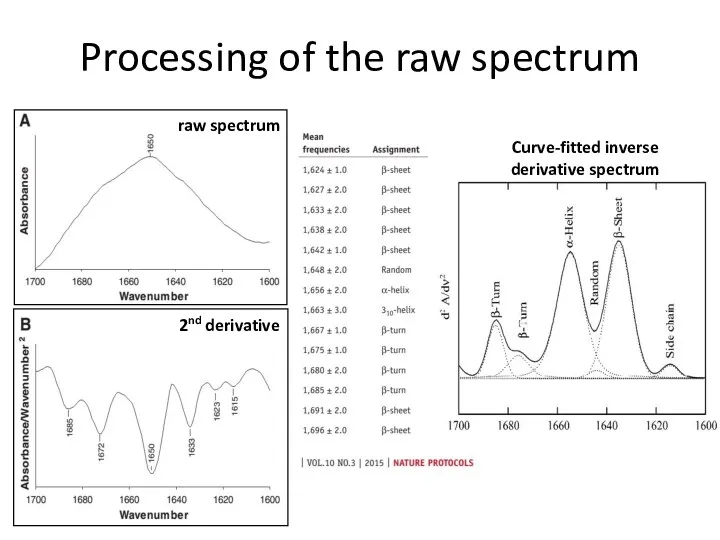
- 61. Processing of the raw spectrum 2nd derivative raw spectrum Curve-fitted inverse derivative spectrum
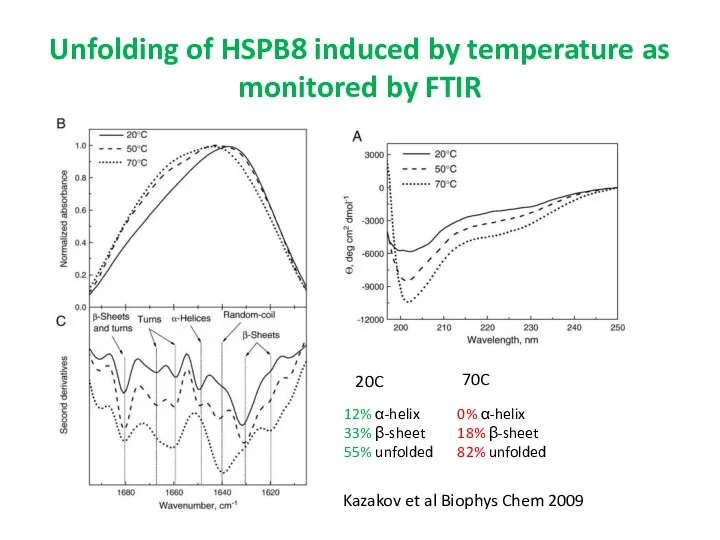
- 62. Unfolding of HSPB8 induced by temperature as monitored by FTIR Kazakov et al Biophys Chem 2009
- 63. Raman spectroscopy Complimentary to IR spectroscopy, but relies on scattering instead of absorption Possibility to measure
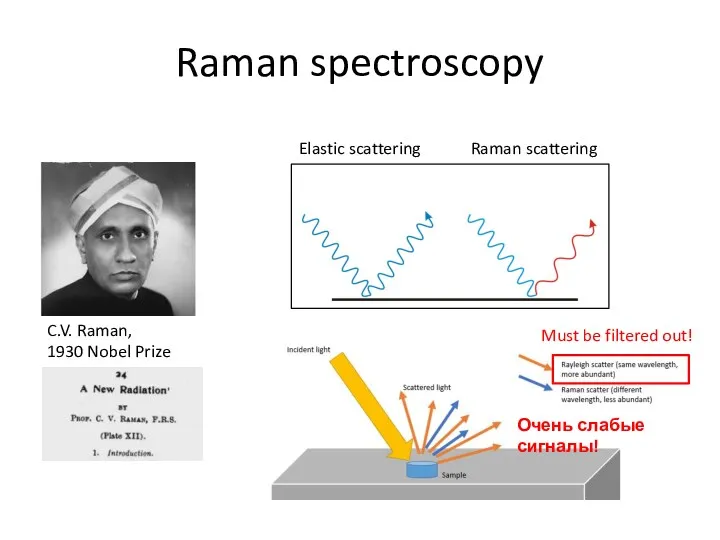
- 64. Raman spectroscopy C.V. Raman, 1930 Nobel Prize Elastic scattering Raman scattering Must be filtered out! Очень
- 65. Комбинационное рассеяние света (эффект Рамана) — неупругое рассеяние оптического излучения на молекулах вещества (твёрдого, жидкого или
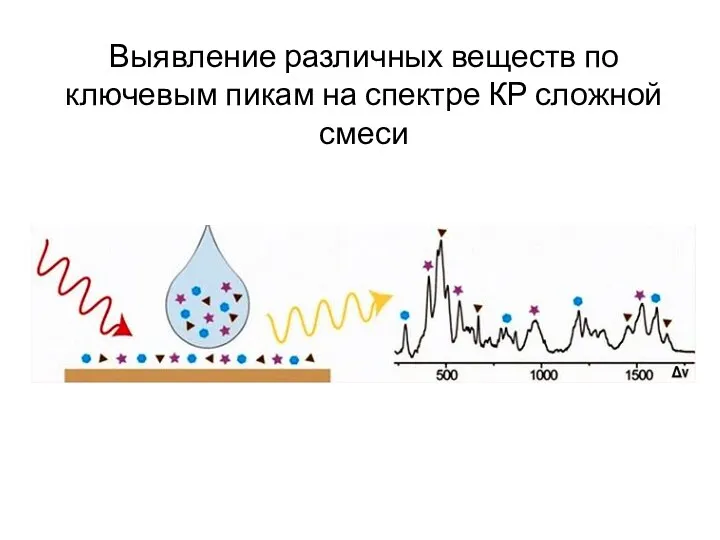
- 66. Выявление различных веществ по ключевым пикам на спектре КР сложной смеси
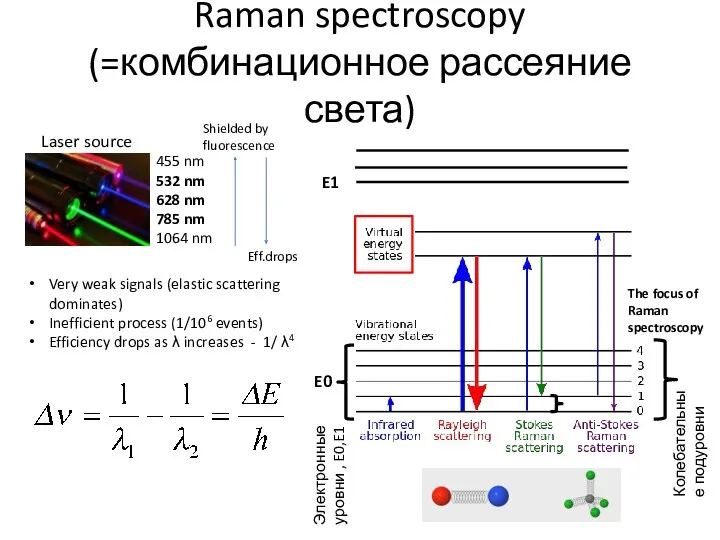
- 67. Raman spectroscopy (=комбинационное рассеяние света) The focus of Raman spectroscopy Very weak signals (elastic scattering dominates)
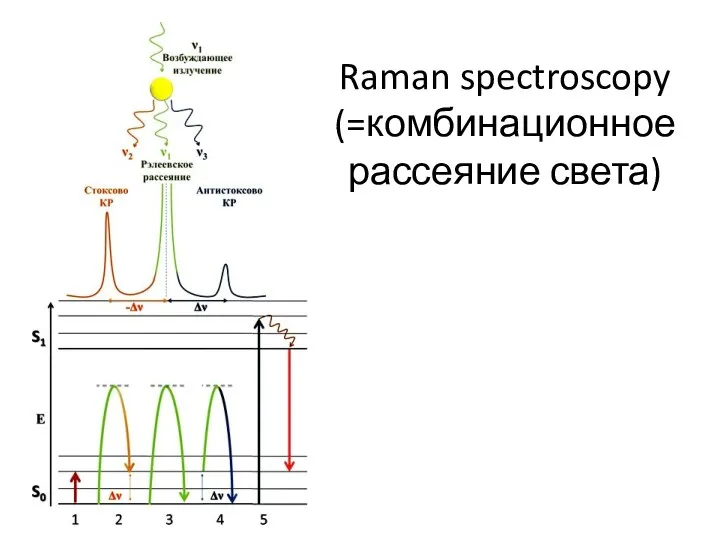
- 68. Raman spectroscopy (=комбинационное рассеяние света)
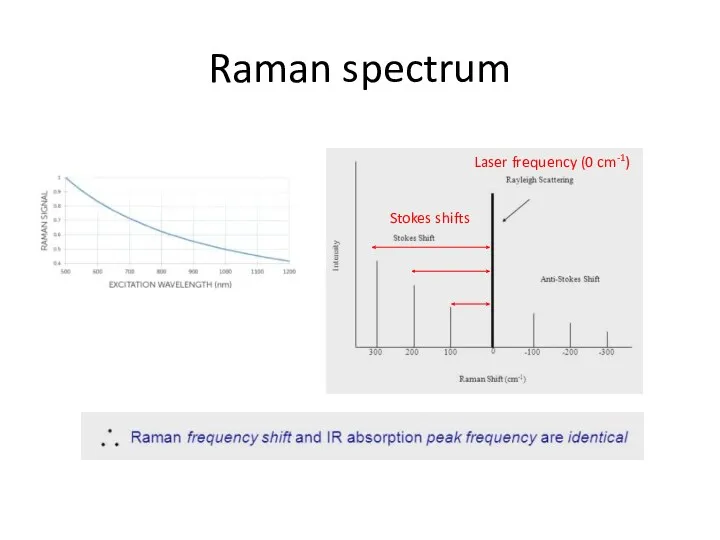
- 69. Raman spectrum Laser frequency (0 cm-1) Stokes shifts
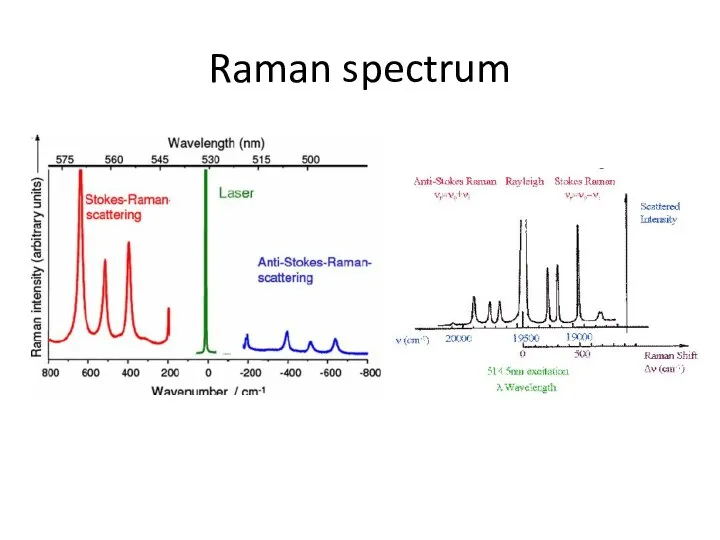
- 70. Raman spectrum
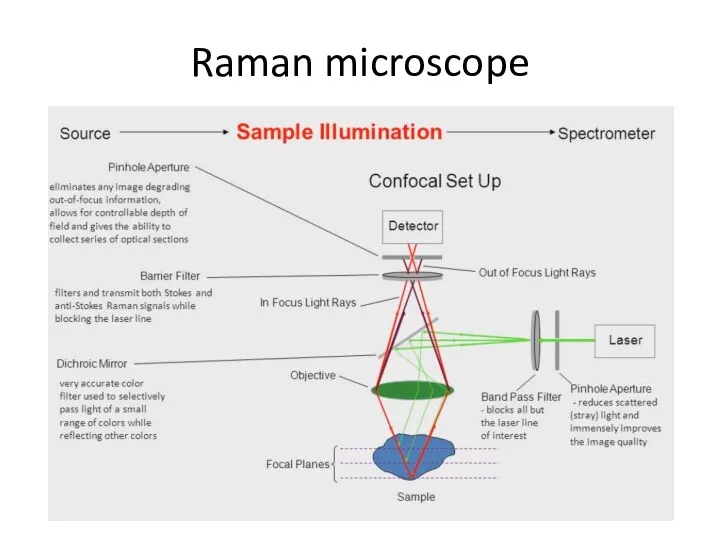
- 71. Raman microscope
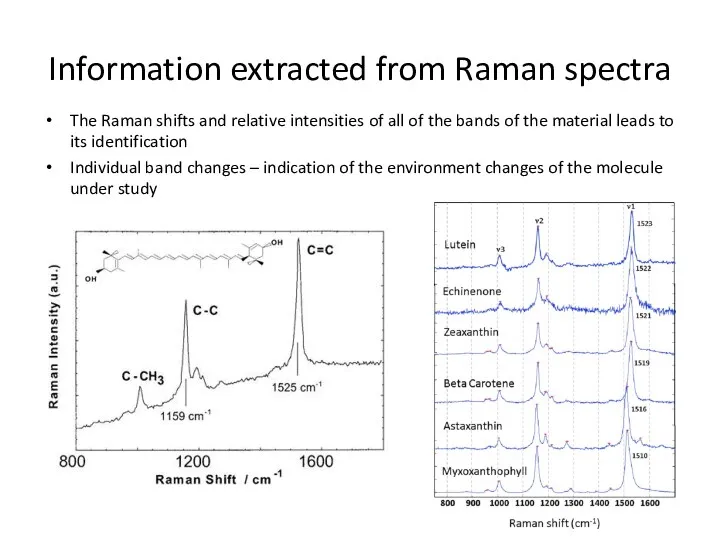
- 72. Information extracted from Raman spectra The Raman shifts and relative intensities of all of the bands
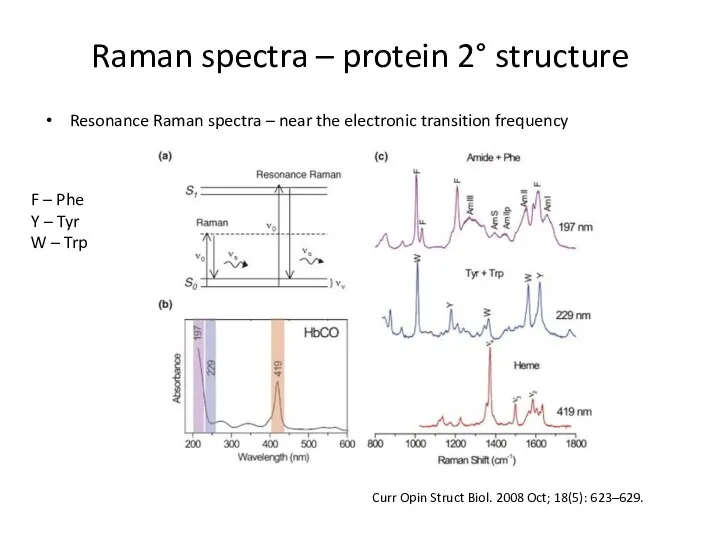
- 73. Raman spectra – protein 2° structure Resonance Raman spectra – near the electronic transition frequency Curr
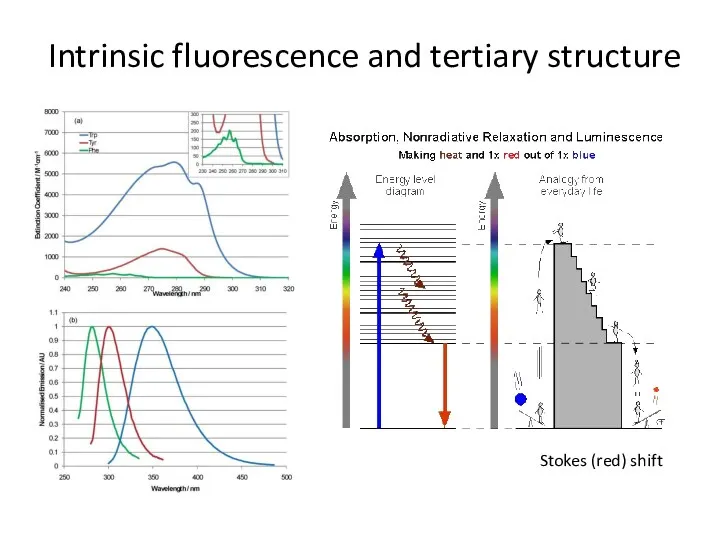
- 74. Intrinsic fluorescence and tertiary structure Stokes (red) shift
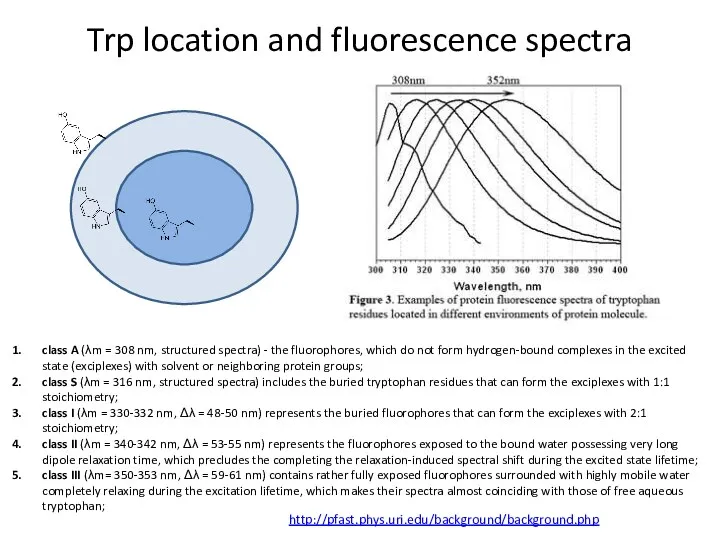
- 75. Trp location and fluorescence spectra class A (λm = 308 nm, structured spectra) - the fluorophores,
- 76. Protein folding
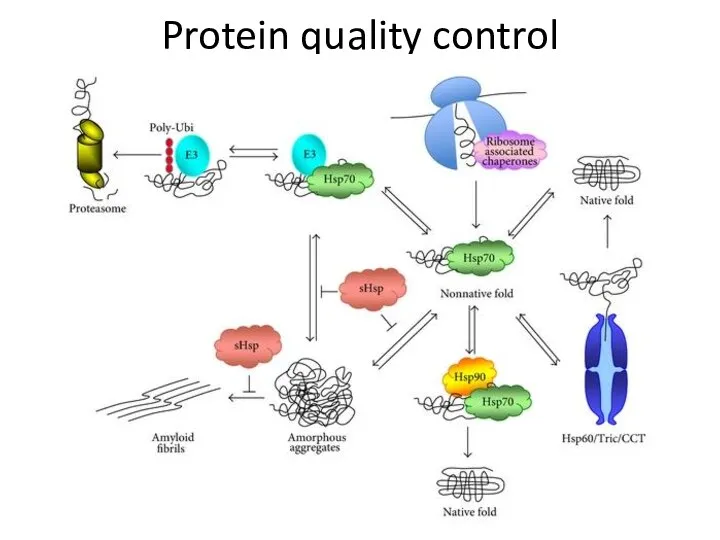
- 77. Protein quality control
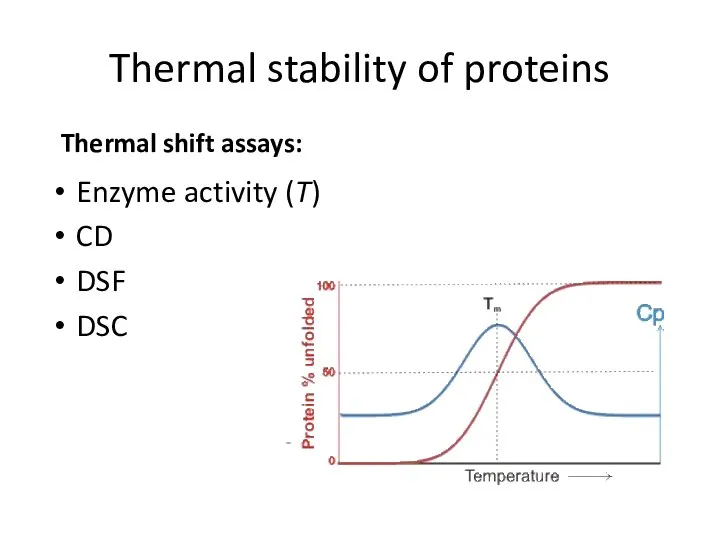
- 78. Thermal stability of proteins Enzyme activity (T) CD DSF DSC Thermal shift assays:
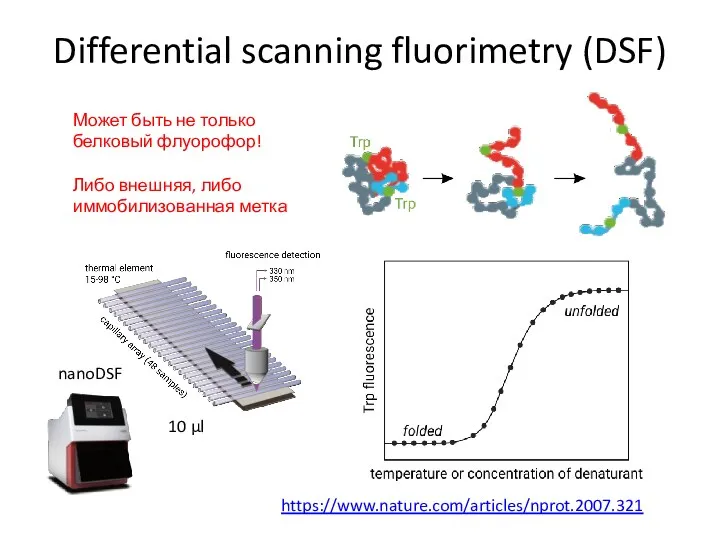
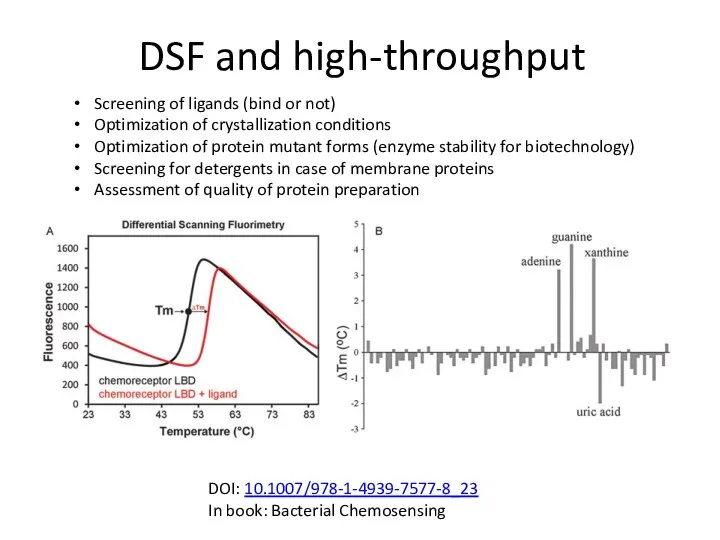
- 79. Differential scanning fluorimetry (DSF) Может быть не только белковый флуорофор! Либо внешняя, либо иммобилизованная метка https://www.nature.com/articles/nprot.2007.321
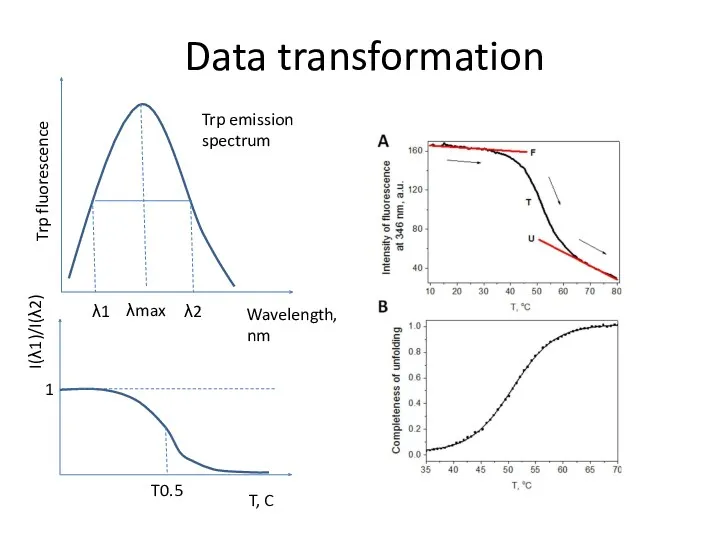
- 80. Data transformation Trp fluorescence Wavelength, nm λ1 λ2 λmax I(λ1)/I(λ2) T, C Trp emission spectrum T0.5
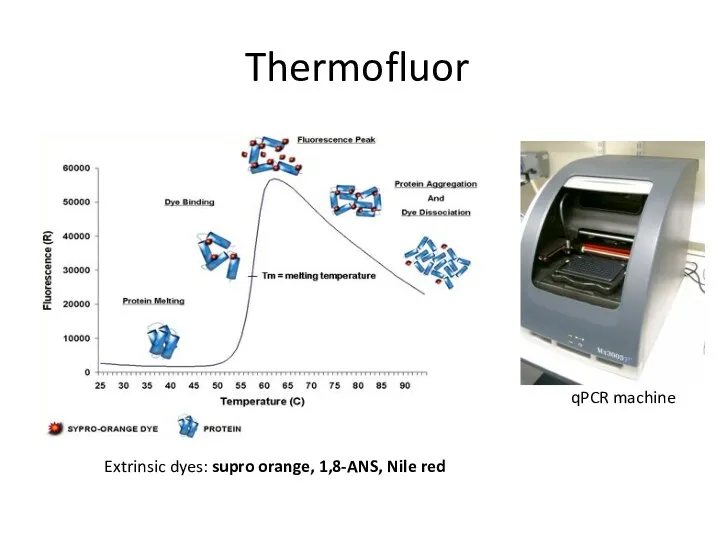
- 81. Thermofluor Extrinsic dyes: supro orange, 1,8-ANS, Nile red qPCR machine
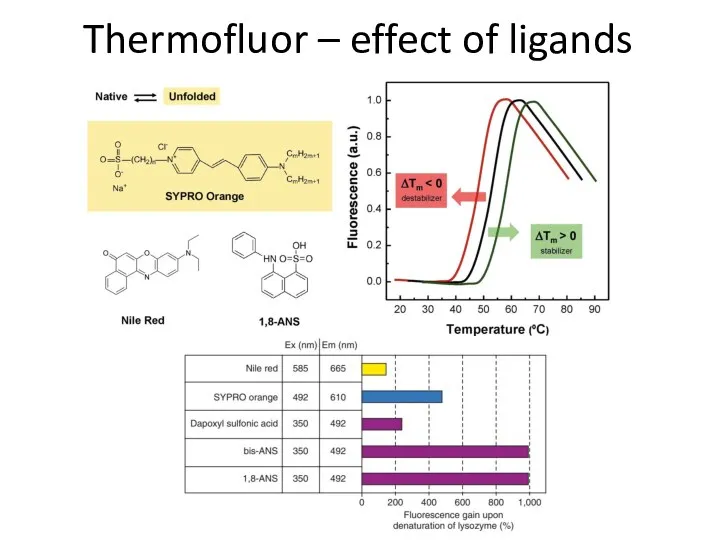
- 82. Thermofluor – effect of ligands
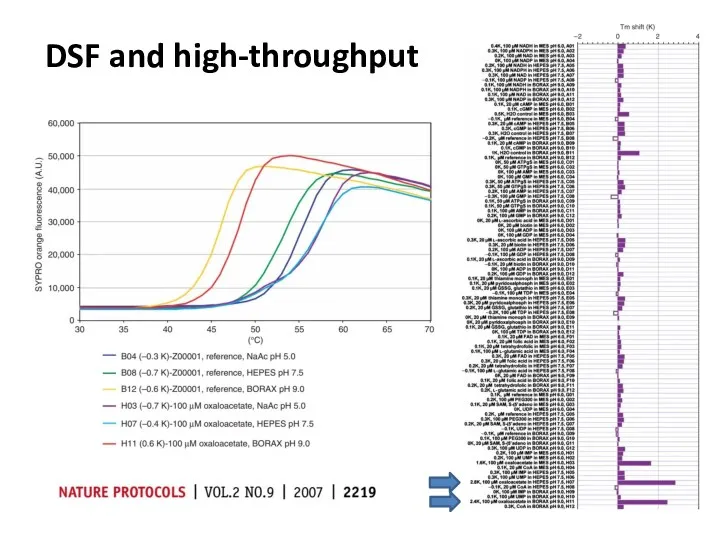
- 83. DSF and high-throughput DOI: 10.1007/978-1-4939-7577-8_23 In book: Bacterial Chemosensing Screening of ligands (bind or not) Optimization
- 84. DSF and high-throughput
- 85. Дифференциальная сканирующая калориметрия (ДСК, DSC)
- 86. Теплота и калория Количество энергии, которое теряет или получает тело в течение времени в форме теплового
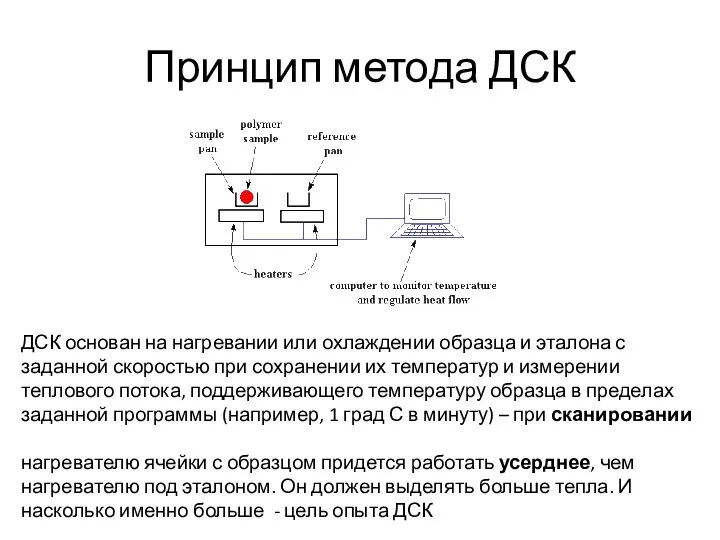
- 87. Принцип метода ДСК ДСК основан на нагревании или охлаждении образца и эталона с заданной скоростью при
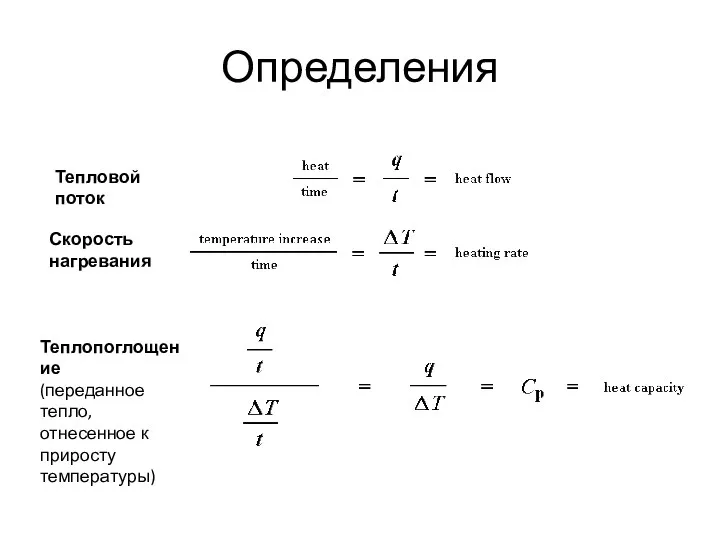
- 88. Определения Тепловой поток Скорость нагревания Теплопоглощение (переданное тепло, отнесенное к приросту температуры)
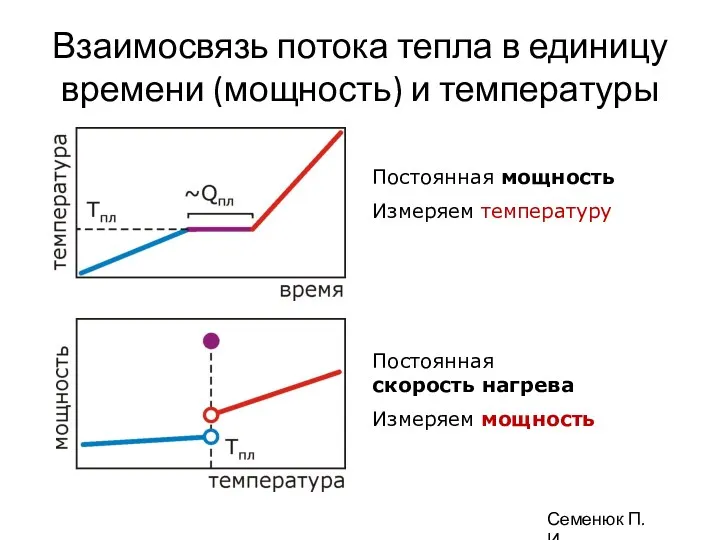
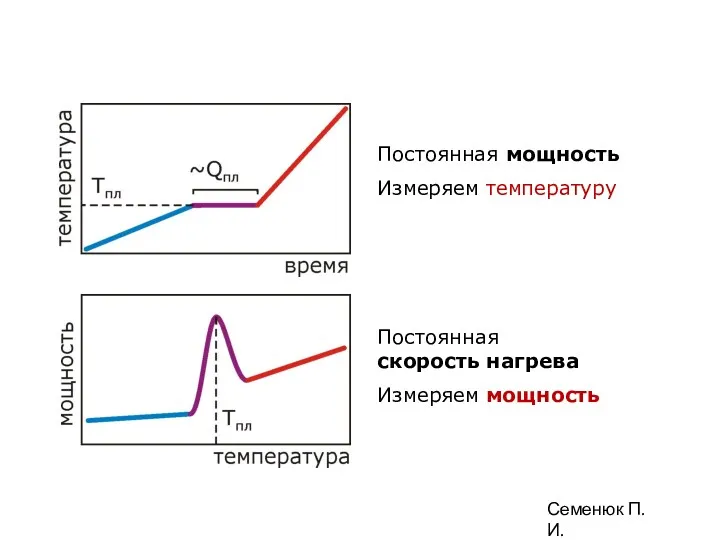
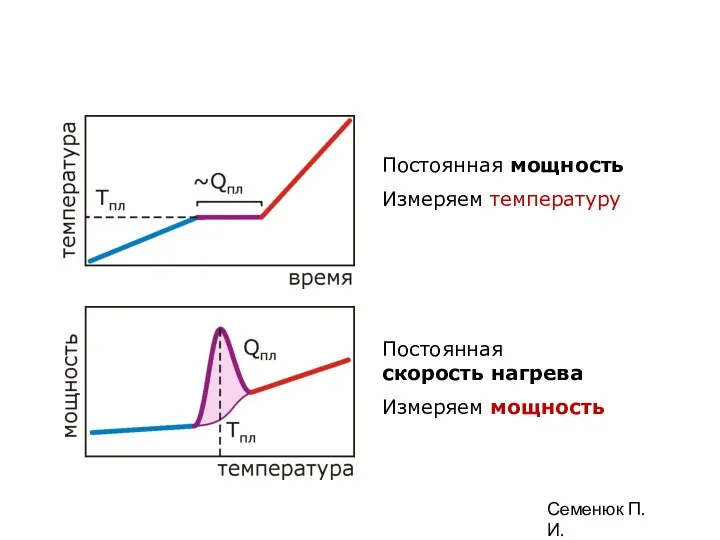
- 89. Взаимосвязь потока тепла в единицу времени (мощность) и температуры Постоянная мощность Измеряем температуру Постоянная скорость нагрева
- 90. Постоянная мощность Измеряем температуру Постоянная скорость нагрева Измеряем мощность Семенюк П.И.
- 91. Постоянная мощность Измеряем температуру Постоянная скорость нагрева Измеряем мощность Семенюк П.И.
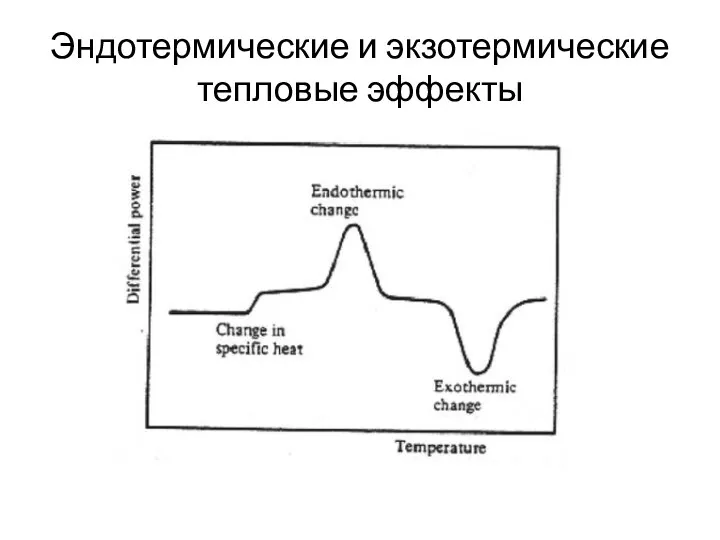
- 92. Эндотермические и экзотермические тепловые эффекты
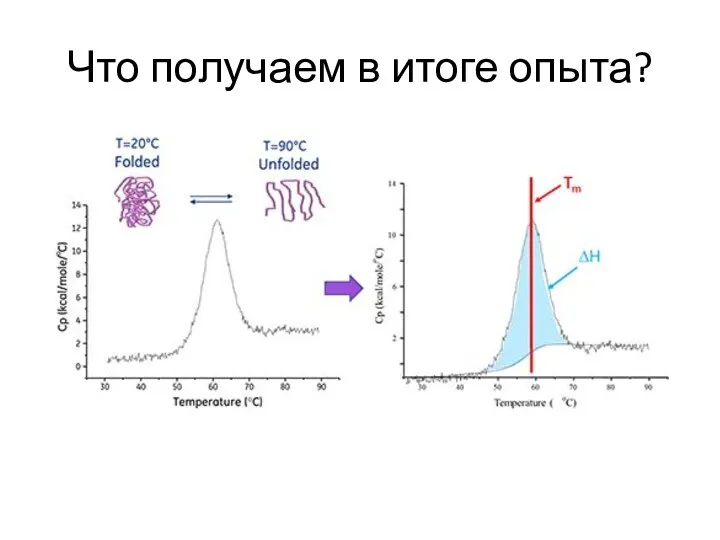
- 93. Что получаем в итоге опыта?
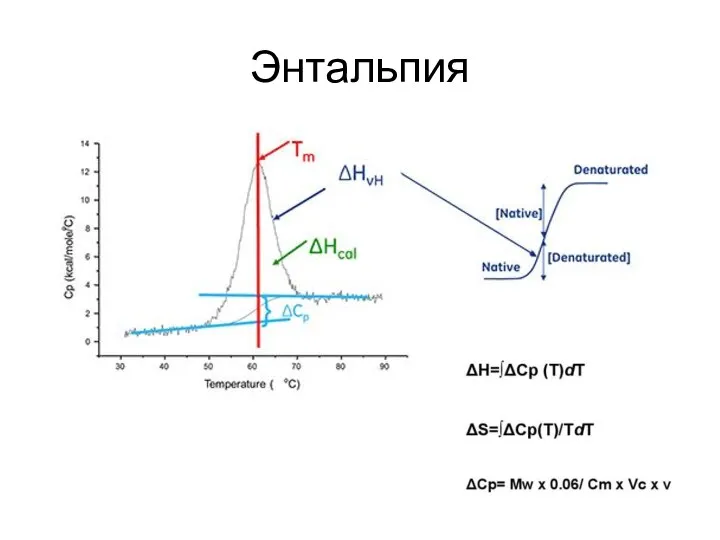
- 94. Энтальпия
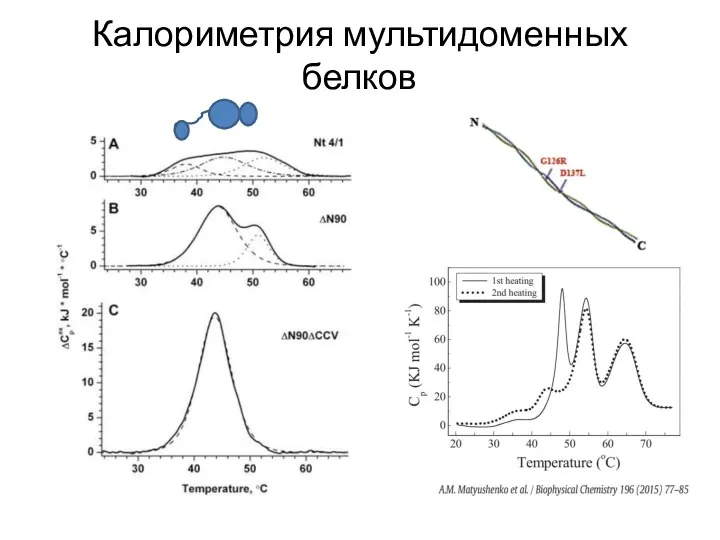
- 95. Калориметрия мультидоменных белков
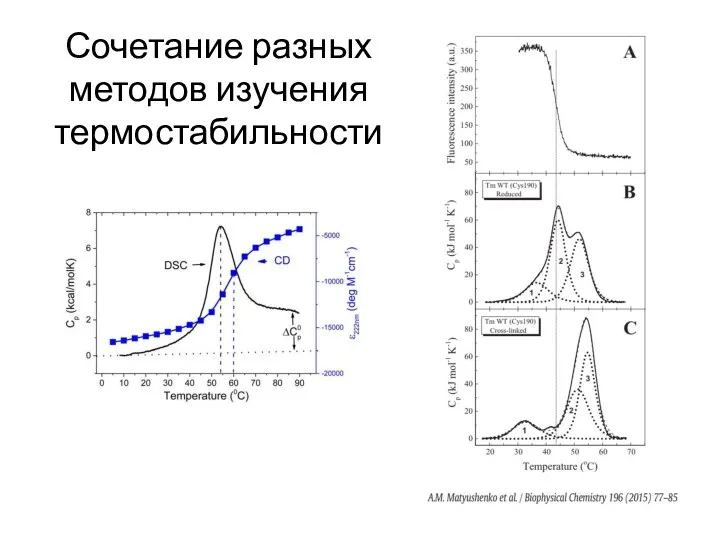
- 96. Сочетание разных методов изучения термостабильности
- 98. Скачать презентацию



















 Нанокаталіз. Активність каталізатора
Нанокаталіз. Активність каталізатора Закономерности изменения свойств химических элементов
Закономерности изменения свойств химических элементов Инструментальные методы исследования органических веществ
Инструментальные методы исследования органических веществ Alkynes
Alkynes Мұнайды өңдеудің химиялық процестерінің теориялық негіздері
Мұнайды өңдеудің химиялық процестерінің теориялық негіздері Сера. Аллотропные модификации
Сера. Аллотропные модификации Атомы и молекулы. Простые и сложные вещества (8 класс)
Атомы и молекулы. Простые и сложные вещества (8 класс) Функціональні матеріали для високоенергетичної електроніки. (Лекція 2)
Функціональні матеріали для високоенергетичної електроніки. (Лекція 2) Пниктогены элеметны VA группы
Пниктогены элеметны VA группы Концентраційні межі поширення полум'я. Лекція 7
Концентраційні межі поширення полум'я. Лекція 7 Эмульсиялардың тұрақтылығына полиэтиленгликоль-баз композицияларының әсері
Эмульсиялардың тұрақтылығына полиэтиленгликоль-баз композицияларының әсері Об изучении окислительно-восстановительных реакций в школьном курсе химии. Степени окисления атомов и формулы веществ
Об изучении окислительно-восстановительных реакций в школьном курсе химии. Степени окисления атомов и формулы веществ Ионы. Определение кислотности раствора
Ионы. Определение кислотности раствора Алюминий
Алюминий Органические вещества. Валентность. Теория химического строения
Органические вещества. Валентность. Теория химического строения Суспензии: получение, свойства
Суспензии: получение, свойства Знаки химических элементов. Периодическая система химических элементов
Знаки химических элементов. Периодическая система химических элементов Металлы и неметаллы
Металлы и неметаллы Галогены. Физические свойства галогенов
Галогены. Физические свойства галогенов Платина. Физические и химические свойства
Платина. Физические и химические свойства Гомополисахариды (углеводы растений)
Гомополисахариды (углеводы растений) Аллотропные модификации алмаза
Аллотропные модификации алмаза Атом. Модели строения атома
Атом. Модели строения атома Амфотерные оксиды и гидроксиды. 8 класс
Амфотерные оксиды и гидроксиды. 8 класс Біріншілік алкилсульфаттар және алкилэтоксисульфаттарды алу
Біріншілік алкилсульфаттар және алкилэтоксисульфаттарды алу Кремний и его соединения
Кремний и его соединения Ансамбль пор в реальном твердом теле
Ансамбль пор в реальном твердом теле Карбоновые кислоты
Карбоновые кислоты